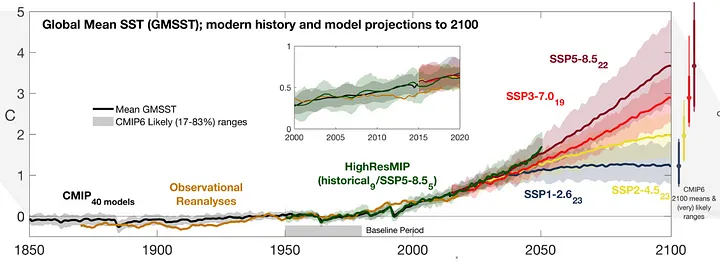
Cambio Climático: Ensamble multimodelo¶
Introducción¶
En este cuadernillo (Notebook) aprenderemos:
Breve introducción a los escenarios de Cambio Climático.
Proyecto de inter-comparación de modelos de clima acoplados - CMIP.
Acceso a los datos CMIP6 en formato Zarr.
Reproduccion de la gráfica de la Temperatura Media Global de la Superficie del Mar - CMIP6.
📚 Descripción general¶
En este cuaderno aprenderás a trabajar con proyecciones de cambio climático del Proyecto de Intercomparación de Modelos Acoplados fase 6 (CMIP6), utilizando datos de temperatura superficial del mar de múltiples modelos climáticos bajo diferentes escenarios de emisiones.
Explorarás cómo acceder a grandes volúmenes de datos climáticos almacenados en formato Zarr en Google Cloud mediante catálogos Intake-ESM. Aplicarás técnicas de ponderación espacial para calcular promedios globales correctos, y aprenderás a construir ensambles multimodelo que combinan proyecciones de diferentes centros de modelación para evaluar la incertidumbre en las proyecciones futuras del clima.
✅ Requisitos previos¶
| Conceptos | Importancia | Notas |
|---|---|---|
| Xarray | Necesario | Manejo de datos multidimensionales espacializados |
| Matplotlib | Necesario | Generación de gráficas |
| CMIP6 | Necesario | Ejemplos y análisis de CMIP6 |
| NetCDF | Útil | Familiaridad con la estructura de datos y metadatos. |
| Intake | Útil | Cátalogo que nos permite acceder a datos de diversas fuentes |
⏱️ Tiempo estimado de aprendizaje: 30 minutos
✍️ Formato: Interactivo
Librerías¶
Importamos las librerías que usaremos a lo largo de este cuadernillo.
import matplotlib.pyplot as plt
import numpy as np
import xarray as xr
from pandas import date_range
from xarrayutils.plotting import shaded_line_plot
from xmip.postprocessing import concat_members, match_metrics
from xmip.preprocessing import combined_preprocessing
from xmip.utils import google_cmip_col
from xarray import DataTree
xr.set_options(keep_attrs=True)
%matplotlib inline
plt.rcParams["figure.figsize"] = (10, 5)1. Introduccion a los escenarios de Cambio Climático¶
Los escenarios de cambio climático son una serie de modelos que se han desarrollado con el fin de comprender nuestro clima y las implicaciones futuras de las continuas emisiones de gases de efecto invernadero en la atmósfera. Estos esfuerzos se han concentrado en el Proyecto de Intercomparacion de Modelos (MIP) que invita a entidades de diferentes partes del mundo a realizar simulaciones utilizando modelos bajo escenarios de forzamiento radiativo centralizado (Abernathey, R. 2021). El más reciente Proyecto de Intercomparación de Modelos Acoplados fase 6 (CMIP6) representa un esfuerzo internacional para enfocar el conocimiento acerca de la posible evolucion del sistema climático futuro, y que se encuentra consignado y resumido en el Informe del Panel Intergubernamental sobre el Cambio Climático.
2. Acceso a los datos CMIP6¶
Los datos de los diferentes modelos se encuentran disponibles en la plataforma en la nube de Google Storage en formato Zarr. Para acceder a los datos de los diferentes escenarios de cambio climático podemos usar la librería xmip. Para crear una conexión con el repositorio de datos de Google usaremos el método google_cmip_col que nos permite acceder a los datos de Pangeo a través de intake de la siguiente manera:
cat = google_cmip_col()
catComo podemos ver hay una gran cantidad de modelaciones de más de 30 instituciones alrededor del mundo. Para efectos pedagógicos, vamos a hacer una consulta de los modelos IPSL, MPI, GFDL, EC, CMCC y CESM2 para el periodo histórico y cada uno de los de los escenarios (SSP - Shared Socioeconomic Pathways) proyectados (ssp126, ssp245, ssp370, ssp585).
Debemos crear un diccionario que nos permita posteriormente filtrar los datos deseados de la siguiente manera:
query = dict(
source_id=[
"IPSL-CM6A-LR",
"MPI-ESM1-2-LR",
# "GFDL-ESM4",
# "EC-Earth3",
"CMCC-ESM2",
# "CESM2",
],
experiment_id=["historical", "ssp126", "ssp370", "ssp245", "ssp585"],
grid_label="gn",
)El parámetro de etiqueta de retícula grid_label hace referencia a si los datos son reportados en retícula original (gn) o fue reprocesado a una nueva retícula (gr) en formato lat y lon.
Para filtrar los datos podemos aplicar el método .search a nuestro catálogo. Le pasamos los filtros previamente definidos incluyendo el identificador de la variable (variable_id), para nuestro caso la temperatura superficial del mar tos. Pasamos también el identificador de miembro member_id que para nuestro caso es r1i1p1f1.
La etiqueta de miembro nos indica lo siguiente:
r = realización
i = inicialización
p = física (parametrización)
f = forzamiento radiativo
Como último parámetro en nuestro ejemplo pasamos el identificador de table table_id que para nuestro caso son datos mensuales del oceano Omon.
cat_filt = cat.search(
**query,
variable_id="tos",
member_id=[
"r1i1p1f1",
], #'r2i1p1f1'
table_id="Omon",
)
cat_filtIntake nos permite acceder a los datos de manera rápida y fácil usando Xarray. Para cargar estos datos en un Dataset podemos aplicarle el método .to_dataset_dict que nos permite crear un diccionario con todos los modelos. Podemos pasar un diccionario kwargs con argumentos que nos permiten realizar preprocesamiento de los datos como: renombrar algunos archivos, corregir coordenadas, unidades, entre otros (como podemos ver acá).
kwargs = dict(
preprocess=combined_preprocessing,
xarray_open_kwargs=dict(use_cftime=True),
aggregate=False,
)
ddict = cat_filt.to_dataset_dict(**kwargs)
--> The keys in the returned dictionary of datasets are constructed as follows:
'activity_id.institution_id.source_id.experiment_id.member_id.table_id.variable_id.grid_label.zstore.dcpp_init_year.version'
print(list(ddict.keys())[:2])['ScenarioMIP.CMCC.CMCC-ESM2.ssp245.r1i1p1f1.Omon.tos.gn.gs://cmip6/CMIP6/ScenarioMIP/CMCC/CMCC-ESM2/ssp245/r1i1p1f1/Omon/tos/gn/v20210129/.20210129', 'ScenarioMIP.MPI-M.MPI-ESM1-2-LR.ssp370.r1i1p1f1.Omon.tos.gn.gs://cmip6/CMIP6/ScenarioMIP/MPI-M/MPI-ESM1-2-LR/ssp370/r1i1p1f1/Omon/tos/gn/v20190710/.20190710']
Revisemos el contenido de uno de estos archivos
ds_test = ddict[
"ScenarioMIP.CMCC.CMCC-ESM2.ssp126.r1i1p1f1.Omon.tos.gn.gs://cmip6/CMIP6/ScenarioMIP/CMCC/CMCC-ESM2/ssp126/r1i1p1f1/Omon/tos/gn/v20210126/.20210126"
]
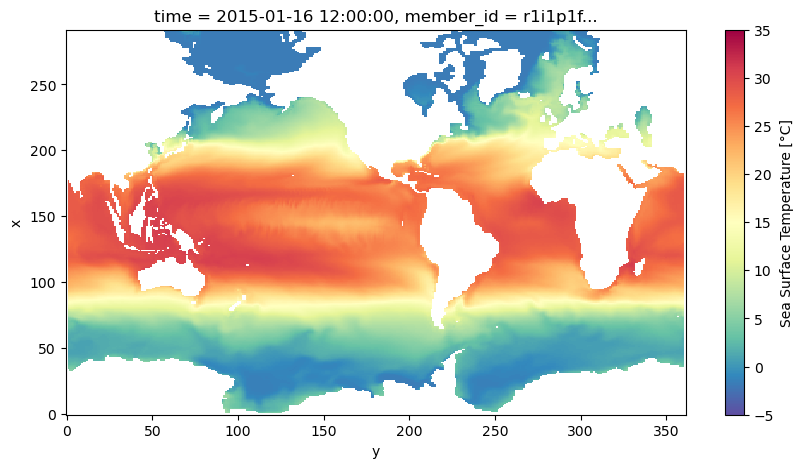
display(ds_test)Ahora una inspección gráfica
ds_test.isel(member_id=0, dcpp_init_year=0, time=0).tos.plot(
cmap="Spectral_r", vmin=-5, vmax=35
);
3. Temperatura media global ponderada¶
La temperatura superficial del mar, y cualquier otra variable o salida de los modelos de cambio climático, debe ser ponderada por el área de cada celda.
Créditos: Gael Forget. Para mas información acerca de las simulaciones y las retículas ver https://
Estas áreas ya estan calculadas y disponibles para su consulta de manera similar a los datos de temperatura. Hagamos una consulta al catálogo similar a la anterior cambiando los campos de variable_id=areacello y table_id=Ofx.
cat_area = cat.search(
**query,
table_id="Ofx",
variable_id="areacello",
)
ddict_area = cat_area.to_dataset_dict(**kwargs)
--> The keys in the returned dictionary of datasets are constructed as follows:
'activity_id.institution_id.source_id.experiment_id.member_id.table_id.variable_id.grid_label.zstore.dcpp_init_year.version'
para realizar el cálculo de la temperatura media ponderada por latitud podemos utilizar el módulo match_metrics de la libreria xmip de python de la siguiente manera:
ddict_w_area = match_metrics(ddict, ddict_area, "areacello", print_statistics=True)Processed 15 datasets.
Exact matches:{'areacello': 0}
Other matches:{'areacello': 15}
No match found:{'areacello': 0}
# ddict_w_areaAhora procederemos a concatenar los miembros en cada uno de los modelos usando el módulo .concat_members
ddict_trimmed = {k: ds.sel(time=slice(None, "2100")) for k, ds in ddict_w_area.items()}
ddict_combined_members = concat_members(
ddict_w_area,
concat_kwargs={"coords": "minimal", "compat": "override", "join": "override"},
)/home/runner/micromamba/envs/cdh-python/lib/python3.13/site-packages/xmip/postprocessing.py:152: FutureWarning: In a future version of xarray the default value for data_vars will change from data_vars='all' to data_vars=None. This is likely to lead to different results when multiple datasets have matching variables with overlapping values. To opt in to new defaults and get rid of these warnings now use `set_options(use_new_combine_kwarg_defaults=True) or set data_vars explicitly.
ds_combined = combine_func(
/home/runner/micromamba/envs/cdh-python/lib/python3.13/site-packages/xmip/postprocessing.py:152: FutureWarning: In a future version of xarray the default value for data_vars will change from data_vars='all' to data_vars=None. This is likely to lead to different results when multiple datasets have matching variables with overlapping values. To opt in to new defaults and get rid of these warnings now use `set_options(use_new_combine_kwarg_defaults=True) or set data_vars explicitly.
ds_combined = combine_func(
/home/runner/micromamba/envs/cdh-python/lib/python3.13/site-packages/xmip/postprocessing.py:152: FutureWarning: In a future version of xarray the default value for data_vars will change from data_vars='all' to data_vars=None. This is likely to lead to different results when multiple datasets have matching variables with overlapping values. To opt in to new defaults and get rid of these warnings now use `set_options(use_new_combine_kwarg_defaults=True) or set data_vars explicitly.
ds_combined = combine_func(
/home/runner/micromamba/envs/cdh-python/lib/python3.13/site-packages/xmip/postprocessing.py:152: FutureWarning: In a future version of xarray the default value for data_vars will change from data_vars='all' to data_vars=None. This is likely to lead to different results when multiple datasets have matching variables with overlapping values. To opt in to new defaults and get rid of these warnings now use `set_options(use_new_combine_kwarg_defaults=True) or set data_vars explicitly.
ds_combined = combine_func(
/home/runner/micromamba/envs/cdh-python/lib/python3.13/site-packages/xmip/postprocessing.py:152: FutureWarning: In a future version of xarray the default value for data_vars will change from data_vars='all' to data_vars=None. This is likely to lead to different results when multiple datasets have matching variables with overlapping values. To opt in to new defaults and get rid of these warnings now use `set_options(use_new_combine_kwarg_defaults=True) or set data_vars explicitly.
ds_combined = combine_func(
Xarray.Dataset no soporta tener múltiples Datasets anidados en un solo objeto de Xarray. Sin embargo, podemos crear un objeto llamado Xarray.datatree que nos permite poner todos nuestros Datasets en un solo objeto de manerar jerárquica. Para entender un poco más los formatos y objeto de tipo jerárquico vea este ejemplo.
¿Qué es DataTree?¶
DataTree es una estructura de datos de xarray que permite organizar múltiples conjuntos de datos (datasets) de forma jerárquica, similar a un sistema de archivos.
Estructura de nuestro DataTree:
ddict_combined_members/
├── IPSL-CM6A-LR/
│ ├── historical/
│ ├── ssp126/
│ ├── ssp245/
│ ├── ssp370/
│ └── ssp585/
├── MPI-ESM1-2-LR/
│ ├── historical/
│ └── ...
└── ...Cada “nodo” (como IPSL-CM6A-LR/historical) contiene un dataset xarray con la variable tos (temperatura superficial del océano) para ese modelo y escenario específico.
Acceso a los datos:
# Usando notación de diccionario tipo path
dt['IPSL-CM6A-LR/historical'].ds # Dataset con datos históricos de IPSL
dt['IPSL-CM6A-LR/ssp245'].ds # Dataset con proyección SSP2-4.5# Crear path: diccionario del dataset, donde el path está basado en cada uno de los atributos del dataset
tree_dict = {
f"{ds.source_id}/{ds.experiment_id}/": ds for ds in ddict_combined_members.values()
}
dt = DataTree.from_dict(tree_dict)
# display(dt)Para acceder a los datos dentro de este Xarray.datatree usamos notación de diccionarios de Python
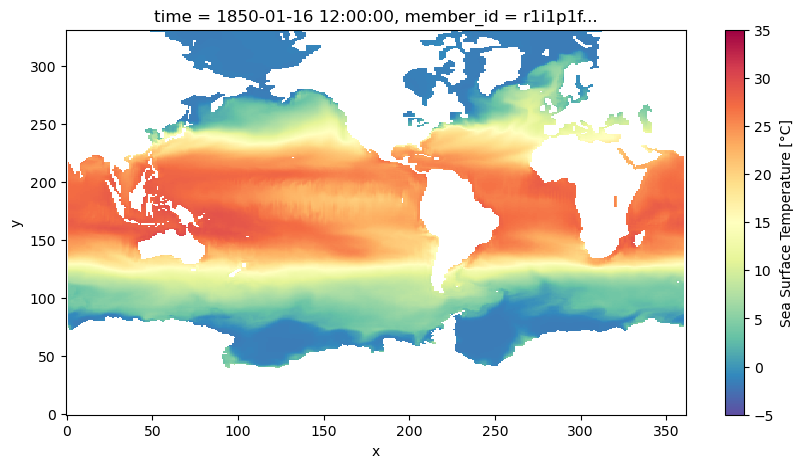
dt["IPSL-CM6A-LR/historical"].dsAhora sí podemos generar un gráfico de la temperatura media global ponderada por el área de cada celda
dt["IPSL-CM6A-LR/historical"].isel(member_id=0, dcpp_init_year=0, time=0).tos.plot(
cmap="Spectral_r", vmin=-5, vmax=35
);
from dask.distributed import Client, LocalCluster
# Create a local cluster with correct arguments
cluster = LocalCluster(
n_workers=4, # Number of worker processes
memory_limit='2GB' # Per worker memory limit
)
client = Client(cluster)
client/home/runner/micromamba/envs/cdh-python/lib/python3.13/site-packages/distributed/node.py:188: UserWarning: Port 8787 is already in use.
Perhaps you already have a cluster running?
Hosting the HTTP server on port 37027 instead
warnings.warn(
Ahora podemos calcular la anomalía de la temperatura superficial media global ponderada por latitud para cada uno de los modelos.
# Temperatura media global
def global_mean_sst(ds):
"""Calculate global mean SST weighted by cell area.
Skips empty datasets (like root DataTree node).
Returns a Dataset (required by map_over_datasets).
"""
# Skip empty datasets (e.g., root DataTree node)
if 'tos' not in ds.data_vars:
return None
# Calculate weighted mean and convert DataArray back to Dataset
result = ds['tos'].weighted(ds.areacello.fillna(0)).mean(["x", "y"]).persist()
return result.to_dataset()
# Map function over all non-empty datasets in the tree
timeseries = dt.map_over_datasets(global_mean_sst)
# timeseriesPodemos generar una serie de tiempo para inspeccionar los resultados. Usemos el modelo IPSl con el escenario ssp585.
ds_IPSL_ssp585 = timeseries["/IPSL-CM6A-LR/ssp585"].ds
ds_IPSL_ssp585 = ds_IPSL_ssp585.assign_coords(
time=("time", ds_IPSL_ssp585.time.data.astype("datetime64[ns]"))
)
ds_IPSL_ssp5854. Anomalía de la temperatura media global ponderada¶
De igual manera podemos remover el ciclo estacional, también llamado “climatología”, para calcular la anomalía de la temperatura superficial del Océano.
# Valor medio histórico de referencia
def get_ref_value(ds):
return ds.sel(time=slice("1950", "1980")).mean("time")
anomaly = DataTree()
for model_name, model in timeseries.children.items():
# model-specific base period
base_period = get_ref_value(model["historical"].ds)
anomaly[model_name] = model - base_period # subtree - Dataset# display(anomaly)Los datos de los modelos de cambio climático traen una estampa de tiempo en formato cftime.DatetimeGregorian. Xarray y Matplotlib generalmente trabajan con estampas de tiempo de datetime64. Debemos convertir estos índices usando la función replace_time. Luego podemos agrupar los datos por escenario usando un diccionario como se muestra a continuación:
# anomaly['/IPSL-CM6A-LR/ssp585'].timedef replace_time(ds):
start_date = ds.time.data[0]
new_time = date_range(
f"{start_date.year}-{start_date.month:02}", freq="1MS", periods=len(ds.time)
)
ds_new_cal = ds.assign_coords(time=new_time, source_id=model_name)
return ds_new_cal
experiment_dict = {
k: [] for k in ["historical", "ssp126", "ssp370", "ssp245", "ssp585"]
}
experiment_dict{'historical': [], 'ssp126': [], 'ssp370': [], 'ssp245': [], 'ssp585': []}Iteramos sobre cada uno de los modelos en el objeto datatree donde tenemos las anomalías de la temperatura superficial del mar y las agregamos a cada una de las llaves del diccionario
for model_name, model in list(anomaly.children.items()):
for experiment_name, experiment in model.children.items():
# replace the time dimension
ds_new_cal = replace_time(experiment.ds)
experiment_dict[experiment_name].append(ds_new_cal.load())Concatenamos cada escenario a lo largo de la dimension source_id
# Concatenar todos los modelos para un determinado experimento
plot_dict = {
k: xr.concat(ds_lst, dim="source_id") for k, ds_lst in experiment_dict.items()
}
plot_dict{'historical': <xarray.Dataset> Size: 40kB
Dimensions: (source_id: 3, member_id: 1, dcpp_init_year: 1, time: 1980)
Coordinates:
* source_id (source_id) <U13 156B 'CMCC-ESM2' ... 'IPSL-CM6A-LR'
* member_id (member_id) object 8B 'r1i1p1f1'
* dcpp_init_year (dcpp_init_year) object 8B None
* time (time) datetime64[ns] 16kB 1850-01-01 ... 2014-12-01
Data variables:
tos (source_id, member_id, dcpp_init_year, time) float32 24kB ...,
'ssp126': <xarray.Dataset> Size: 21kB
Dimensions: (source_id: 3, member_id: 1, dcpp_init_year: 1, time: 1032)
Coordinates:
* source_id (source_id) <U13 156B 'CMCC-ESM2' ... 'IPSL-CM6A-LR'
* member_id (member_id) object 8B 'r1i1p1f1'
* dcpp_init_year (dcpp_init_year) object 8B None
* time (time) datetime64[ns] 8kB 2015-01-01 ... 2100-12-01
Data variables:
tos (source_id, member_id, dcpp_init_year, time) float32 12kB ...,
'ssp370': <xarray.Dataset> Size: 21kB
Dimensions: (source_id: 3, member_id: 1, dcpp_init_year: 1, time: 1032)
Coordinates:
* source_id (source_id) <U13 156B 'CMCC-ESM2' ... 'IPSL-CM6A-LR'
* member_id (member_id) object 8B 'r1i1p1f1'
* dcpp_init_year (dcpp_init_year) object 8B None
* time (time) datetime64[ns] 8kB 2015-01-01 ... 2100-12-01
Data variables:
tos (source_id, member_id, dcpp_init_year, time) float32 12kB ...,
'ssp245': <xarray.Dataset> Size: 21kB
Dimensions: (source_id: 3, member_id: 1, dcpp_init_year: 1, time: 1032)
Coordinates:
* source_id (source_id) <U13 156B 'CMCC-ESM2' ... 'IPSL-CM6A-LR'
* member_id (member_id) object 8B 'r1i1p1f1'
* dcpp_init_year (dcpp_init_year) object 8B None
* time (time) datetime64[ns] 8kB 2015-01-01 ... 2100-12-01
Data variables:
tos (source_id, member_id, dcpp_init_year, time) float32 12kB ...,
'ssp585': <xarray.Dataset> Size: 21kB
Dimensions: (source_id: 3, member_id: 1, dcpp_init_year: 1, time: 1032)
Coordinates:
* source_id (source_id) <U13 156B 'CMCC-ESM2' ... 'IPSL-CM6A-LR'
* member_id (member_id) object 8B 'r1i1p1f1'
* dcpp_init_year (dcpp_init_year) object 8B None
* time (time) datetime64[ns] 8kB 2015-01-01 ... 2100-12-01
Data variables:
tos (source_id, member_id, dcpp_init_year, time) float32 12kB ...}Comprobamos que nuestras estampas de tiempo esten en el formato datetime64
plot_dict["historical"].time5. Ensamble Multimodelo¶
Finalmente generamos nuestra gráfica de las proyecciones de la anomalía de temperatura superficial del mar bajo los distintos escenarios. Para esto podemos usar la función shaded_line_plot que produce un gráfico de líneas con intervalos sombreados según la extensión del datarray en la dimensión source_id. Creamos nuestro ensamble multimodelo!!!
fig, ax = plt.subplots(figsize=(8, 4))
color_dict = {
"historical": "0.5",
"ssp126": "C2",
"ssp245": "gold",
"ssp370": "C1",
"ssp585": "C3",
"observations": "C5",
}
for experiment, ds in plot_dict.items():
color = color_dict[experiment]
smooth = (
ds["tos"]
.sel(time=slice(None, "2100"))
.isel(member_id=0)
.rolling(time=2 * 12)
.mean()
.squeeze(["dcpp_init_year"])
)
lw = 2 if experiment == "observations" else 1.5
shaded_line_plot(
smooth,
"source_id",
ax=ax,
spreads=[2.0],
alphas=[0.2],
line_kwargs=dict(
color=color, label=f"{experiment} ({len(ds.source_id)})", lw=lw
),
)
plt.legend(loc=2)
plt.grid()
plt.tight_layout()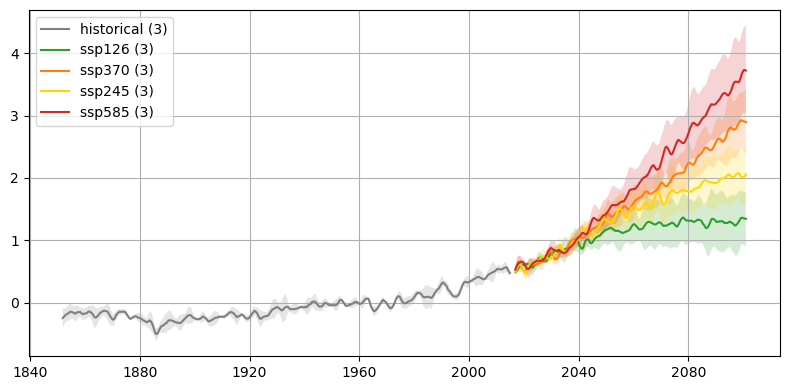
Resumen final¶
En este cuadernillo (notebook) revisamos temas relacionados con el acceso a la información proveniente de diferentes escenarios de Cambio Climático a partir de los conjuntos de datos disponibles en CMIP. Promediamos la temperatura superficial teniendo en cuenta el tamaño de las celdas del modelo y, reprodujimos la gráfica multimodelo de las proyecciones de cambio climático.
¿Qué sigue?¶
Con los conocimientos sobre análisis de temperatura global con CMIP6, puedes explorar:
3.3. Datos de reanálisis ERA5 - Análisis de variables atmosféricas con datos de reanálisis de alta resolución
3.1. ENSO - Relación entre temperatura superficial del mar y eventos El Niño/La Niña
Análisis regional - Aplicar estas técnicas a regiones específicas de América Latina
Proyecto sugerido:¶
Adapta este análisis para explorar:
Proyecciones de temperatura para Colombia o América Latina usando selecciones regionales
Comparación cuantitativa entre escenarios SSP2-4.5 vs SSP5-8.5
Análisis de incertidumbre entre diferentes modelos CMIP6 (rango de proyecciones)
Fuentes y referencias¶
ClimateMatch Academy: Computational Tools for Climate Science. (s. f.). https://
comptools .climatematch .io /tutorials /intro .html Abernathey, R. 2023. An Introduction to Earth and Environmental Data Science. Retrieved from Earth and Environmental Data Science: https://
earth -env -data -science .github .io /intro .html Easy IPCC Part 1: Multi-Model Datatree. Nicholas T. 2019. https://
medium .com /pangeo /easy -ipcc -part -1 -multi -model -datatree -469b87cf9114