NumPy, Pandas y Xarray
Introducción
En este cuadernillo (Notebook) aprenderemos acerca de librerías útiles en la programación científica:
Introducción numpy
Introducción pandas
Introducción xarray
Este cuadernillo contiene información simplificada de Pythia Fundations
Prerequisitos
Conceptos |
Importancia |
Notas |
|---|---|---|
Necesario |
Información complementaria |
|
Necesario |
Información complementaria |
|
Necesario |
Información complementaria |
|
Necesario |
Entender estampas de tiempo |
Tiempo de aprendizaje: 30 minutos
Librerías
A continuación presentamos las librerías que vamos a usar durante este cuadernillo
from datetime import timedelta # manejo de estampas de tiempo
import numpy as np # Manejo de matrices multidimensionales
import pandas as pd # Manejo de datos tabulares y series de tiempo
import xarray as xr # Manejo óptimo de datos multidimensionales
from pythia_datasets import DATASETS # datos disponibles en Pythia
1. NumPy
Numpy es un paquete o librería fundamental en Python que nos permite trabajar principalmente con arreglos y matrices multidimensionales. Con NumPy podemos realizar operaciones matemáticas, reorganización de matrices, operaciones básicas de álgebra lineal, análisis estadísticos básicos, entre muchas otras.
¿Quién usa Numpy?
Todo aquel que en su campo de estudio necesite de una herramienta flexible y vectorizada que permita el manejo de datos en diferentes formatos tal que se ajusten a su paradigama de codificación. En otras palabras, Todo aquel que lo encuentre útil para su problema de estudio.
Ventajas de NumPy
Está vectorizado, lo cual significa que no se necesita un bucle explícito, indexación, etc., para lograr alguno métodos. Además, está optminizado en C, tal que es mucho más rápido que una programación en Python básica.
Es consciso y más fácil de leer, nos ahorra líneas de código y hace las operaciones multidimensionales más sencillas para el usuario.
Diferencias entre los array de NumPy y listas de Python
Los NumPy array tienen un tamaño fijo en la creación.
Todos los elementos de un NumPy array deben ser del mismo tipo de datos.
Los NumPy array facilitan operaciones matemáticas avanzadas y de otro tipo en grandes cantidades de datos.
1.1 Creación de vectores
Con NumPy podemos realizar creacion de arreglos y vectores de múltiples dimensiones usando diferentes métodos. La manera más común de crear un arreglo o matriz es usando el método np.array.
vector = np.array([1, 2, 3])
vector
array([1, 2, 3])
Los objetos del tipo numpy.ndarray (array de NumPy) tienen métodos autocontenidos que nos permiten obterner propiedades como dimensión ndim, tamaño shape o tipo de datos dtype.
vector.ndim
1
vector.shape
(3,)
vector.dtype
dtype('int64')
Ahora podemos crear una matriz de dos dimensiones de la misma manera
matriz_2d = np.array([[0, 1, 2], [3, 4, 5]])
matriz_2d
array([[0, 1, 2],
[3, 4, 5]])
print(
f"dimensiones = {matriz_2d.ndim}, forma = {matriz_2d.shape}, y tipo {matriz_2d.dtype}"
)
dimensiones = 2, forma = (2, 3), y tipo int64
1.2 Generación de matrices y vectores
NumPy ofrece funciones y métodos que permiten generar matrices o arreglos igualmente espaciados. Generalmente NumPy usa reglas de indexación de la siguiente manera
.arange(comienzo, fin, paso)crea un arreglo o matriz de valores en el intervalo[comienzo, fin)espaciado cadapaso.linspace(comienzo, fin, número de divisiones)crea un arreglo o matriz de valores en el intervalo[comienzo, fin)igualmente espaciado usandonúmero de divisiones
arreglo = np.arange(10)
arreglo
array([0, 1, 2, 3, 4, 5, 6, 7, 8, 9])
arreglo_espaciado = np.linspace(1, 10, 10)
arreglo_espaciado
array([ 1., 2., 3., 4., 5., 6., 7., 8., 9., 10.])
1.3 Operaciones básica usando NumPy
Podemos realizar operaciones matemáticas usando NumPy teniendo en cuenta que los arreglos o matrices deben tener el mismo tamaño. Las operaciones se realizarán elemento a elemento en cada arreglo matricial
a = np.arange(0, 6, 2)
a
array([0, 2, 4])
b = np.array([-1, 200, 1.3])
b
array([ -1. , 200. , 1.3])
a + b
array([ -1. , 202. , 5.3])
a - b
array([ 1. , -198. , 2.7])
a * b
array([ -0. , 400. , 5.2])
a / b
array([-0. , 0.01 , 3.07692308])
1.4 Operaciones matemáticas más complejas
NumPy soporta operaciones matemáticas mas complejas elemento a elemento en cada arreglo matricial. Por ejemplo, calculemos el seno de una matriz
matriz_2d = np.array([[0, 1, 2], [3, 4, 5]])
matriz_2d
array([[0, 1, 2],
[3, 4, 5]])
np.sin(matriz_2d)
array([[ 0. , 0.84147098, 0.90929743],
[ 0.14112001, -0.7568025 , -0.95892427]])
Ahora usando la constante pi
t = np.arange(0, 2 * np.pi + np.pi / 4, np.pi / 4)
t
array([0. , 0.78539816, 1.57079633, 2.35619449, 3.14159265,
3.92699082, 4.71238898, 5.49778714, 6.28318531])
t / np.pi
array([0. , 0.25, 0.5 , 0.75, 1. , 1.25, 1.5 , 1.75, 2. ])
cos_t = np.cos(t)
cos_t
array([ 1.00000000e+00, 7.07106781e-01, 6.12323400e-17, -7.07106781e-01,
-1.00000000e+00, -7.07106781e-01, -1.83697020e-16, 7.07106781e-01,
1.00000000e+00])
Podemos redondear las cifras usando el método round
np.round(cos_t, 2)
array([ 1. , 0.71, 0. , -0.71, -1. , -0.71, -0. , 0.71, 1. ])
También podemos sumar todos los elementos de un arreglo usando np.sum
np.sum(cos_t)
0.9999999999999996
Para mas detalles, les dejamos el link a la documentación de operaciones matemáticas con NumPy y el link a las funciones de álgebra lineal.
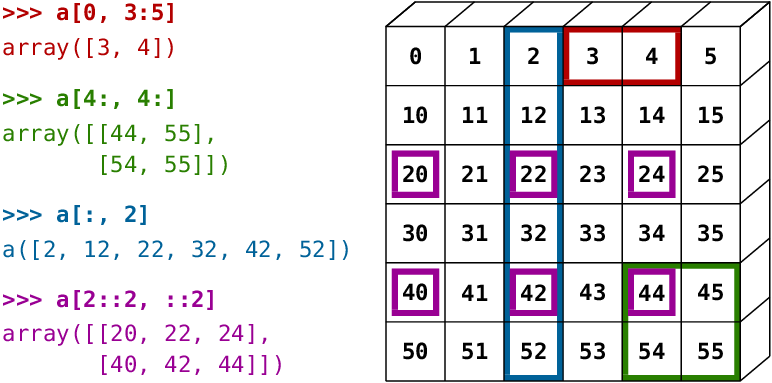
1.5 Indexado y selección de datos
Podemos acceder a los valores dentro de un arreglo matricial multidimensional utilizando el índice del vector o matriz. Recordemos que en Python, el índice comienza en 0 y, el acceso se realiza usando la notación [fila, columna].
matriz = np.arange(12).reshape(3, 4)
matriz
array([[ 0, 1, 2, 3],
[ 4, 5, 6, 7],
[ 8, 9, 10, 11]])
Podemos acceder al primer elemento de la matriz de la siguinte manera:
matriz[0, 0]
0
Para acceder al elemento ubicado en la fila 2 y la columna 4
matriz[1, 3]
7
Para acceder a los últimos elementos del arreglo, podemos usar el índice en “reversa”
matriz[-1, 0]
8
matriz[0, -1]
3
matriz[-1, -1]
11
Para seleccionar un rango de valores dentro del arreglo matricial usamos la notación [comienzo:final[:paso]]. Por ejemplo, tratemos de seleccionar la primera fila:
matriz[0, 0:4]
array([0, 1, 2, 3])
Ahora la primera fila sin incluir el último elemento:
matriz[0, 0:-1]
array([0, 1, 2])
Podemos crear un arreglo unidimensional con mayor número de elementos para observar la selección de un rango de elementos usando un paso determinado
arreglo_largo = np.arange(0, 15, 1)
arreglo_largo
array([ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14])
arreglo_largo[::2]
array([ 0, 2, 4, 6, 8, 10, 12, 14])
Ahora incluyendo comienzo=3, final=13 paso=2
arreglo_largo[3:13:2]
array([ 3, 5, 7, 9, 11])
Precaución
El índice en la selección de rango no incluye el valor de la derecha
arreglo_largo[0:3]
array([0, 1, 2])
En el arreglo anterior la selección se realizó entre el índice 0 y el 3 no incluyente
En resumen, podemos seleccionar fácilmente subconjuntos de datos en nuestros numpy.ndarrays.
2. Pandas
Pandas es una de las librerías de código abierto más potentes en el ámbito de la programación científica que permite la manipulación rápida y fácil de datos tabulares en diversos formatos (Excel, texto plano separado por comas -csv-, bases de datos, pickle, entre muchos otros). La manipulación de datos tabulares y series de tiempo se realiza mediante etiquetas que nos permiten escribir códigos robustos/consistentes.
2.1 Pandas DataFrame
Es un conjunto de datos tabulares, similar a un hoja de calculo de excel, una tabla de datos o un data.frame en R, que usa etiquetas como índices. Los DataFrames estan compuestos por columnas y filas.
Dentro de cada columna/fila podemos tener datos de diferente tipo incluyendo números, texto, estampas de tiempo, entre otros. En la imagen anterior (Cortesía de Pythia Fundations. 2023, CC-BY), la columna de la izquierda, sombreada en color gris, es conocida como el índice de las filas. Análogamente, la parte superior del DataFrame, podemos encontrar el índice de las columnas. Estos índices, de columna y fila, pueden ser de tipo numérico, caracteres, estampas de tiempo, entre muchos otros.
A continuación, se puede observar un DataFrame de anomalías de la temperatura superfical del mar en las diferentes regiones de El Niño:
filepath = DATASETS.fetch("enso_data.csv")
Downloading file 'enso_data.csv' from 'https://github.com/ProjectPythia/pythia-datasets/raw/main/data/enso_data.csv' to '/home/runner/.cache/pythia-datasets'.
df = pd.read_csv(filepath)
df.head()
| datetime | Nino12 | Nino12anom | Nino3 | Nino3anom | Nino4 | Nino4anom | Nino34 | Nino34anom | |
|---|---|---|---|---|---|---|---|---|---|
| 0 | 1982-01-01 | 24.29 | -0.17 | 25.87 | 0.24 | 28.30 | 0.00 | 26.72 | 0.15 |
| 1 | 1982-02-01 | 25.49 | -0.58 | 26.38 | 0.01 | 28.21 | 0.11 | 26.70 | -0.02 |
| 2 | 1982-03-01 | 25.21 | -1.31 | 26.98 | -0.16 | 28.41 | 0.22 | 27.20 | -0.02 |
| 3 | 1982-04-01 | 24.50 | -0.97 | 27.68 | 0.18 | 28.92 | 0.42 | 28.02 | 0.24 |
| 4 | 1982-05-01 | 23.97 | -0.23 | 27.79 | 0.71 | 29.49 | 0.70 | 28.54 | 0.69 |
Como podemos observar, el índice, tanto en filas y columnas, se resaltan en negrita. En la filas el índice por defecto es una secuencia numerada que incia en 0 y termina en el número de filas del set de datos. Para acceder al índice en filas podemos usar el atributo .index y en columnas .columns.
df.index
RangeIndex(start=0, stop=472, step=1)
Hasta el momento, aún no hemos sacado aprovechado de las ventajas de Pandas y etiquetas en los índices.
Utilicemos la columna datetime como índice de las filas en formato de estampa de tiempo. Para hacer esto podemos pasar múltiples argumentos (index_col, parser_dates) al método pd.read_csv de acuerdo con la documentación oficial.
df = pd.read_csv(filepath, index_col=0, parse_dates=True)
df.head()
| Nino12 | Nino12anom | Nino3 | Nino3anom | Nino4 | Nino4anom | Nino34 | Nino34anom | |
|---|---|---|---|---|---|---|---|---|
| datetime | ||||||||
| 1982-01-01 | 24.29 | -0.17 | 25.87 | 0.24 | 28.30 | 0.00 | 26.72 | 0.15 |
| 1982-02-01 | 25.49 | -0.58 | 26.38 | 0.01 | 28.21 | 0.11 | 26.70 | -0.02 |
| 1982-03-01 | 25.21 | -1.31 | 26.98 | -0.16 | 28.41 | 0.22 | 27.20 | -0.02 |
| 1982-04-01 | 24.50 | -0.97 | 27.68 | 0.18 | 28.92 | 0.42 | 28.02 | 0.24 |
| 1982-05-01 | 23.97 | -0.23 | 27.79 | 0.71 | 29.49 | 0.70 | 28.54 | 0.69 |
Como podemos ver, el índice del DataFrame ahora es la columna datetime y está en formato timestamp
df.index
DatetimeIndex(['1982-01-01', '1982-02-01', '1982-03-01', '1982-04-01',
'1982-05-01', '1982-06-01', '1982-07-01', '1982-08-01',
'1982-09-01', '1982-10-01',
...
'2020-07-01', '2020-08-01', '2020-09-01', '2020-10-01',
'2020-11-01', '2020-12-01', '2021-01-01', '2021-02-01',
'2021-03-01', '2021-04-01'],
dtype='datetime64[ns]', name='datetime', length=472, freq=None)
De igual manera, podemos ver los índices / nombres de las columnas de la siguiente manera:
df.columns
Index(['Nino12', 'Nino12anom', 'Nino3', 'Nino3anom', 'Nino4', 'Nino4anom',
'Nino34', 'Nino34anom'],
dtype='object')
2.2. Pandas Series
Una serie de datos en Pandas hace refencia a datos tabulares que continenen una sola columna; al igual que un DataFrame puede contener cualquir tipo de dato o variable. En el siguiente ejemplo extraeremos la serie de datos de la anomalía de la temperatura superficial de niño en la región 3-4 usando el método de llave-valor [''].
series = df["Nino34anom"]
series.head()
datetime
1982-01-01 0.15
1982-02-01 -0.02
1982-03-01 -0.02
1982-04-01 0.24
1982-05-01 0.69
Name: Nino34anom, dtype: float64
Alternativamente, podemos acceder a misma serie de datos usando el método punto de la siguiente manera:
series = df.Nino34anom
series.head()
datetime
1982-01-01 0.15
1982-02-01 -0.02
1982-03-01 -0.02
1982-04-01 0.24
1982-05-01 0.69
Name: Nino34anom, dtype: float64
2.3 Selección de series y set de datos
Como mencionamos anteriormente, las etiquetas en los índices nos permiten seleccionar un subconjunto de datos de manera rápida y fácil utilizando las ventajas de Pandas. En el ejemplo anterior utilizamos las etiquetas de columna para acceder a la serie de datos correspondiente (Columna). Para acceder a una fila de datos podemos usar la notación e indexación sugerida por NumPy sin embargo esta manera no es recomendada.
series[0]
/tmp/ipykernel_3675/878281628.py:1: FutureWarning: Series.__getitem__ treating keys as positions is deprecated. In a future version, integer keys will always be treated as labels (consistent with DataFrame behavior). To access a value by position, use `ser.iloc[pos]`
series[0]
0.15
Preferiblemente, para utilizar las potencialidades y ventajas de Pandas, se recomienda usar las etiquetas de la fila de la siguiente manera:
series["1982-01-01"]
0.15
Si queremos extraer un intervalo de datos podemos usar las etiquetas de índice de filas usando la notacion [comienzo:fin]
series["2000-01-01":"2001-12-01"]
datetime
2000-01-01 -1.92
2000-02-01 -1.53
2000-03-01 -1.14
2000-04-01 -0.77
2000-05-01 -0.73
2000-06-01 -0.62
2000-07-01 -0.50
2000-08-01 -0.37
2000-09-01 -0.51
2000-10-01 -0.73
2000-11-01 -0.87
2000-12-01 -0.98
2001-01-01 -0.83
2001-02-01 -0.61
2001-03-01 -0.38
2001-04-01 -0.26
2001-05-01 -0.25
2001-06-01 0.03
2001-07-01 0.10
2001-08-01 0.05
2001-09-01 -0.17
2001-10-01 -0.10
2001-11-01 -0.20
2001-12-01 -0.40
Name: Nino34anom, dtype: float64
Python tiene incorporado una clase muy útil para hacer selección de datos llamada slice. Esta función nos permite crear un conjunto de índices especificados por los argumentos comienzo, fin y paso usando la notación [comiezo, fin, paso]
slice("2000-01-01", "2001-12-01")
slice('2000-01-01', '2001-12-01', None)
Seleccionemos nuestros datos utilizando el método slice
series[slice("2000-01-01", "2001-12-01")]
datetime
2000-01-01 -1.92
2000-02-01 -1.53
2000-03-01 -1.14
2000-04-01 -0.77
2000-05-01 -0.73
2000-06-01 -0.62
2000-07-01 -0.50
2000-08-01 -0.37
2000-09-01 -0.51
2000-10-01 -0.73
2000-11-01 -0.87
2000-12-01 -0.98
2001-01-01 -0.83
2001-02-01 -0.61
2001-03-01 -0.38
2001-04-01 -0.26
2001-05-01 -0.25
2001-06-01 0.03
2001-07-01 0.10
2001-08-01 0.05
2001-09-01 -0.17
2001-10-01 -0.10
2001-11-01 -0.20
2001-12-01 -0.40
Name: Nino34anom, dtype: float64
Adicionalmente, podemos usar el método loc que también nos permite acceder por etiquetas
series.loc["1982-01-01"]
0.15
o su equivalente usando el índice iloc
series.iloc[0]
0.15
Ahora que sabemos los fundamentos básicos de seleccion de datos en series temporales, podemos pasar a seleccionar datos en DataFrames. Para accerder a una sola columna usamos la notación de diccionario llave/valor como vimos anteriormente
df["Nino34anom"].head() # para una sola columna
datetime
1982-01-01 0.15
1982-02-01 -0.02
1982-03-01 -0.02
1982-04-01 0.24
1982-05-01 0.69
Name: Nino34anom, dtype: float64
Para seleccionar multiples columnas se utiliza doble corchete cuadrado anidado [['col1', 'col2', ..., 'coln']]
df[["Nino34", "Nino34anom"]].head()
| Nino34 | Nino34anom | |
|---|---|---|
| datetime | ||
| 1982-01-01 | 26.72 | 0.15 |
| 1982-02-01 | 26.70 | -0.02 |
| 1982-03-01 | 27.20 | -0.02 |
| 1982-04-01 | 28.02 | 0.24 |
| 1982-05-01 | 28.54 | 0.69 |
Seleccionar datos usando etiquetas de fila y columnas se puede llevar a cabo usando el método loc de la siguiente manera: .loc[filas, columnas]
df.loc["1982-04-01", "Nino34"]
28.02
Si seleccionamos datos únicamente por la etiqueta de la fila nos retornará una serie con los datos de todas las columnas
df.loc["1982-04-01"]
Nino12 24.50
Nino12anom -0.97
Nino3 27.68
Nino3anom 0.18
Nino4 28.92
Nino4anom 0.42
Nino34 28.02
Nino34anom 0.24
Name: 1982-04-01 00:00:00, dtype: float64
Podemos seleccionar un rango de fechas para todas las columnas
df.loc["1982-01-01":"1982-12-01"]
| Nino12 | Nino12anom | Nino3 | Nino3anom | Nino4 | Nino4anom | Nino34 | Nino34anom | |
|---|---|---|---|---|---|---|---|---|
| datetime | ||||||||
| 1982-01-01 | 24.29 | -0.17 | 25.87 | 0.24 | 28.30 | 0.00 | 26.72 | 0.15 |
| 1982-02-01 | 25.49 | -0.58 | 26.38 | 0.01 | 28.21 | 0.11 | 26.70 | -0.02 |
| 1982-03-01 | 25.21 | -1.31 | 26.98 | -0.16 | 28.41 | 0.22 | 27.20 | -0.02 |
| 1982-04-01 | 24.50 | -0.97 | 27.68 | 0.18 | 28.92 | 0.42 | 28.02 | 0.24 |
| 1982-05-01 | 23.97 | -0.23 | 27.79 | 0.71 | 29.49 | 0.70 | 28.54 | 0.69 |
| 1982-06-01 | 22.89 | 0.07 | 27.46 | 1.03 | 29.76 | 0.92 | 28.75 | 1.10 |
| 1982-07-01 | 22.47 | 0.87 | 26.44 | 0.82 | 29.38 | 0.58 | 28.10 | 0.88 |
| 1982-08-01 | 21.75 | 1.10 | 26.15 | 1.16 | 29.04 | 0.36 | 27.93 | 1.11 |
| 1982-09-01 | 21.80 | 1.44 | 26.52 | 1.67 | 29.16 | 0.47 | 28.11 | 1.39 |
| 1982-10-01 | 22.94 | 2.12 | 27.11 | 2.19 | 29.38 | 0.72 | 28.64 | 1.95 |
| 1982-11-01 | 24.59 | 3.00 | 27.62 | 2.64 | 29.23 | 0.60 | 28.81 | 2.16 |
| 1982-12-01 | 26.13 | 3.34 | 28.39 | 3.25 | 29.15 | 0.66 | 29.21 | 2.64 |
De igual modo podemos seleccionar un set de datos combinando los métodos anteriormente mecionados
df.loc["1982-01-01":"1982-12-01", ["Nino34", "Nino34anom"]]
| Nino34 | Nino34anom | |
|---|---|---|
| datetime | ||
| 1982-01-01 | 26.72 | 0.15 |
| 1982-02-01 | 26.70 | -0.02 |
| 1982-03-01 | 27.20 | -0.02 |
| 1982-04-01 | 28.02 | 0.24 |
| 1982-05-01 | 28.54 | 0.69 |
| 1982-06-01 | 28.75 | 1.10 |
| 1982-07-01 | 28.10 | 0.88 |
| 1982-08-01 | 27.93 | 1.11 |
| 1982-09-01 | 28.11 | 1.39 |
| 1982-10-01 | 28.64 | 1.95 |
| 1982-11-01 | 28.81 | 2.16 |
| 1982-12-01 | 29.21 | 2.64 |
2.4 Análisis exploratorios
Pandas nos permite visualizar las primeras y últimas filas de los DataFrames usando .head() y .tail()
df.head()
| Nino12 | Nino12anom | Nino3 | Nino3anom | Nino4 | Nino4anom | Nino34 | Nino34anom | |
|---|---|---|---|---|---|---|---|---|
| datetime | ||||||||
| 1982-01-01 | 24.29 | -0.17 | 25.87 | 0.24 | 28.30 | 0.00 | 26.72 | 0.15 |
| 1982-02-01 | 25.49 | -0.58 | 26.38 | 0.01 | 28.21 | 0.11 | 26.70 | -0.02 |
| 1982-03-01 | 25.21 | -1.31 | 26.98 | -0.16 | 28.41 | 0.22 | 27.20 | -0.02 |
| 1982-04-01 | 24.50 | -0.97 | 27.68 | 0.18 | 28.92 | 0.42 | 28.02 | 0.24 |
| 1982-05-01 | 23.97 | -0.23 | 27.79 | 0.71 | 29.49 | 0.70 | 28.54 | 0.69 |
df.tail()
| Nino12 | Nino12anom | Nino3 | Nino3anom | Nino4 | Nino4anom | Nino34 | Nino34anom | |
|---|---|---|---|---|---|---|---|---|
| datetime | ||||||||
| 2020-12-01 | 22.16 | -0.60 | 24.38 | -0.83 | 27.65 | -0.95 | 25.53 | -1.12 |
| 2021-01-01 | 23.89 | -0.64 | 25.06 | -0.55 | 27.10 | -1.25 | 25.58 | -0.99 |
| 2021-02-01 | 25.55 | -0.66 | 25.80 | -0.57 | 27.20 | -1.00 | 25.81 | -0.92 |
| 2021-03-01 | 26.48 | -0.26 | 26.80 | -0.39 | 27.79 | -0.55 | 26.75 | -0.51 |
| 2021-04-01 | 24.89 | -0.80 | 26.96 | -0.65 | 28.47 | -0.21 | 27.40 | -0.49 |
Para conocer la información de tipo de datos, número de datos faltantes y otras propiedades del DataFrame podemos usar el método .info()
df.info()
<class 'pandas.core.frame.DataFrame'>
DatetimeIndex: 472 entries, 1982-01-01 to 2021-04-01
Data columns (total 8 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 Nino12 472 non-null float64
1 Nino12anom 472 non-null float64
2 Nino3 472 non-null float64
3 Nino3anom 472 non-null float64
4 Nino4 472 non-null float64
5 Nino4anom 472 non-null float64
6 Nino34 472 non-null float64
7 Nino34anom 472 non-null float64
dtypes: float64(8)
memory usage: 49.4 KB
Para acceder a una descripción estadística rápida del DataFrame podemos usar el método .describe()
df.describe()
| Nino12 | Nino12anom | Nino3 | Nino3anom | Nino4 | Nino4anom | Nino34 | Nino34anom | |
|---|---|---|---|---|---|---|---|---|
| count | 472.000000 | 472.000000 | 472.000000 | 472.000000 | 472.000000 | 472.000000 | 472.000000 | 472.000000 |
| mean | 23.209619 | 0.059725 | 25.936568 | 0.039428 | 28.625064 | 0.063814 | 27.076780 | 0.034894 |
| std | 2.431522 | 1.157590 | 1.349621 | 0.965464 | 0.755422 | 0.709401 | 1.063004 | 0.947936 |
| min | 18.570000 | -2.100000 | 23.030000 | -2.070000 | 26.430000 | -1.870000 | 24.270000 | -2.380000 |
| 25% | 21.152500 | -0.712500 | 24.850000 | -0.600000 | 28.140000 | -0.430000 | 26.330000 | -0.572500 |
| 50% | 22.980000 | -0.160000 | 25.885000 | -0.115000 | 28.760000 | 0.205000 | 27.100000 | 0.015000 |
| 75% | 25.322500 | 0.515000 | 26.962500 | 0.512500 | 29.190000 | 0.630000 | 27.792500 | 0.565000 |
| max | 29.150000 | 4.620000 | 29.140000 | 3.620000 | 30.300000 | 1.670000 | 29.600000 | 2.950000 |
Podemos calcular el valor medio de una serie o un DataFrame usando el método .mean()
df["Nino34anom"].mean()
0.03489406779661016
df.mean()
Nino12 23.209619
Nino12anom 0.059725
Nino3 25.936568
Nino3anom 0.039428
Nino4 28.625064
Nino4anom 0.063814
Nino34 27.076780
Nino34anom 0.034894
dtype: float64
El método .mean() calcula el valor medio a lo largo de las columnas, sin embargo podemos calcular la media a lo largo de las filas usando el argumento axis
df.mean(axis=1)
datetime
1982-01-01 13.17500
1982-02-01 13.28750
1982-03-01 13.31625
1982-04-01 13.62375
1982-05-01 13.95750
...
2020-12-01 12.02750
2021-01-01 12.27500
2021-02-01 12.65125
2021-03-01 13.26375
2021-04-01 13.19625
Length: 472, dtype: float64
De manera similar, podemos calcular la desviación estándar usando el método .std()
df.std()
Nino12 2.431522
Nino12anom 1.157590
Nino3 1.349621
Nino3anom 0.965464
Nino4 0.755422
Nino4anom 0.709401
Nino34 1.063004
Nino34anom 0.947936
dtype: float64
Para más funciones y operaciones se puede consultar la documentación oficial de Pandas.
2.6 Gráficos rápidos
Pandas nos permite generar gráficos rápidos usando el método .plot.
df.Nino34.plot();
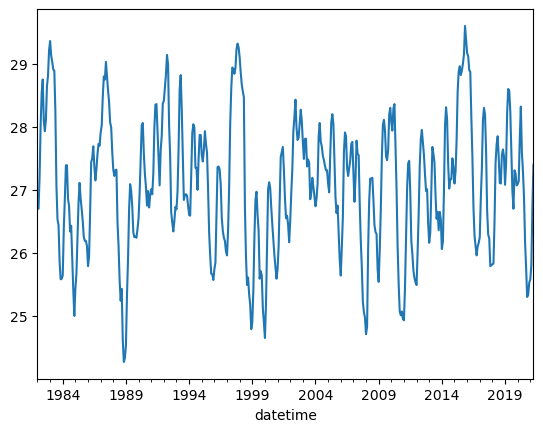
el método .plot() genera un gráfico de tipo linea simple. Sin embargo, podemos generar gráficos más complejos utilizando otros métodos como .hist que retorna un histográma
df.Nino34.plot.hist();
# df[['Nino12', 'Nino34']].plot.hist();
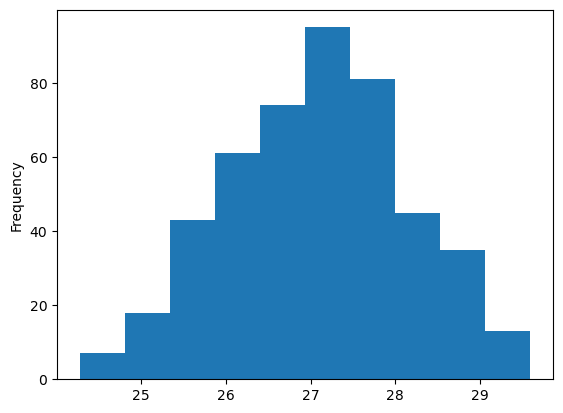
O simplemente un diagrama de cajas que nos permitiría visualizar los datos de otra manera.
df[["Nino12", "Nino34"]].plot.box();
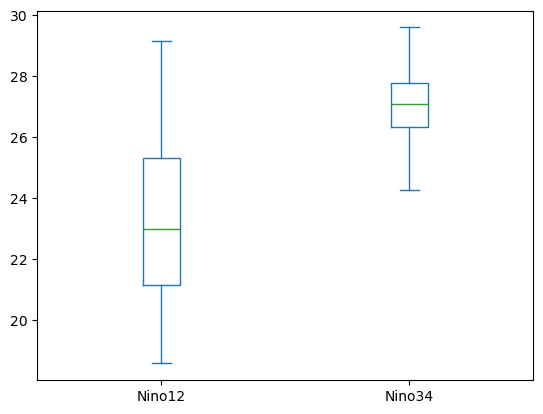
Para mas información de gráficos pueden consultar este link.
3. Xarray
Xarray es una librería ampliamente utilizada en el área de las geociencias para el análisis de datos multidimensionales (2-D, 3-D, …, N-D). Al igual que Pandas las principales ventajas de Xarray radican en la manipulación de datos mediante etiquetas y coordenadas en cada una de sus dimensiones.
3.1 Xarray Datarray
Un Datarray es uno de los elementos más básicos de Xarray. Es similar a un numpy ndarray pero con las ventajas de tener coordenadas y atributuos. Veámos a qué hacen referencia estos dos aspectos.
Creemos una matriz aleatorea de temperaturas en grados Kelvin usando np.random.randn
data = 283 + 5 * np.random.randn(5, 3, 4)
data
array([[[287.87755624, 275.91963442, 277.87309247, 276.69263921],
[282.56878386, 290.85745874, 276.92356699, 280.69811637],
[292.79027497, 274.32176611, 285.60354887, 281.16514173]],
[[291.12212505, 279.15078687, 278.32551482, 281.85382458],
[285.10396737, 286.83636715, 279.95691408, 272.39701826],
[286.79423417, 276.86956424, 281.44343178, 283.0223496 ]],
[[281.58941736, 278.08241595, 282.90405651, 283.02512777],
[280.46491261, 291.7964626 , 277.40373448, 280.12522522],
[288.59798337, 281.52384386, 286.404524 , 278.38774246]],
[[279.10658423, 282.29602275, 280.57023329, 286.75118019],
[280.57066666, 283.27367412, 292.21456115, 276.25085766],
[291.38463358, 290.69912818, 292.36657515, 285.29245063]],
[[280.42917345, 273.60188692, 285.81598692, 285.1425535 ],
[276.65672513, 278.02379509, 286.0873545 , 276.94489524],
[269.951582 , 283.47688802, 288.06938248, 288.28651941]]])
las dimensiones y forma de la matriz son:
data.ndim, data.shape
(3, (5, 3, 4))
Hasta ahora solo hemos creado un arreglo matricial. Ahora, como primer intento, creemos una Datarray usando este numpy.array
temp = xr.DataArray(data)
temp
<xarray.DataArray (dim_0: 5, dim_1: 3, dim_2: 4)>
array([[[287.87755624, 275.91963442, 277.87309247, 276.69263921],
[282.56878386, 290.85745874, 276.92356699, 280.69811637],
[292.79027497, 274.32176611, 285.60354887, 281.16514173]],
[[291.12212505, 279.15078687, 278.32551482, 281.85382458],
[285.10396737, 286.83636715, 279.95691408, 272.39701826],
[286.79423417, 276.86956424, 281.44343178, 283.0223496 ]],
[[281.58941736, 278.08241595, 282.90405651, 283.02512777],
[280.46491261, 291.7964626 , 277.40373448, 280.12522522],
[288.59798337, 281.52384386, 286.404524 , 278.38774246]],
[[279.10658423, 282.29602275, 280.57023329, 286.75118019],
[280.57066666, 283.27367412, 292.21456115, 276.25085766],
[291.38463358, 290.69912818, 292.36657515, 285.29245063]],
[[280.42917345, 273.60188692, 285.81598692, 285.1425535 ],
[276.65672513, 278.02379509, 286.0873545 , 276.94489524],
[269.951582 , 283.47688802, 288.06938248, 288.28651941]]])
Dimensions without coordinates: dim_0, dim_1, dim_2Dos cosas para anotar:
Dado que
NumPyno utiliza dimensiones ni etiquetas, nuesto nuevoDatarraytoma nombres en sus dimensiones (dim_0,dim_1ydim_2).De ser ejecutado en
Jupyter Notebookse genera una visualización completa de los datos contenidos dentro delDatarrayque nos permite explorar ladata, lascoordenadas, losíndicesy losatributos.
Ahora tratemos de poner nombre a las dimensiones para dar claridad/autodescripción al objeto. Esto lo podemos lograr pasando el argumento dims durante la creación del Datarray
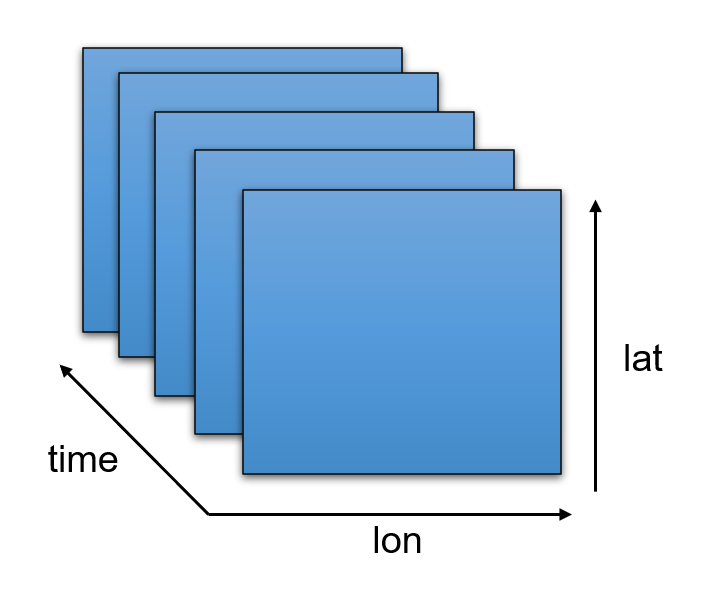
temp = xr.DataArray(data, dims=["time", "lat", "lon"])
temp
<xarray.DataArray (time: 5, lat: 3, lon: 4)>
array([[[287.87755624, 275.91963442, 277.87309247, 276.69263921],
[282.56878386, 290.85745874, 276.92356699, 280.69811637],
[292.79027497, 274.32176611, 285.60354887, 281.16514173]],
[[291.12212505, 279.15078687, 278.32551482, 281.85382458],
[285.10396737, 286.83636715, 279.95691408, 272.39701826],
[286.79423417, 276.86956424, 281.44343178, 283.0223496 ]],
[[281.58941736, 278.08241595, 282.90405651, 283.02512777],
[280.46491261, 291.7964626 , 277.40373448, 280.12522522],
[288.59798337, 281.52384386, 286.404524 , 278.38774246]],
[[279.10658423, 282.29602275, 280.57023329, 286.75118019],
[280.57066666, 283.27367412, 292.21456115, 276.25085766],
[291.38463358, 290.69912818, 292.36657515, 285.29245063]],
[[280.42917345, 273.60188692, 285.81598692, 285.1425535 ],
[276.65672513, 278.02379509, 286.0873545 , 276.94489524],
[269.951582 , 283.47688802, 288.06938248, 288.28651941]]])
Dimensions without coordinates: time, lat, lonCreamos un objeto con datos y dimensiones que gráficamente se podría representar de la siguiente manera
Nuestro Datarray ahora tienen dimesiones de tiempo (time), latitud (lat) y longitud (lon). Ahora podemos hacer de nuestro Datarray algo aún más “poderoso” incluyéndole coordenadas a las dimensiones. Sabemos que las dimensiones son: tiempo, latitud y longitud. Comencemos creando un índice de tiempo que empiece en 2018-01-01 y contenga 5 periodos en total
times = pd.date_range("2018-01-01", periods=5)
times
DatetimeIndex(['2018-01-01', '2018-01-02', '2018-01-03', '2018-01-04',
'2018-01-05'],
dtype='datetime64[ns]', freq='D')
Ahora asociemos la longitud y latitud a una ubicación geográfica:
lons = np.linspace(-120, -90, 4)
lats = np.linspace(25, 55, 3)
lons, lats
(array([-120., -110., -100., -90.]), array([25., 40., 55.]))
Usando toda la información podemos crear nuestro Datarray aprovechando las ventajas que nos brinda Xarray
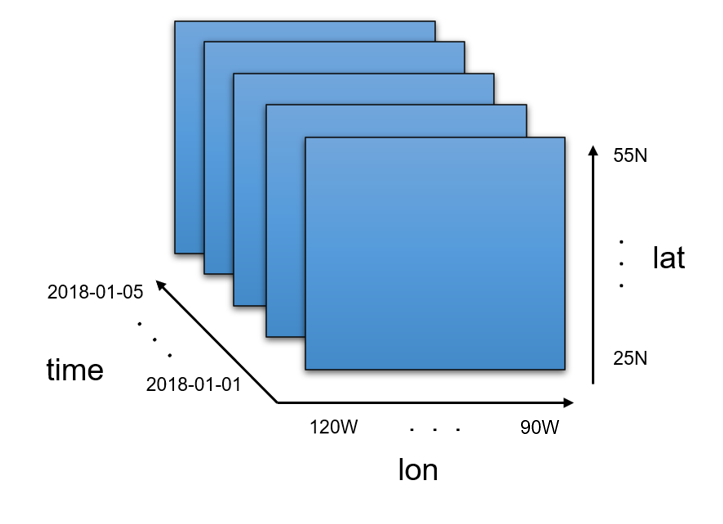
temp = xr.DataArray(data, coords=[times, lats, lons], dims=["time", "lat", "lon"])
temp
<xarray.DataArray (time: 5, lat: 3, lon: 4)>
array([[[287.87755624, 275.91963442, 277.87309247, 276.69263921],
[282.56878386, 290.85745874, 276.92356699, 280.69811637],
[292.79027497, 274.32176611, 285.60354887, 281.16514173]],
[[291.12212505, 279.15078687, 278.32551482, 281.85382458],
[285.10396737, 286.83636715, 279.95691408, 272.39701826],
[286.79423417, 276.86956424, 281.44343178, 283.0223496 ]],
[[281.58941736, 278.08241595, 282.90405651, 283.02512777],
[280.46491261, 291.7964626 , 277.40373448, 280.12522522],
[288.59798337, 281.52384386, 286.404524 , 278.38774246]],
[[279.10658423, 282.29602275, 280.57023329, 286.75118019],
[280.57066666, 283.27367412, 292.21456115, 276.25085766],
[291.38463358, 290.69912818, 292.36657515, 285.29245063]],
[[280.42917345, 273.60188692, 285.81598692, 285.1425535 ],
[276.65672513, 278.02379509, 286.0873545 , 276.94489524],
[269.951582 , 283.47688802, 288.06938248, 288.28651941]]])
Coordinates:
* time (time) datetime64[ns] 2018-01-01 2018-01-02 ... 2018-01-05
* lat (lat) float64 25.0 40.0 55.0
* lon (lon) float64 -120.0 -110.0 -100.0 -90.0Visualmente sería algo así
Podemos establecer atributos útiles, como las unidades de la variable o un nombre estándar, que nos permitan describir el conjunto de datos en el Datarray
temp.attrs["units"] = "kelvin"
temp.attrs["standard_name"] = "air_temperature"
temp
<xarray.DataArray (time: 5, lat: 3, lon: 4)>
array([[[287.87755624, 275.91963442, 277.87309247, 276.69263921],
[282.56878386, 290.85745874, 276.92356699, 280.69811637],
[292.79027497, 274.32176611, 285.60354887, 281.16514173]],
[[291.12212505, 279.15078687, 278.32551482, 281.85382458],
[285.10396737, 286.83636715, 279.95691408, 272.39701826],
[286.79423417, 276.86956424, 281.44343178, 283.0223496 ]],
[[281.58941736, 278.08241595, 282.90405651, 283.02512777],
[280.46491261, 291.7964626 , 277.40373448, 280.12522522],
[288.59798337, 281.52384386, 286.404524 , 278.38774246]],
[[279.10658423, 282.29602275, 280.57023329, 286.75118019],
[280.57066666, 283.27367412, 292.21456115, 276.25085766],
[291.38463358, 290.69912818, 292.36657515, 285.29245063]],
[[280.42917345, 273.60188692, 285.81598692, 285.1425535 ],
[276.65672513, 278.02379509, 286.0873545 , 276.94489524],
[269.951582 , 283.47688802, 288.06938248, 288.28651941]]])
Coordinates:
* time (time) datetime64[ns] 2018-01-01 2018-01-02 ... 2018-01-05
* lat (lat) float64 25.0 40.0 55.0
* lon (lon) float64 -120.0 -110.0 -100.0 -90.0
Attributes:
units: kelvin
standard_name: air_temperature3.2 Xarray Dataset
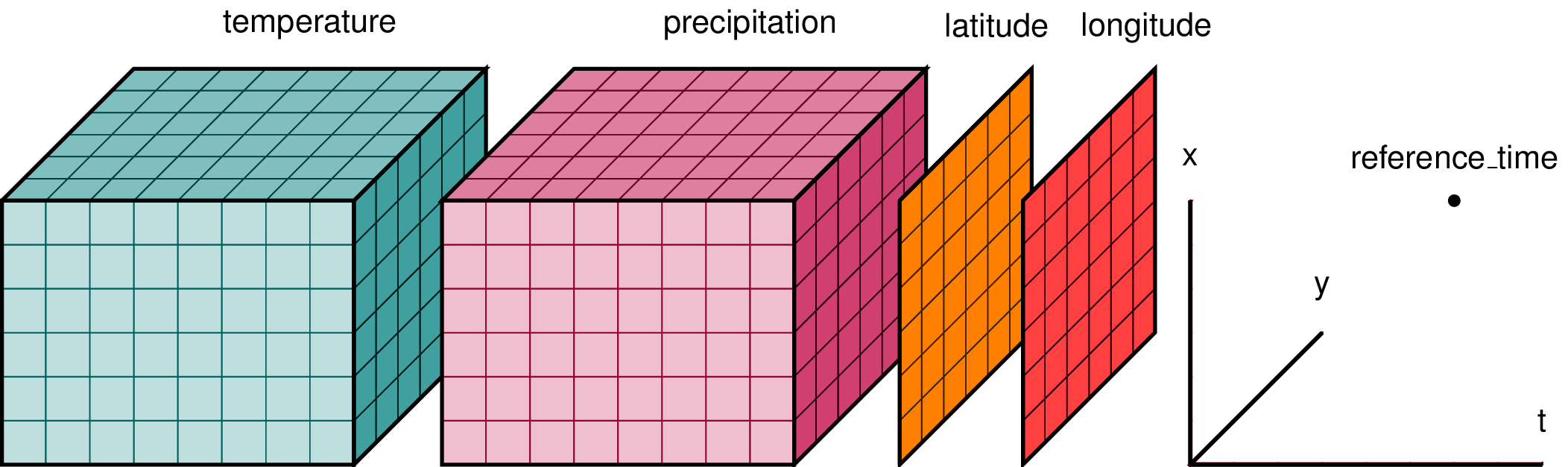
Un Dataset no es mas que un contenedor de multiples Datarrays que pueden o no compartir dimensiones y coordenadas. Estos Datasets son más comunes en la comunidad científica dado que permite almacenar mas de una variable.
A continuación, vamos a crear una variable de presión y humedad relativa, que junto a la temperatura conformarán nuestro Dataset.
pressure_data = 1000.0 + 5 * np.random.randn(5, 3, 4)
pressure = xr.DataArray(
pressure_data, coords=[times, lats, lons], dims=["time", "lat", "lon"]
)
pressure.attrs["units"] = "hPa"
pressure.attrs["standard_name"] = "air_pressure"
pressure
<xarray.DataArray (time: 5, lat: 3, lon: 4)>
array([[[1000.59026733, 1007.79182912, 994.88908546, 997.80983353],
[ 996.47593487, 999.74123255, 1000.26134142, 1000.34471008],
[ 997.59365125, 1003.38338055, 996.60530168, 992.94319677]],
[[1002.43311786, 992.51993314, 1002.19429728, 998.94867604],
[ 995.30034481, 1000.62267702, 995.27309615, 992.11682561],
[ 999.8896964 , 995.10352232, 999.5503185 , 999.86276817]],
[[1004.74511232, 1001.91041223, 993.62663213, 1000.42216916],
[1000.88879683, 996.01582129, 990.22960482, 1002.12398633],
[1005.27595717, 1001.43006161, 992.88130165, 1006.33445873]],
[[ 994.84882263, 996.59091324, 997.65508599, 1001.28686964],
[ 998.1338952 , 999.22203503, 1002.93428368, 1006.41271922],
[ 993.91652817, 999.61211606, 1004.06380345, 997.41250573]],
[[1000.5689944 , 996.51022926, 999.45196581, 1000.6425751 ],
[ 987.81604244, 1002.97780765, 997.65509264, 997.41472409],
[ 995.65473943, 998.93714963, 1001.41371419, 999.41049549]]])
Coordinates:
* time (time) datetime64[ns] 2018-01-01 2018-01-02 ... 2018-01-05
* lat (lat) float64 25.0 40.0 55.0
* lon (lon) float64 -120.0 -110.0 -100.0 -90.0
Attributes:
units: hPa
standard_name: air_pressureLa humedad relativa sólo dependerá del tiempo
hr_data = np.random.uniform(low=60, high=100, size=5)
hr_data
array([73.79989203, 92.39480706, 60.22376359, 84.02715899, 98.99887537])
hr = xr.DataArray(hr_data, coords=[times], dims=["time"])
hr.attrs["units"] = "%"
hr.attrs["standard_name"] = "relative_humidity"
hr
<xarray.DataArray (time: 5)>
array([73.79989203, 92.39480706, 60.22376359, 84.02715899, 98.99887537])
Coordinates:
* time (time) datetime64[ns] 2018-01-01 2018-01-02 ... 2018-01-05
Attributes:
units: %
standard_name: relative_humidityLa manera mas fácil para crear un Dataset es mediante un diccionario de Python donde la llave es el nombre y el valor es el Datarray que ya previamente creamos.
dt = {"Temperature": temp, "Pressure": pressure, "HR": hr}
dt
{'Temperature': <xarray.DataArray (time: 5, lat: 3, lon: 4)>
array([[[287.87755624, 275.91963442, 277.87309247, 276.69263921],
[282.56878386, 290.85745874, 276.92356699, 280.69811637],
[292.79027497, 274.32176611, 285.60354887, 281.16514173]],
[[291.12212505, 279.15078687, 278.32551482, 281.85382458],
[285.10396737, 286.83636715, 279.95691408, 272.39701826],
[286.79423417, 276.86956424, 281.44343178, 283.0223496 ]],
[[281.58941736, 278.08241595, 282.90405651, 283.02512777],
[280.46491261, 291.7964626 , 277.40373448, 280.12522522],
[288.59798337, 281.52384386, 286.404524 , 278.38774246]],
[[279.10658423, 282.29602275, 280.57023329, 286.75118019],
[280.57066666, 283.27367412, 292.21456115, 276.25085766],
[291.38463358, 290.69912818, 292.36657515, 285.29245063]],
[[280.42917345, 273.60188692, 285.81598692, 285.1425535 ],
[276.65672513, 278.02379509, 286.0873545 , 276.94489524],
[269.951582 , 283.47688802, 288.06938248, 288.28651941]]])
Coordinates:
* time (time) datetime64[ns] 2018-01-01 2018-01-02 ... 2018-01-05
* lat (lat) float64 25.0 40.0 55.0
* lon (lon) float64 -120.0 -110.0 -100.0 -90.0
Attributes:
units: kelvin
standard_name: air_temperature,
'Pressure': <xarray.DataArray (time: 5, lat: 3, lon: 4)>
array([[[1000.59026733, 1007.79182912, 994.88908546, 997.80983353],
[ 996.47593487, 999.74123255, 1000.26134142, 1000.34471008],
[ 997.59365125, 1003.38338055, 996.60530168, 992.94319677]],
[[1002.43311786, 992.51993314, 1002.19429728, 998.94867604],
[ 995.30034481, 1000.62267702, 995.27309615, 992.11682561],
[ 999.8896964 , 995.10352232, 999.5503185 , 999.86276817]],
[[1004.74511232, 1001.91041223, 993.62663213, 1000.42216916],
[1000.88879683, 996.01582129, 990.22960482, 1002.12398633],
[1005.27595717, 1001.43006161, 992.88130165, 1006.33445873]],
[[ 994.84882263, 996.59091324, 997.65508599, 1001.28686964],
[ 998.1338952 , 999.22203503, 1002.93428368, 1006.41271922],
[ 993.91652817, 999.61211606, 1004.06380345, 997.41250573]],
[[1000.5689944 , 996.51022926, 999.45196581, 1000.6425751 ],
[ 987.81604244, 1002.97780765, 997.65509264, 997.41472409],
[ 995.65473943, 998.93714963, 1001.41371419, 999.41049549]]])
Coordinates:
* time (time) datetime64[ns] 2018-01-01 2018-01-02 ... 2018-01-05
* lat (lat) float64 25.0 40.0 55.0
* lon (lon) float64 -120.0 -110.0 -100.0 -90.0
Attributes:
units: hPa
standard_name: air_pressure,
'HR': <xarray.DataArray (time: 5)>
array([73.79989203, 92.39480706, 60.22376359, 84.02715899, 98.99887537])
Coordinates:
* time (time) datetime64[ns] 2018-01-01 2018-01-02 ... 2018-01-05
Attributes:
units: %
standard_name: relative_humidity}
Ahora podemos proceder a crear nuestro contenedor de datos Dataset.
ds = xr.Dataset(data_vars=dt)
ds
<xarray.Dataset>
Dimensions: (time: 5, lat: 3, lon: 4)
Coordinates:
* time (time) datetime64[ns] 2018-01-01 2018-01-02 ... 2018-01-05
* lat (lat) float64 25.0 40.0 55.0
* lon (lon) float64 -120.0 -110.0 -100.0 -90.0
Data variables:
Temperature (time, lat, lon) float64 287.9 275.9 277.9 ... 288.1 288.3
Pressure (time, lat, lon) float64 1.001e+03 1.008e+03 ... 999.4
HR (time) float64 73.8 92.39 60.22 84.03 99.0Visualmente, y tomando la imagen de la documentación ofical de Xarray, tenemos que nuestro Dataset tomaría la siguiente forma
3.3 Seleccion de datos y valores por coordenadas
La potencialidad de Xarray radica en la facilidad de seleccionar y acceder a los datos dentro del Dataset. Para seleccionar una variable de interés podemos hacerlo mendiante el método punto
ds.Temperature
<xarray.DataArray 'Temperature' (time: 5, lat: 3, lon: 4)>
array([[[287.87755624, 275.91963442, 277.87309247, 276.69263921],
[282.56878386, 290.85745874, 276.92356699, 280.69811637],
[292.79027497, 274.32176611, 285.60354887, 281.16514173]],
[[291.12212505, 279.15078687, 278.32551482, 281.85382458],
[285.10396737, 286.83636715, 279.95691408, 272.39701826],
[286.79423417, 276.86956424, 281.44343178, 283.0223496 ]],
[[281.58941736, 278.08241595, 282.90405651, 283.02512777],
[280.46491261, 291.7964626 , 277.40373448, 280.12522522],
[288.59798337, 281.52384386, 286.404524 , 278.38774246]],
[[279.10658423, 282.29602275, 280.57023329, 286.75118019],
[280.57066666, 283.27367412, 292.21456115, 276.25085766],
[291.38463358, 290.69912818, 292.36657515, 285.29245063]],
[[280.42917345, 273.60188692, 285.81598692, 285.1425535 ],
[276.65672513, 278.02379509, 286.0873545 , 276.94489524],
[269.951582 , 283.47688802, 288.06938248, 288.28651941]]])
Coordinates:
* time (time) datetime64[ns] 2018-01-01 2018-01-02 ... 2018-01-05
* lat (lat) float64 25.0 40.0 55.0
* lon (lon) float64 -120.0 -110.0 -100.0 -90.0
Attributes:
units: kelvin
standard_name: air_temperatureo usando la notación de diccionarios de Python
ds["Pressure"]
<xarray.DataArray 'Pressure' (time: 5, lat: 3, lon: 4)>
array([[[1000.59026733, 1007.79182912, 994.88908546, 997.80983353],
[ 996.47593487, 999.74123255, 1000.26134142, 1000.34471008],
[ 997.59365125, 1003.38338055, 996.60530168, 992.94319677]],
[[1002.43311786, 992.51993314, 1002.19429728, 998.94867604],
[ 995.30034481, 1000.62267702, 995.27309615, 992.11682561],
[ 999.8896964 , 995.10352232, 999.5503185 , 999.86276817]],
[[1004.74511232, 1001.91041223, 993.62663213, 1000.42216916],
[1000.88879683, 996.01582129, 990.22960482, 1002.12398633],
[1005.27595717, 1001.43006161, 992.88130165, 1006.33445873]],
[[ 994.84882263, 996.59091324, 997.65508599, 1001.28686964],
[ 998.1338952 , 999.22203503, 1002.93428368, 1006.41271922],
[ 993.91652817, 999.61211606, 1004.06380345, 997.41250573]],
[[1000.5689944 , 996.51022926, 999.45196581, 1000.6425751 ],
[ 987.81604244, 1002.97780765, 997.65509264, 997.41472409],
[ 995.65473943, 998.93714963, 1001.41371419, 999.41049549]]])
Coordinates:
* time (time) datetime64[ns] 2018-01-01 2018-01-02 ... 2018-01-05
* lat (lat) float64 25.0 40.0 55.0
* lon (lon) float64 -120.0 -110.0 -100.0 -90.0
Attributes:
units: hPa
standard_name: air_pressurePodemos seleccionar y filtrar datos dentro del Dataset usando las coordenadas asignadas. El método .sel nos permite realizar selecciones (slicing) a lo largo de las coordenadas
ds.sel(time="2018-01-01")
<xarray.Dataset>
Dimensions: (lat: 3, lon: 4)
Coordinates:
time datetime64[ns] 2018-01-01
* lat (lat) float64 25.0 40.0 55.0
* lon (lon) float64 -120.0 -110.0 -100.0 -90.0
Data variables:
Temperature (lat, lon) float64 287.9 275.9 277.9 ... 274.3 285.6 281.2
Pressure (lat, lon) float64 1.001e+03 1.008e+03 994.9 ... 996.6 992.9
HR float64 73.8El método sel puede recibir una o más coordenadas a la hora de realizar nuestra selección
ds.sel(time="2018-01-01", lat=25, lon=-120)
<xarray.Dataset>
Dimensions: ()
Coordinates:
time datetime64[ns] 2018-01-01
lat float64 25.0
lon float64 -120.0
Data variables:
Temperature float64 287.9
Pressure float64 1.001e+03
HR float64 73.8Para seleccionar un rango de datos podemos utilzar el método slice como lo vimos con Pandas
ds.sel(time=slice("2018-01-01", "2018-01-03"))
<xarray.Dataset>
Dimensions: (time: 3, lat: 3, lon: 4)
Coordinates:
* time (time) datetime64[ns] 2018-01-01 2018-01-02 2018-01-03
* lat (lat) float64 25.0 40.0 55.0
* lon (lon) float64 -120.0 -110.0 -100.0 -90.0
Data variables:
Temperature (time, lat, lon) float64 287.9 275.9 277.9 ... 286.4 278.4
Pressure (time, lat, lon) float64 1.001e+03 1.008e+03 ... 1.006e+03
HR (time) float64 73.8 92.39 60.22¿Qué pasa si, por ejemplo, los datos en las coordenadas no coinciden exactamente con nuestra consulta? Podemos pasar ciertos parametros como method y torelance que nos permiten buscar el dato más cercano
ds.sel(time="2018-01-07", method="nearest", tolerance=timedelta(days=2))
<xarray.Dataset>
Dimensions: (lat: 3, lon: 4)
Coordinates:
time datetime64[ns] 2018-01-05
* lat (lat) float64 25.0 40.0 55.0
* lon (lon) float64 -120.0 -110.0 -100.0 -90.0
Data variables:
Temperature (lat, lon) float64 280.4 273.6 285.8 ... 283.5 288.1 288.3
Pressure (lat, lon) float64 1.001e+03 996.5 999.5 ... 1.001e+03 999.4
HR float64 99.0para el caso de la longitud o latitud
ds.sel(lat=35, method="nearest")
<xarray.Dataset>
Dimensions: (time: 5, lon: 4)
Coordinates:
* time (time) datetime64[ns] 2018-01-01 2018-01-02 ... 2018-01-05
lat float64 40.0
* lon (lon) float64 -120.0 -110.0 -100.0 -90.0
Data variables:
Temperature (time, lon) float64 282.6 290.9 276.9 ... 278.0 286.1 276.9
Pressure (time, lon) float64 996.5 999.7 1e+03 ... 1.003e+03 997.7 997.4
HR (time) float64 73.8 92.39 60.22 84.03 99.0Al igual que con Numpy, podemos realizar selección de los datos por su índice usando el método isel
ds.isel(lat=0)
<xarray.Dataset>
Dimensions: (time: 5, lon: 4)
Coordinates:
* time (time) datetime64[ns] 2018-01-01 2018-01-02 ... 2018-01-05
lat float64 25.0
* lon (lon) float64 -120.0 -110.0 -100.0 -90.0
Data variables:
Temperature (time, lon) float64 287.9 275.9 277.9 ... 273.6 285.8 285.1
Pressure (time, lon) float64 1.001e+03 1.008e+03 ... 999.5 1.001e+03
HR (time) float64 73.8 92.39 60.22 84.03 99.03.3 Operaciones Básicas
Xarray nos permite aplicar operaciones básicas como la media mean, desviación estándar std, quantiles quantile entre muchas otras operaciones a lo largo de una dimensión determinada. En el siguiente ejemplo aplicaremos la media a lo largo de las 5 estampas de tiempo
ds.Temperature.mean("time")
<xarray.DataArray 'Temperature' (lat: 3, lon: 4)>
array([[284.02497127, 277.81014938, 281.0977768 , 282.69306505],
[281.07301113, 286.15755154, 282.51722624, 277.28322255],
[285.90374162, 281.37823808, 286.77749246, 283.23084077]])
Coordinates:
* lat (lat) float64 25.0 40.0 55.0
* lon (lon) float64 -120.0 -110.0 -100.0 -90.0De igual manera lo podemos aplicar a lo largo de la latitud o la longitud
ds.Temperature.mean("lon")
<xarray.DataArray 'Temperature' (time: 5, lat: 3)>
array([[279.59073058, 282.76198149, 283.47018292],
[282.61306283, 281.07356671, 282.03239495],
[281.4002544 , 282.44758373, 283.72852342],
[282.18100512, 283.0774399 , 289.93569689],
[281.2474002 , 279.42819249, 282.44609298]])
Coordinates:
* time (time) datetime64[ns] 2018-01-01 2018-01-02 ... 2018-01-05
* lat (lat) float64 25.0 40.0 55.0Xarray tambien nos permite realizar interpolaciones a puntos que no estan explícitamente definidos dentro de las coordenadas. Tratemos de generar una serie de temperatura para el punto con lat=40 y lon=-105
ds.Temperature.interp(lon=-105, lat=40)
<xarray.DataArray 'Temperature' (time: 5)>
array([283.89051286, 283.39664061, 284.60009854, 287.74411764,
282.0555748 ])
Coordinates:
* time (time) datetime64[ns] 2018-01-01 2018-01-02 ... 2018-01-05
lon int64 -105
lat int64 40
Attributes:
units: kelvin
standard_name: air_temperature3.5 Generación de gráficas
Al igual que Pandas, Xarray posee un módulo autocontenido para realizar gráficos sin necesidad de usar la librería Matplotlib. Para esto debemos usar el método .plot. Generemos un gráfico para un punto cualquiera donde se vea la evolución de la temperatura en el tiempo.
ds.Temperature.sel(lat=40, lon=-100, method="nearest").plot()
[<matplotlib.lines.Line2D at 0x7f2e92734d50>]
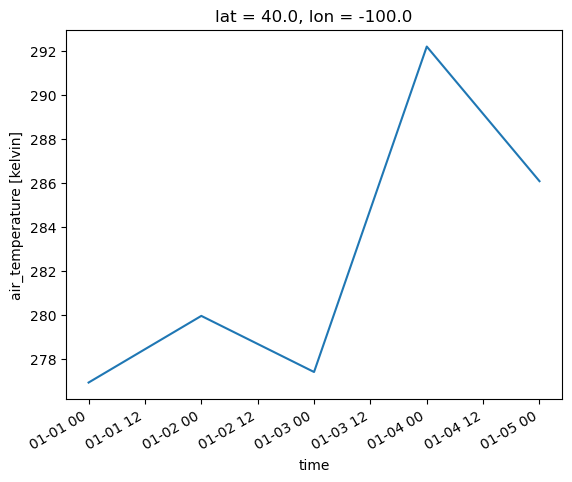
Ahora un gráfico donde se vea la distribución espacial de la temperatura para un tiempo en específico t=3
ds.Temperature.isel(time=3).plot()
<matplotlib.collections.QuadMesh at 0x7f2e911ffb50>
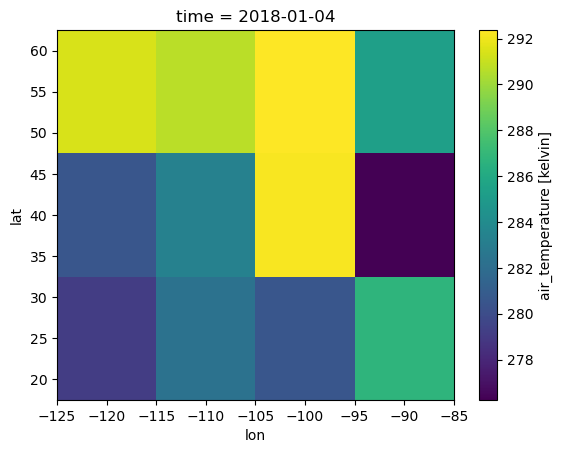
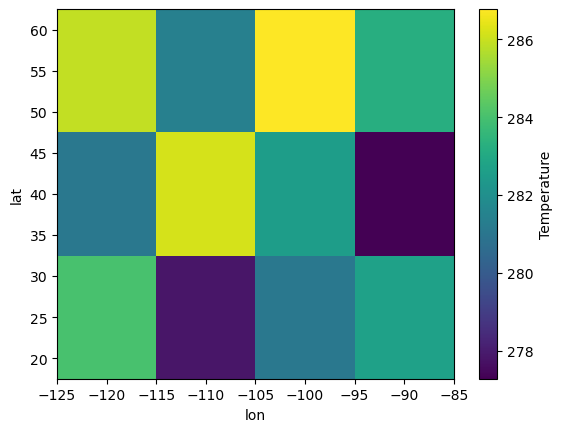
O simplemente el gráfico espacial de la temperatura media a lo largo de la dimensión temporal
ds.Temperature.mean("time").plot()
<matplotlib.collections.QuadMesh at 0x7f2e91131dd0>
3.6 Lectura de datos en formato netCDF
Xarray nos permite leer archivo en formato netCDF, GRIB, Zarr, entre muchos otros. A continuación, mostraremos un ejemplo de la lectura de datos de Temperatura Superficial del Mar (TSM) proveniente del Modelo del Sistema Terrestre Comunitario v2 (CESM2).
filepath = DATASETS.fetch("CESM2_sst_data.nc")
ds = xr.open_dataset(filepath)
ds
Downloading file 'CESM2_sst_data.nc' from 'https://github.com/ProjectPythia/pythia-datasets/raw/main/data/CESM2_sst_data.nc' to '/home/runner/.cache/pythia-datasets'.
0.3.0
/usr/share/miniconda3/envs/atmoscol2023/lib/python3.11/site-packages/xarray/conventions.py:428: SerializationWarning: variable 'tos' has multiple fill values {1e+20, 1e+20}, decoding all values to NaN.
new_vars[k] = decode_cf_variable(
<xarray.Dataset>
Dimensions: (time: 180, d2: 2, lat: 180, lon: 360)
Coordinates:
* time (time) object 2000-01-15 12:00:00 ... 2014-12-15 12:00:00
* lat (lat) float64 -89.5 -88.5 -87.5 -86.5 ... 86.5 87.5 88.5 89.5
* lon (lon) float64 0.5 1.5 2.5 3.5 4.5 ... 356.5 357.5 358.5 359.5
Dimensions without coordinates: d2
Data variables:
time_bnds (time, d2) object ...
lat_bnds (lat, d2) float64 ...
lon_bnds (lon, d2) float64 ...
tos (time, lat, lon) float32 ...
Attributes: (12/45)
Conventions: CF-1.7 CMIP-6.2
activity_id: CMIP
branch_method: standard
branch_time_in_child: 674885.0
branch_time_in_parent: 219000.0
case_id: 972
... ...
sub_experiment_id: none
table_id: Omon
tracking_id: hdl:21.14100/2975ffd3-1d7b-47e3-961a-33f212ea4eb2
variable_id: tos
variant_info: CMIP6 20th century experiments (1850-2014) with C...
variant_label: r11i1p1f1ds["time"] = ds.indexes["time"].to_datetimeindex()
/tmp/ipykernel_3675/3354600616.py:1: RuntimeWarning: Converting a CFTimeIndex with dates from a non-standard calendar, 'noleap', to a pandas.DatetimeIndex, which uses dates from the standard calendar. This may lead to subtle errors in operations that depend on the length of time between dates.
ds["time"] = ds.indexes["time"].to_datetimeindex()
# ds.time
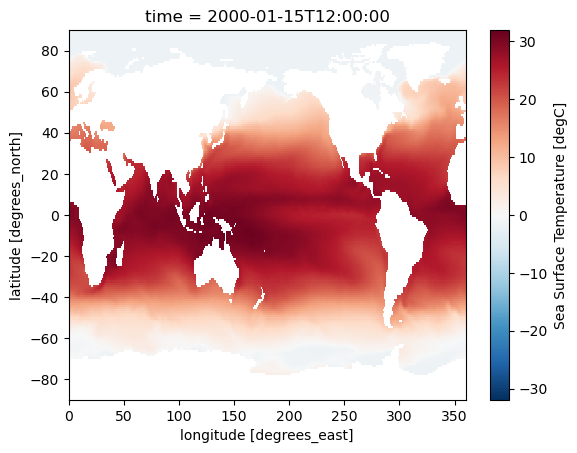
Podemos acceder a la variable tos y generar un plot para visualizar su contenido
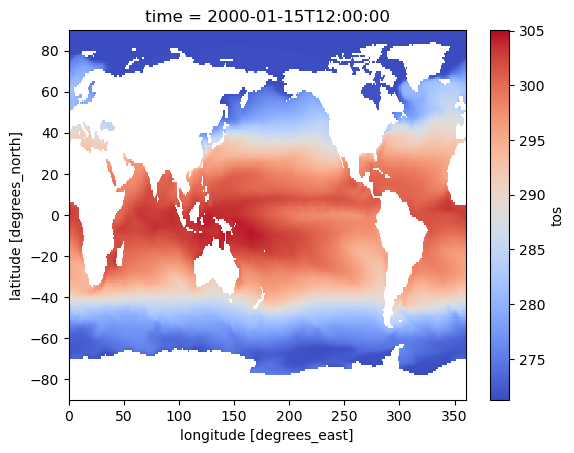
ds.tos.isel(time=0).plot()
<matplotlib.collections.QuadMesh at 0x7f2e87837a50>
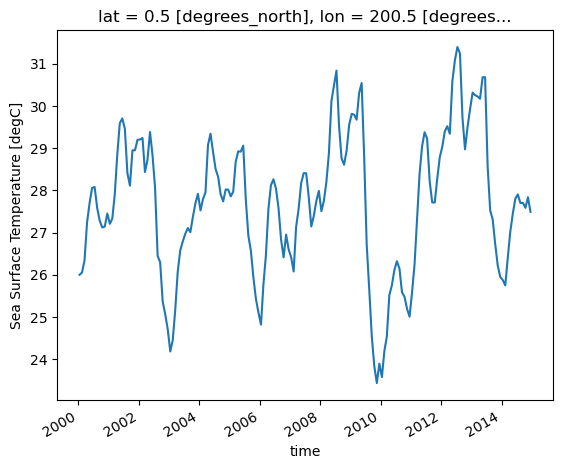
ds.tos.sel(lon=200, lat=0, method="nearest").plot()
[<matplotlib.lines.Line2D at 0x7f2e87801990>]
Para pasar de grados Celcius a Kelvin podemos realizar la siguiente operación
temp_kel = ds.tos + 273.15
temp_kel.isel(time=0).plot(cmap="coolwarm")
<matplotlib.collections.QuadMesh at 0x7f2e877b78d0>
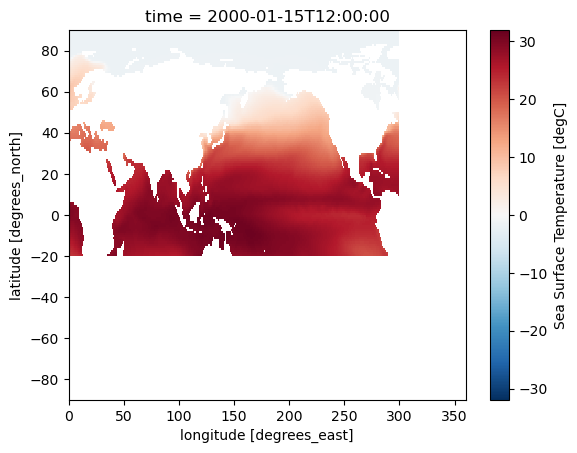
Podemos aplicar una máscara a la data para seleccionar o visualizar algunos datos de interés. Esto lo podemos llevar a cabo utilizando el método .where
ds.tos.isel(time=0).where(ds.lat >= -20).where(ds.lon < 300).plot()
<matplotlib.collections.QuadMesh at 0x7f2e8482e650>
Con esto finalizamos nuestro tutorial básico en NumPy, Pandas y Xarray.
Conclusiones
En el presente cuadernillo aprendimos aspectos básicos como la creación, operación y selección de arrays (NumPy), dataframes (Pandas) y datasets/datarrays (Xarray). Estas librerías nos permitirán entonces manipular cualquier dato de caracter ambiental utilizando las potencialidades de cada librería.