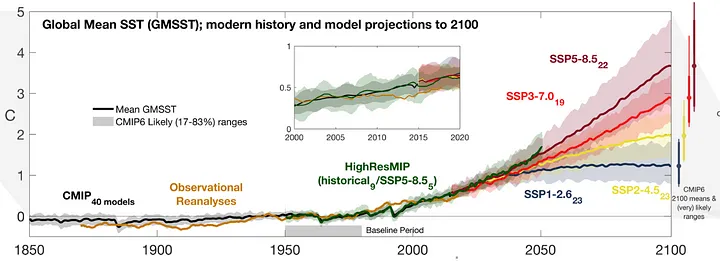
Cambio Climático: Ensambles multimodelo
Introducción
En este cuadernillo (Notebook) aprenderemos:
Breve introducción a los escenarios de Cambio Climático.
Proyecto de inter-comparación de modelos de clima acoplados - CMIP.
Acceso a los datos CMIP6 en formato Zarr.
Reproduccion de la gráfica de la Temperatura Media Global de la Superficie del Mar - CMIP6.
Prerequisitos
Conceptos |
Importancia |
Notas |
|---|---|---|
Necesario |
Manejo de datos multidimensionales espacializados |
|
Necesario |
Generación de gráficas |
|
Necesario |
Ejemplos y análisis de CMIP6 |
|
Útil |
Familiaridad con la estructura de datos y metadatos. |
|
Útil |
Cátalogo que nos permite acceder a datos de diversas fuentes |
Tiempo de aprendizaje: 30 minutos.
Librerías
Importamos las librerías que usaremos a lo largo de este cuadernillo.
import matplotlib.pyplot as plt
import numpy as np
import xarray as xr
from datatree import DataTree
from pandas import date_range
from xarrayutils.plotting import shaded_line_plot
from xmip.postprocessing import concat_members, match_metrics
from xmip.preprocessing import combined_preprocessing
from xmip.utils import google_cmip_col
xr.set_options(keep_attrs=True)
%matplotlib inline
plt.rcParams["figure.figsize"] = (10, 5)
1. Introduccion a los escenarios de Cambio Climático
Los escenarios de cambio climático son una serie de modelos que se han desarrollado con el fin de comprender nuestro clima y las implicaciones futuras de las continuas emisiones de gases de efecto invernadero en la atmósfera. Estos esfuerzos se han concentrado en el Proyecto de Intercomparacion de Modelos (MIP) que invita a entidades de diferentes partes del mundo a realizar simulaciones utilizando modelos bajo escenarios de forzamiento radiativo centralizado (Abernathey, R. 2021). El más reciente Proyecto de Intercomparación de Modelos Acoplados fase 6 (CMIP6) representa un esfuerzo internacional para enfocar el conocimiento acerca de la posible evolucion del sistema climático futuro, y que se encuentra consignado y resumido en el Informe del Panel Intergubernamental sobre el Cambio Climático.
A continuación podemos ver una presentación corta que nos permite entender un poco más que hay detras del Cambio Climático y la modelación climática cortesía de Climate Match Academy.
from IPython.display import IFrame
from ipywidgets import widgets
link_id = "y2bdn"
download_link = f"https://osf.io/download/{link_id}/"
render_link = f"https://mfr.ca-1.osf.io/render?url=https://osf.io/{link_id}/?direct%26mode=render%26action=download%26mode=render"
# @markdown
out = widgets.Output()
with out:
display(IFrame(src=f"{render_link}", width=730, height=410))
display(out)
print("Cortesia: Climate Match Academy (CC BY 4.0)")
Cortesia: Climate Match Academy (CC BY 4.0)
2. Acceso a los datos CMIP6
Los datos de los diferentes modelos se encuentran disponibles en la plataforma en la nube de Google Storage en formato Zarr. Para acceder a los datos de los diferentes escenarios de cambio climático podemos usar la librería xmip. Para crear una conexión con el repositorio de datos de Google usaremos el método google_cmip_col que nos permite acceder a los datos de Pangeo a través de intake de la siguiente manera:
cat = google_cmip_col()
cat
pangeo-cmip6 catalog with 7674 dataset(s) from 514818 asset(s):
| unique | |
|---|---|
| activity_id | 18 |
| institution_id | 36 |
| source_id | 88 |
| experiment_id | 170 |
| member_id | 657 |
| table_id | 37 |
| variable_id | 700 |
| grid_label | 10 |
| zstore | 514818 |
| dcpp_init_year | 60 |
| version | 736 |
| derived_variable_id | 0 |
Como podemos ver hay una gran cantidad de modelaciones de más de 30 instituciones alrededor del mundo. Para efectos pedagógicos, vamos a hacer una consulta de los modelos IPSL, MPI, GFDL, EC, CMCC y CESM2 para el periodo histórico y cada uno de los de los escenarios (SSP - Shared Socioeconomic Pathways) proyectados (ssp126, ssp245, ssp370, ssp585).
Debemos crear un diccionario que nos permita posteriormente filtrar los datos deseados de la siguiente manera:
query = dict(
source_id=[
"IPSL-CM6A-LR",
"MPI-ESM1-2-LR",
# "GFDL-ESM4",
# "EC-Earth3",
"CMCC-ESM2",
# "CESM2",
],
experiment_id=["historical", "ssp126", "ssp370", "ssp245", "ssp585"],
grid_label="gn",
)
El parámetro de etiqueta de reticula grid_label hace referencia a si los datos son reportados en retícula original (gn) o fue reprocesado a una nueva retícula (gr) en formato lat y lon.
Para filtar los datos podemos aplicar el método .search a nuestro catálogo. Le pasamos los filtros previamente definidos incluyendo el identificador de la variable (variable_id), para nuestro caso la temperatura superficial del mar tos. Pasamos también el identificador de miembro member_id que para nuestro caso es r1i1p1f1.
La etiqueta de miembro nos indica lo siguiente:
r = realización
i = inicialización
p = física (parametrización)
f = forzamiento radiativo
Como último parámetro en nuestro ejemplo pasamos el identificador de table table_id que para nuestro caso son datos mensuales del oceano Omon.
cat_filt = cat.search(
**query,
variable_id="tos",
member_id=[
"r1i1p1f1",
], #'r2i1p1f1'
table_id="Omon",
)
cat_filt
pangeo-cmip6 catalog with 15 dataset(s) from 15 asset(s):
| unique | |
|---|---|
| activity_id | 2 |
| institution_id | 3 |
| source_id | 3 |
| experiment_id | 5 |
| member_id | 1 |
| table_id | 1 |
| variable_id | 1 |
| grid_label | 1 |
| zstore | 15 |
| dcpp_init_year | 0 |
| version | 8 |
| derived_variable_id | 0 |
Intake nos permite acceder a los datos de manera rápida y fácil usando Xarray. Para cargar estos datos en un Dataset podemos aplicarle el método .to_dataset_dict que nos permite crear un diccionario con todos los modelos. Podemos pasar un diccionario kwargs con argumentos que nos permiten realizar preprocesamiento de los datos como: renombrar algunos archivos, corregir coordenadas, unidades, entre otros (como podemos ver acá).
kwargs = dict(
preprocess=combined_preprocessing,
xarray_open_kwargs=dict(use_cftime=True),
aggregate=False,
)
ddict = cat_filt.to_dataset_dict(**kwargs)
--> The keys in the returned dictionary of datasets are constructed as follows:
'activity_id.institution_id.source_id.experiment_id.member_id.table_id.variable_id.grid_label.zstore.dcpp_init_year.version'
0.3.0
print(list(ddict.keys())[:2])
['ScenarioMIP.IPSL.IPSL-CM6A-LR.ssp126.r1i1p1f1.Omon.tos.gn.gs://cmip6/CMIP6/ScenarioMIP/IPSL/IPSL-CM6A-LR/ssp126/r1i1p1f1/Omon/tos/gn/v20190903/.20190903', 'ScenarioMIP.MPI-M.MPI-ESM1-2-LR.ssp126.r1i1p1f1.Omon.tos.gn.gs://cmip6/CMIP6/ScenarioMIP/MPI-M/MPI-ESM1-2-LR/ssp126/r1i1p1f1/Omon/tos/gn/v20190710/.20190710']
Revisemos el contenido de uno de estos archivos
ds_test = ddict[
"ScenarioMIP.CMCC.CMCC-ESM2.ssp126.r1i1p1f1.Omon.tos.gn.gs://cmip6/CMIP6/ScenarioMIP/CMCC/CMCC-ESM2/ssp126/r1i1p1f1/Omon/tos/gn/v20210126/.20210126"
]
display(ds_test)
<xarray.Dataset>
Dimensions: (member_id: 1, dcpp_init_year: 1, time: 1032, x: 292,
y: 362, vertex: 4, bnds: 2)
Coordinates:
lat (x, y) float64 dask.array<chunksize=(292, 362), meta=np.ndarray>
lon (x, y) float64 dask.array<chunksize=(292, 362), meta=np.ndarray>
* time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00
lat_verticies (x, y, vertex) float64 dask.array<chunksize=(292, 362, 4), meta=np.ndarray>
lon_verticies (x, y, vertex) float64 dask.array<chunksize=(292, 362, 4), meta=np.ndarray>
time_bounds (time, bnds) object dask.array<chunksize=(1032, 2), meta=np.ndarray>
* x (x) int64 0 1 2 3 4 5 6 7 ... 285 286 287 288 289 290 291
* y (y) int64 0 1 2 3 4 5 6 7 ... 355 356 357 358 359 360 361
lon_bounds (bnds, x, y) float64 dask.array<chunksize=(1, 292, 362), meta=np.ndarray>
lat_bounds (bnds, x, y) float64 dask.array<chunksize=(1, 292, 362), meta=np.ndarray>
* member_id (member_id) object 'r1i1p1f1'
* dcpp_init_year (dcpp_init_year) float64 nan
Dimensions without coordinates: vertex, bnds
Data variables:
tos (member_id, dcpp_init_year, time, x, y) float32 dask.array<chunksize=(1, 1, 253, 292, 362), meta=np.ndarray>
Attributes: (12/64)
Conventions: CF-1.7 CMIP-6.2
activity_id: ScenarioMIP
branch_method: standard
branch_time_in_child: 60225.0
branch_time_in_parent: 60225.0
cmor_version: 3.6.0
... ...
intake_esm_attrs:variable_id: tos
intake_esm_attrs:grid_label: gn
intake_esm_attrs:zstore: gs://cmip6/CMIP6/ScenarioMIP/CMCC/CMCC-...
intake_esm_attrs:version: 20210126
intake_esm_attrs:_data_format_: zarr
intake_esm_dataset_key: ScenarioMIP.CMCC.CMCC-ESM2.ssp126.r1i1p...- member_id: 1
- dcpp_init_year: 1
- time: 1032
- x: 292
- y: 362
- vertex: 4
- bnds: 2
- lat(x, y)float64dask.array<chunksize=(292, 362), meta=np.ndarray>
- bounds :
- vertices_latitude
- long_name :
- latitude
- standard_name :
- latitude
- units :
- degrees_north
Array Chunk Bytes 825.81 kiB 825.81 kiB Shape (292, 362) (292, 362) Dask graph 1 chunks in 5 graph layers Data type float64 numpy.ndarray - lon(x, y)float64dask.array<chunksize=(292, 362), meta=np.ndarray>
- bounds :
- vertices_longitude
- long_name :
- longitude
- standard_name :
- longitude
- units :
- degrees_east
Array Chunk Bytes 825.81 kiB 825.81 kiB Shape (292, 362) (292, 362) Dask graph 1 chunks in 8 graph layers Data type float64 numpy.ndarray - time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bnds
- long_name :
- time
- standard_name :
- time
array([cftime.DatetimeNoLeap(2015, 1, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2015, 2, 15, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2015, 3, 16, 12, 0, 0, 0, has_year_zero=True), ..., cftime.DatetimeNoLeap(2100, 10, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2100, 11, 16, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2100, 12, 16, 12, 0, 0, 0, has_year_zero=True)], dtype=object) - lat_verticies(x, y, vertex)float64dask.array<chunksize=(292, 362, 4), meta=np.ndarray>
- units :
- degrees_north
Array Chunk Bytes 3.23 MiB 3.23 MiB Shape (292, 362, 4) (292, 362, 4) Dask graph 1 chunks in 3 graph layers Data type float64 numpy.ndarray - lon_verticies(x, y, vertex)float64dask.array<chunksize=(292, 362, 4), meta=np.ndarray>
- units :
- degrees_east
Array Chunk Bytes 3.23 MiB 3.23 MiB Shape (292, 362, 4) (292, 362, 4) Dask graph 1 chunks in 6 graph layers Data type float64 numpy.ndarray - time_bounds(time, bnds)objectdask.array<chunksize=(1032, 2), meta=np.ndarray>
Array Chunk Bytes 16.12 kiB 16.12 kiB Shape (1032, 2) (1032, 2) Dask graph 1 chunks in 2 graph layers Data type object numpy.ndarray - x(x)int640 1 2 3 4 5 ... 287 288 289 290 291
array([ 0, 1, 2, ..., 289, 290, 291])
- y(y)int640 1 2 3 4 5 ... 357 358 359 360 361
array([ 0, 1, 2, ..., 359, 360, 361])
- lon_bounds(bnds, x, y)float64dask.array<chunksize=(1, 292, 362), meta=np.ndarray>
- units :
- degrees_east
Array Chunk Bytes 1.61 MiB 825.81 kiB Shape (2, 292, 362) (1, 292, 362) Dask graph 2 chunks in 15 graph layers Data type float64 numpy.ndarray - lat_bounds(bnds, x, y)float64dask.array<chunksize=(1, 292, 362), meta=np.ndarray>
- units :
- degrees_north
Array Chunk Bytes 1.61 MiB 825.81 kiB Shape (2, 292, 362) (1, 292, 362) Dask graph 2 chunks in 12 graph layers Data type float64 numpy.ndarray - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- tos(member_id, dcpp_init_year, time, x, y)float32dask.array<chunksize=(1, 1, 253, 292, 362), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- comment :
- Temperature of upper boundary of the liquid ocean, including temperatures below sea-ice and floating ice shelves.
- long_name :
- Sea Surface Temperature
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 416.13 MiB 102.02 MiB Shape (1, 1, 1032, 292, 362) (1, 1, 253, 292, 362) Dask graph 5 chunks in 3 graph layers Data type float32 numpy.ndarray
- timePandasIndex
PandasIndex(CFTimeIndex([2015-01-16 12:00:00, 2015-02-15 00:00:00, 2015-03-16 12:00:00, 2015-04-16 00:00:00, 2015-05-16 12:00:00, 2015-06-16 00:00:00, 2015-07-16 12:00:00, 2015-08-16 12:00:00, 2015-09-16 00:00:00, 2015-10-16 12:00:00, ... 2100-03-16 12:00:00, 2100-04-16 00:00:00, 2100-05-16 12:00:00, 2100-06-16 00:00:00, 2100-07-16 12:00:00, 2100-08-16 12:00:00, 2100-09-16 00:00:00, 2100-10-16 12:00:00, 2100-11-16 00:00:00, 2100-12-16 12:00:00], dtype='object', length=1032, calendar='noleap', freq='None')) - xPandasIndex
PandasIndex(RangeIndex(start=0, stop=292, step=1, name='x'))
- yPandasIndex
PandasIndex(RangeIndex(start=0, stop=362, step=1, name='y'))
- member_idPandasIndex
PandasIndex(Index(['r1i1p1f1'], dtype='object', name='member_id'))
- dcpp_init_yearPandasIndex
PandasIndex(Index([nan], dtype='float64', name='dcpp_init_year'))
- Conventions :
- CF-1.7 CMIP-6.2
- activity_id :
- ScenarioMIP
- branch_method :
- standard
- branch_time_in_child :
- 60225.0
- branch_time_in_parent :
- 60225.0
- cmor_version :
- 3.6.0
- comment :
- none
- contact :
- T. Lovato
- creation_date :
- 2021-01-25T13:28:44Z
- data_specs_version :
- 01.00.31
- experiment :
- update of RCP2.6 based on SSP1
- experiment_id :
- ssp126
- external_variables :
- areacello
- forcing_index :
- 1
- frequency :
- mon
- further_info_url :
- https://furtherinfo.es-doc.org/CMIP6.CMCC.CMCC-ESM2.ssp126.none.r1i1p1f1
- grid :
- native ocean curvilinear grid
- grid_label :
- gn
- history :
- 2021-01-25T13:28:44Z ;rewrote data to be consistent with ScenarioMIP for variable tos found in table Omon.; none
- initialization_index :
- 1
- institution :
- Fondazione Centro Euro-Mediterraneo sui Cambiamenti Climatici, Lecce 73100, Italy
- institution_id :
- CMCC
- license :
- CMIP6 model data produced by CMCC is licensed under a Creative Commons Attribution ShareAlike 4.0 International License (https://creativecommons.org/licenses). Consult https://pcmdi.llnl.gov/CMIP6/TermsOfUse for terms of use governing CMIP6 output, including citation requirements and proper acknowledgment. Further information about this data, including some limitations, can be found via the further_info_url (recorded as a global attribute in this file) and at https:///pcmdi.llnl.gov/. The data producers and data providers make no warranty, either express or implied, including, but not limited to, warranties of merchantability and fitness for a particular purpose. All liabilities arising from the supply of the information (including any liability arising in negligence) are excluded to the fullest extent permitted by law.
- mip_era :
- CMIP6
- netcdf_tracking_ids :
- hdl:21.14100/b0e80d51-362b-4bcb-8046-d2ed66a64815
- nominal_resolution :
- 100 km
- parent_activity_id :
- CMIP
- parent_experiment_id :
- historical
- parent_mip_era :
- CMIP6
- parent_source_id :
- CMCC-ESM2
- parent_time_units :
- days since 1850-01-01
- parent_variant_label :
- r1i1p1f1
- physics_index :
- 1
- product :
- model-output
- realization_index :
- 1
- realm :
- ocean
- references :
- none
- run_variant :
- 1st realization
- source :
- CMCC-ESM2 (2017): aerosol: MAM3 atmos: CAM5.3 (1deg; 288 x 192 longitude/latitude; 30 levels; top at ~2 hPa) atmosChem: none land: CLM4.5 (BGC mode) landIce: none ocean: NEMO3.6 (ORCA1 tripolar primarly 1 deg lat/lon with meridional refinement down to 1/3 degree in the tropics; 362 x 292 longitude/latitude; 50 vertical levels; top grid cell 0-1 m) ocnBgchem: BFM5.1 seaIce: CICE4.0
- source_id :
- CMCC-ESM2
- source_type :
- AOGCM BGC
- status :
- 2021-04-07;created; by gcs.cmip6.ldeo@gmail.com
- sub_experiment :
- none
- sub_experiment_id :
- none
- table_id :
- Omon
- table_info :
- Creation Date:(05 February 2020) MD5:6a248fd76c55aa6d6f7b3cc6866b5faf
- title :
- CMCC-ESM2 output prepared for CMIP6
- tracking_id :
- hdl:21.14100/b0e80d51-362b-4bcb-8046-d2ed66a64815
- variable_id :
- tos
- variant_label :
- r1i1p1f1
- version_id :
- v20210126
- intake_esm_vars :
- ['tos']
- intake_esm_attrs:activity_id :
- ScenarioMIP
- intake_esm_attrs:institution_id :
- CMCC
- intake_esm_attrs:source_id :
- CMCC-ESM2
- intake_esm_attrs:experiment_id :
- ssp126
- intake_esm_attrs:member_id :
- r1i1p1f1
- intake_esm_attrs:table_id :
- Omon
- intake_esm_attrs:variable_id :
- tos
- intake_esm_attrs:grid_label :
- gn
- intake_esm_attrs:zstore :
- gs://cmip6/CMIP6/ScenarioMIP/CMCC/CMCC-ESM2/ssp126/r1i1p1f1/Omon/tos/gn/v20210126/
- intake_esm_attrs:version :
- 20210126
- intake_esm_attrs:_data_format_ :
- zarr
- intake_esm_dataset_key :
- ScenarioMIP.CMCC.CMCC-ESM2.ssp126.r1i1p1f1.Omon.tos.gn.gs://cmip6/CMIP6/ScenarioMIP/CMCC/CMCC-ESM2/ssp126/r1i1p1f1/Omon/tos/gn/v20210126/.20210126
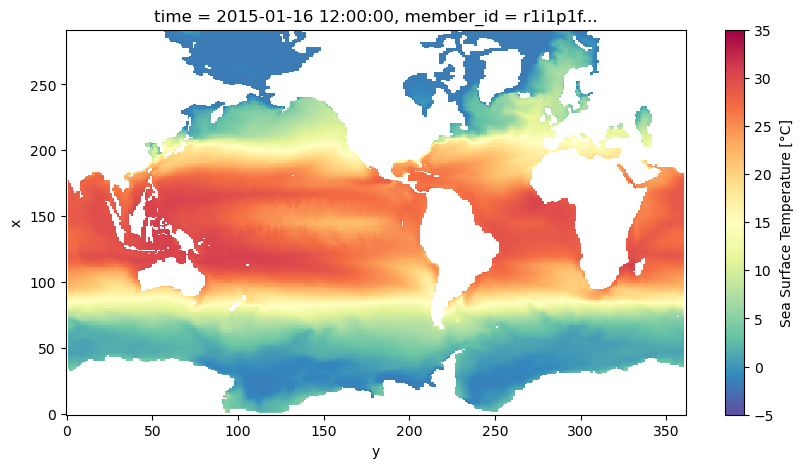
Ahora una inspección gráfica
ds_test.isel(member_id=0, dcpp_init_year=0, time=0).tos.plot(
cmap="Spectral_r", vmin=-5, vmax=35
)
<matplotlib.collections.QuadMesh at 0x7f0508d83950>
3. Temperatura media global ponderada
La temperatura superficial del mar, y cualquier otra variable o salida de los modelos de cambio climático, debe ser ponderada por el área de cada celda.
Créditos: Gael Forget. Para mas información acerca de las simulaciones y las retículas ver https://doi.org/10.5194/gmd-8-3071-2015
Estas áreas ya estan calculadas y disponibles para su consulta de manera similar a los datos de temperatura. Hagamos una consulta al catálogo similar a la anterior cambiando los campos de variable_id=areacello y table_id=Ofx.
cat_area = cat.search(
**query,
table_id="Ofx",
variable_id="areacello",
)
ddict_area = cat_area.to_dataset_dict(**kwargs)
--> The keys in the returned dictionary of datasets are constructed as follows:
'activity_id.institution_id.source_id.experiment_id.member_id.table_id.variable_id.grid_label.zstore.dcpp_init_year.version'
para realizar el cálculo de la temperatura media ponderada por latitud podemos utilizar el módulo match_metrics de la libreria xmip de python de la siguiente manera:
ddict_w_area = match_metrics(ddict, ddict_area, "areacello", print_statistics=True)
Processed 15 datasets.
Exact matches:{'areacello': 0}
Other matches:{'areacello': 15}
No match found:{'areacello': 0}
# ddict_w_area
Ahora procederemos a concatenar los miembros en cada uno de los modelos usando el módulo .concat_members
ddict_trimmed = {k: ds.sel(time=slice(None, "2100")) for k, ds in ddict_w_area.items()}
ddict_combined_members = concat_members(
ddict_w_area,
concat_kwargs={"coords": "minimal", "compat": "override", "join": "override"},
)
Xarray.Dataset no soporta tener múltiples Datasets anidados en un solo objeto de Xarray. Sin embargo, podemos crear un objeto llamado Xarray.datatree que nos permite poner todos nuestros Datasets en un solo objeto de manerar jerárquica. Para entender un poco más los formatos y objeto de tipo jerárquico vea este ejemplo.
# Crear path: diccionario del dataset, donde el path está basado en cada uno de los atributos del dataset
tree_dict = {
f"{ds.source_id}/{ds.experiment_id}/": ds for ds in ddict_combined_members.values()
}
dt = DataTree.from_dict(tree_dict)
display(dt)
<xarray.DatasetView>
Dimensions: ()
Data variables:
*empty*- member_id: 1
- y: 332
- x: 362
- dcpp_init_year: 1
- time: 1032
- vertex: 4
- bnds: 2
- lat(y, x)float32dask.array<chunksize=(332, 362), meta=np.ndarray>
- bounds :
- bounds_nav_lat
- long_name :
- Latitude
- standard_name :
- latitude
- units :
- degrees_north
Array Chunk Bytes 469.47 kiB 469.47 kiB Shape (332, 362) (332, 362) Dask graph 1 chunks in 5 graph layers Data type float32 numpy.ndarray - lon(y, x)float32dask.array<chunksize=(332, 362), meta=np.ndarray>
- bounds :
- bounds_nav_lon
- long_name :
- Longitude
- standard_name :
- longitude
- units :
- degrees_east
Array Chunk Bytes 469.47 kiB 469.47 kiB Shape (332, 362) (332, 362) Dask graph 1 chunks in 8 graph layers Data type float32 numpy.ndarray - time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bounds
- long_name :
- Time axis
- standard_name :
- time
- time_origin :
- 2015-01-01 00:00:00
array([cftime.DatetimeGregorian(2015, 1, 16, 12, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2015, 2, 15, 0, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2015, 3, 16, 12, 0, 0, 0, has_year_zero=False), ..., cftime.DatetimeGregorian(2100, 10, 16, 12, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2100, 11, 16, 0, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2100, 12, 16, 12, 0, 0, 0, has_year_zero=False)], dtype=object) - lat_verticies(y, x, vertex)float32dask.array<chunksize=(332, 362, 4), meta=np.ndarray>
Array Chunk Bytes 1.83 MiB 1.83 MiB Shape (332, 362, 4) (332, 362, 4) Dask graph 1 chunks in 3 graph layers Data type float32 numpy.ndarray - lon_verticies(y, x, vertex)float32dask.array<chunksize=(332, 362, 4), meta=np.ndarray>
Array Chunk Bytes 1.83 MiB 1.83 MiB Shape (332, 362, 4) (332, 362, 4) Dask graph 1 chunks in 6 graph layers Data type float32 numpy.ndarray - time_bounds(time, bnds)objectdask.array<chunksize=(1032, 2), meta=np.ndarray>
Array Chunk Bytes 16.12 kiB 16.12 kiB Shape (1032, 2) (1032, 2) Dask graph 1 chunks in 2 graph layers Data type object numpy.ndarray - y(y)int640 1 2 3 4 5 ... 327 328 329 330 331
array([ 0, 1, 2, ..., 329, 330, 331])
- x(x)int640 1 2 3 4 5 ... 357 358 359 360 361
array([ 0, 1, 2, ..., 359, 360, 361])
- lon_bounds(bnds, y, x)float32dask.array<chunksize=(1, 332, 362), meta=np.ndarray>
Array Chunk Bytes 0.92 MiB 469.47 kiB Shape (2, 332, 362) (1, 332, 362) Dask graph 2 chunks in 15 graph layers Data type float32 numpy.ndarray - lat_bounds(bnds, y, x)float32dask.array<chunksize=(1, 332, 362), meta=np.ndarray>
Array Chunk Bytes 0.92 MiB 469.47 kiB Shape (2, 332, 362) (1, 332, 362) Dask graph 2 chunks in 12 graph layers Data type float32 numpy.ndarray - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- areacello(member_id, dcpp_init_year, y, x)float32dask.array<chunksize=(1, 1, 332, 362), meta=np.ndarray>
- cell_measures :
- area: area
- cell_methods :
- area: sum
- description :
- Cell areas for any grid used to report ocean variables and variables which are requested as used on the model ocean grid (e.g. hfsso, which is a downward heat flux from the atmosphere interpolated onto the ocean grid). These cell areas should be defined to enable exact calculation of global integrals (e.g., of vertical fluxes of energy at the surface and top of the atmosphere).
- history :
- none
- long_name :
- Grid-Cell Area
- online_operation :
- once
- standard_name :
- cell_area
- units :
- m²
- original_key :
- ScenarioMIP.IPSL.IPSL-CM6A-LR.ssp126.r1i1p1f1.Ofx.areacello.gn.gs://cmip6/CMIP6/ScenarioMIP/IPSL/IPSL-CM6A-LR/ssp126/r1i1p1f1/Ofx/areacello/gn/v20190903/.20190903
- parsed_with :
- xmip/postprocessing/_parse_metric
Array Chunk Bytes 469.47 kiB 469.47 kiB Shape (1, 1, 332, 362) (1, 1, 332, 362) Dask graph 1 chunks in 3 graph layers Data type float32 numpy.ndarray
- area(member_id, y, x)float32dask.array<chunksize=(1, 332, 362), meta=np.ndarray>
- standard_name :
- cell_area
- units :
- m²
Array Chunk Bytes 469.47 kiB 469.47 kiB Shape (1, 332, 362) (1, 332, 362) Dask graph 1 chunks in 3 graph layers Data type float32 numpy.ndarray - tos(member_id, dcpp_init_year, time, y, x)float32dask.array<chunksize=(1, 1, 250, 332, 362), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- description :
- This may differ from "surface temperature" in regions of sea ice or floating ice shelves. For models using conservative temperature as the prognostic field, they should report the top ocean layer as surface potential temperature, which is the same as surface in situ temperature.
- history :
- none
- interval_operation :
- 2700 s
- interval_write :
- 1 month
- long_name :
- Sea Surface Temperature
- online_operation :
- average
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 473.14 MiB 114.62 MiB Shape (1, 1, 1032, 332, 362) (1, 1, 250, 332, 362) Dask graph 5 chunks in 3 graph layers Data type float32 numpy.ndarray
- CMIP6_CV_version :
- cv=6.2.3.5-2-g63b123e
- Conventions :
- CF-1.7 CMIP-6.2
- EXPID :
- ssp126
- activity_id :
- ScenarioMIP
- branch_method :
- standard
- branch_time_in_child :
- 0.0
- branch_time_in_parent :
- 60265.0
- contact :
- ipsl-cmip6@listes.ipsl.fr
- creation_date :
- 2018-12-18T21:01:59Z
- data_specs_version :
- 01.00.28
- description :
- Future scenario with low radiative forcing by the end of century. Following approximately RCP2.6 global forcing pathway but with new forcing based on SSP1. Concentration-driven. As a tier 2 option, this simulation should be extended to year 2300
- dr2xml_md5sum :
- c2dce418e78ca835be1e2ff817c2c403
- dr2xml_version :
- 1.16
- experiment :
- update of RCP2.6 based on SSP1
- experiment_id :
- ssp126
- external_variables :
- areacello
- forcing_index :
- 1
- frequency :
- mon
- further_info_url :
- https://furtherinfo.es-doc.org/CMIP6.IPSL.IPSL-CM6A-LR.ssp126.none.r1i1p1f1
- grid :
- native ocean tri-polar grid with 105 k ocean cells
- grid_label :
- gn
- history :
- none
- initialization_index :
- 1
- institution :
- Institut Pierre Simon Laplace, Paris 75252, France
- institution_id :
- IPSL
- license :
- CMIP6 model data produced by IPSL is licensed under a Creative Commons Attribution-NonCommercial-ShareAlike 4.0 International License (https://creativecommons.org/licenses). Consult https://pcmdi.llnl.gov/CMIP6/TermsOfUse for terms of use governing CMIP6 output, including citation requirements and proper acknowledgment. Further information about this data, including some limitations, can be found via the further_info_url (recorded as a global attribute in this file) and at https://cmc.ipsl.fr/. The data producers and data providers make no warranty, either express or implied, including, but not limited to, warranties of merchantability and fitness for a particular purpose. All liabilities arising from the supply of the information (including any liability arising in negligence) are excluded to the fullest extent permitted by law.
- mip_era :
- CMIP6
- model_version :
- 6.1.8
- name :
- /ccc/work/cont003/gencmip6/oboucher/IGCM_OUT/IPSLCM6/PROD/ssp126/CM61-LR-scen-ssp126/CMIP6/OCE/tos_Omon_IPSL-CM6A-LR_ssp126_r1i1p1f1_gn_%start_date%-%end_date%
- nominal_resolution :
- 100 km
- parent_activity_id :
- CMIP
- parent_experiment_id :
- historical
- parent_mip_era :
- CMIP6
- parent_source_id :
- IPSL-CM6A-LR
- parent_time_units :
- days since 1850-01-01 00:00:00
- parent_variant_label :
- r1i1p1f1
- physics_index :
- 1
- product :
- model-output
- realization_index :
- 1
- realm :
- ocean
- source :
- IPSL-CM6A-LR (2017): atmos: LMDZ (NPv6, N96; 144 x 143 longitude/latitude; 79 levels; top level 40000 m) land: ORCHIDEE (v2.0, Water/Carbon/Energy mode) ocean: NEMO-OPA (eORCA1.3, tripolar primarily 1deg; 362 x 332 longitude/latitude; 75 levels; top grid cell 0-2 m) ocnBgchem: NEMO-PISCES seaIce: NEMO-LIM3
- source_id :
- IPSL-CM6A-LR
- source_type :
- AOGCM BGC
- sub_experiment :
- none
- sub_experiment_id :
- none
- table_id :
- Omon
- title :
- IPSL-CM6A-LR model output prepared for CMIP6 / ScenarioMIP ssp126
- tracking_id :
- hdl:21.14100/fc941f96-e04a-4757-bed0-f89f1669a4a1
- variable_id :
- tos
- variant_info :
- Each member starts from the corresponding member of its parent experiment. Information provided by this attribute may in some cases be flawed. Users can find more comprehensive and up-to-date documentation via the further_info_url global attribute.
- variant_label :
- r1i1p1f1
- status :
- 2019-11-09;created;by nhn2@columbia.edu
- netcdf_tracking_ids :
- hdl:21.14100/fc941f96-e04a-4757-bed0-f89f1669a4a1
- version_id :
- v20190903
- intake_esm_vars :
- ['tos']
- intake_esm_attrs:activity_id :
- ScenarioMIP
- intake_esm_attrs:institution_id :
- IPSL
- intake_esm_attrs:source_id :
- IPSL-CM6A-LR
- intake_esm_attrs:experiment_id :
- ssp126
- intake_esm_attrs:member_id :
- r1i1p1f1
- intake_esm_attrs:table_id :
- Omon
- intake_esm_attrs:variable_id :
- tos
- intake_esm_attrs:grid_label :
- gn
- intake_esm_attrs:zstore :
- gs://cmip6/CMIP6/ScenarioMIP/IPSL/IPSL-CM6A-LR/ssp126/r1i1p1f1/Omon/tos/gn/v20190903/
- intake_esm_attrs:version :
- 20190903
- intake_esm_attrs:_data_format_ :
- zarr
- intake_esm_dataset_key :
- ScenarioMIP.IPSL.IPSL-CM6A-LR.ssp126.r1i1p1f1.Omon.tos.gn.gs://cmip6/CMIP6/ScenarioMIP/IPSL/IPSL-CM6A-LR/ssp126/r1i1p1f1/Omon/tos/gn/v20190903/.20190903
<xarray.DatasetView> Dimensions: (member_id: 1, y: 332, x: 362, dcpp_init_year: 1, time: 1032, vertex: 4, bnds: 2) Coordinates: (12/13) lat (y, x) float32 dask.array<chunksize=(332, 362), meta=np.ndarray> lon (y, x) float32 dask.array<chunksize=(332, 362), meta=np.ndarray> * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 lat_verticies (y, x, vertex) float32 dask.array<chunksize=(332, 362, 4), meta=np.ndarray> lon_verticies (y, x, vertex) float32 dask.array<chunksize=(332, 362, 4), meta=np.ndarray> time_bounds (time, bnds) object dask.array<chunksize=(1032, 2), meta=np.ndarray> ... ... * x (x) int64 0 1 2 3 4 5 6 7 ... 355 356 357 358 359 360 361 lon_bounds (bnds, y, x) float32 dask.array<chunksize=(1, 332, 362), meta=np.ndarray> lat_bounds (bnds, y, x) float32 dask.array<chunksize=(1, 332, 362), meta=np.ndarray> * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan areacello (member_id, dcpp_init_year, y, x) float32 dask.array<chunksize=(1, 1, 332, 362), meta=np.ndarray> Dimensions without coordinates: vertex, bnds Data variables: area (member_id, y, x) float32 dask.array<chunksize=(1, 332, 362), meta=np.ndarray> tos (member_id, dcpp_init_year, time, y, x) float32 dask.array<chunksize=(1, 1, 250, 332, 362), meta=np.ndarray> Attributes: (12/67) CMIP6_CV_version: cv=6.2.3.5-2-g63b123e Conventions: CF-1.7 CMIP-6.2 EXPID: ssp126 activity_id: ScenarioMIP branch_method: standard branch_time_in_child: 0.0 ... ... intake_esm_attrs:variable_id: tos intake_esm_attrs:grid_label: gn intake_esm_attrs:zstore: gs://cmip6/CMIP6/ScenarioMIP/IPSL/IPSL-... intake_esm_attrs:version: 20190903 intake_esm_attrs:_data_format_: zarr intake_esm_dataset_key: ScenarioMIP.IPSL.IPSL-CM6A-LR.ssp126.r1...ssp126- member_id: 1
- y: 332
- x: 362
- dcpp_init_year: 1
- time: 1032
- vertex: 4
- bnds: 2
- lat(y, x)float32dask.array<chunksize=(332, 362), meta=np.ndarray>
- bounds :
- bounds_nav_lat
- long_name :
- Latitude
- standard_name :
- latitude
- units :
- degrees_north
Array Chunk Bytes 469.47 kiB 469.47 kiB Shape (332, 362) (332, 362) Dask graph 1 chunks in 5 graph layers Data type float32 numpy.ndarray - lon(y, x)float32dask.array<chunksize=(332, 362), meta=np.ndarray>
- bounds :
- bounds_nav_lon
- long_name :
- Longitude
- standard_name :
- longitude
- units :
- degrees_east
Array Chunk Bytes 469.47 kiB 469.47 kiB Shape (332, 362) (332, 362) Dask graph 1 chunks in 8 graph layers Data type float32 numpy.ndarray - time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bounds
- long_name :
- Time axis
- standard_name :
- time
- time_origin :
- 2015-01-01 00:00:00
array([cftime.DatetimeGregorian(2015, 1, 16, 12, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2015, 2, 15, 0, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2015, 3, 16, 12, 0, 0, 0, has_year_zero=False), ..., cftime.DatetimeGregorian(2100, 10, 16, 12, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2100, 11, 16, 0, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2100, 12, 16, 12, 0, 0, 0, has_year_zero=False)], dtype=object) - lat_verticies(y, x, vertex)float32dask.array<chunksize=(332, 362, 4), meta=np.ndarray>
Array Chunk Bytes 1.83 MiB 1.83 MiB Shape (332, 362, 4) (332, 362, 4) Dask graph 1 chunks in 3 graph layers Data type float32 numpy.ndarray - lon_verticies(y, x, vertex)float32dask.array<chunksize=(332, 362, 4), meta=np.ndarray>
Array Chunk Bytes 1.83 MiB 1.83 MiB Shape (332, 362, 4) (332, 362, 4) Dask graph 1 chunks in 6 graph layers Data type float32 numpy.ndarray - time_bounds(time, bnds)objectdask.array<chunksize=(1032, 2), meta=np.ndarray>
Array Chunk Bytes 16.12 kiB 16.12 kiB Shape (1032, 2) (1032, 2) Dask graph 1 chunks in 2 graph layers Data type object numpy.ndarray - y(y)int640 1 2 3 4 5 ... 327 328 329 330 331
array([ 0, 1, 2, ..., 329, 330, 331])
- x(x)int640 1 2 3 4 5 ... 357 358 359 360 361
array([ 0, 1, 2, ..., 359, 360, 361])
- lon_bounds(bnds, y, x)float32dask.array<chunksize=(1, 332, 362), meta=np.ndarray>
Array Chunk Bytes 0.92 MiB 469.47 kiB Shape (2, 332, 362) (1, 332, 362) Dask graph 2 chunks in 15 graph layers Data type float32 numpy.ndarray - lat_bounds(bnds, y, x)float32dask.array<chunksize=(1, 332, 362), meta=np.ndarray>
Array Chunk Bytes 0.92 MiB 469.47 kiB Shape (2, 332, 362) (1, 332, 362) Dask graph 2 chunks in 12 graph layers Data type float32 numpy.ndarray - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- areacello(member_id, dcpp_init_year, y, x)float32dask.array<chunksize=(1, 1, 332, 362), meta=np.ndarray>
- cell_measures :
- area: area
- cell_methods :
- area: sum
- description :
- Cell areas for any grid used to report ocean variables and variables which are requested as used on the model ocean grid (e.g. hfsso, which is a downward heat flux from the atmosphere interpolated onto the ocean grid). These cell areas should be defined to enable exact calculation of global integrals (e.g., of vertical fluxes of energy at the surface and top of the atmosphere).
- history :
- none
- long_name :
- Grid-Cell Area
- online_operation :
- once
- standard_name :
- cell_area
- units :
- m²
- original_key :
- ScenarioMIP.IPSL.IPSL-CM6A-LR.ssp585.r1i1p1f1.Ofx.areacello.gn.gs://cmip6/CMIP6/ScenarioMIP/IPSL/IPSL-CM6A-LR/ssp585/r1i1p1f1/Ofx/areacello/gn/v20190903/.20190903
- parsed_with :
- xmip/postprocessing/_parse_metric
Array Chunk Bytes 469.47 kiB 469.47 kiB Shape (1, 1, 332, 362) (1, 1, 332, 362) Dask graph 1 chunks in 3 graph layers Data type float32 numpy.ndarray
- area(member_id, y, x)float32dask.array<chunksize=(1, 332, 362), meta=np.ndarray>
- standard_name :
- cell_area
- units :
- m²
Array Chunk Bytes 469.47 kiB 469.47 kiB Shape (1, 332, 362) (1, 332, 362) Dask graph 1 chunks in 3 graph layers Data type float32 numpy.ndarray - tos(member_id, dcpp_init_year, time, y, x)float32dask.array<chunksize=(1, 1, 120, 332, 362), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- description :
- This may differ from "surface temperature" in regions of sea ice or floating ice shelves. For models using conservative temperature as the prognostic field, they should report the top ocean layer as surface potential temperature, which is the same as surface in situ temperature.
- history :
- none
- interval_operation :
- 2700 s
- interval_write :
- 1 month
- long_name :
- Sea Surface Temperature
- online_operation :
- average
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 473.14 MiB 55.02 MiB Shape (1, 1, 1032, 332, 362) (1, 1, 120, 332, 362) Dask graph 9 chunks in 3 graph layers Data type float32 numpy.ndarray
- CMIP6_CV_version :
- cv=6.2.3.5-2-g63b123e
- Conventions :
- CF-1.7 CMIP-6.2
- EXPID :
- ssp585
- activity_id :
- ScenarioMIP
- branch_method :
- standard
- branch_time_in_child :
- 0.0
- branch_time_in_parent :
- 60265.0
- contact :
- ipsl-cmip6@listes.ipsl.fr
- creation_date :
- 2018-12-18T21:02:33Z
- data_specs_version :
- 01.00.28
- description :
- Future scenario with high radiative forcing by the end of century. Following approximately RCP8.5 global forcing pathway but with new forcing based on SSP5. Concentration-driven. As a tier 2 option, this simulation should be extended to year 2300
- dr2xml_md5sum :
- c2dce418e78ca835be1e2ff817c2c403
- dr2xml_version :
- 1.16
- experiment :
- update of RCP8.5 based on SSP5
- experiment_id :
- ssp585
- external_variables :
- areacello
- forcing_index :
- 1
- frequency :
- mon
- further_info_url :
- https://furtherinfo.es-doc.org/CMIP6.IPSL.IPSL-CM6A-LR.ssp585.none.r1i1p1f1
- grid :
- native ocean tri-polar grid with 105 k ocean cells
- grid_label :
- gn
- history :
- none
- initialization_index :
- 1
- institution :
- Institut Pierre Simon Laplace, Paris 75252, France
- institution_id :
- IPSL
- license :
- CMIP6 model data produced by IPSL is licensed under a Creative Commons Attribution-NonCommercial-ShareAlike 4.0 International License (https://creativecommons.org/licenses). Consult https://pcmdi.llnl.gov/CMIP6/TermsOfUse for terms of use governing CMIP6 output, including citation requirements and proper acknowledgment. Further information about this data, including some limitations, can be found via the further_info_url (recorded as a global attribute in this file) and at https://cmc.ipsl.fr/. The data producers and data providers make no warranty, either express or implied, including, but not limited to, warranties of merchantability and fitness for a particular purpose. All liabilities arising from the supply of the information (including any liability arising in negligence) are excluded to the fullest extent permitted by law.
- mip_era :
- CMIP6
- model_version :
- 6.1.8
- name :
- /ccc/work/cont003/gencmip6/oboucher/IGCM_OUT/IPSLCM6/PROD/ssp585/CM61-LR-scen-ssp585/CMIP6/OCE/tos_Omon_IPSL-CM6A-LR_ssp585_r1i1p1f1_gn_%start_date%-%end_date%
- nominal_resolution :
- 100 km
- parent_activity_id :
- CMIP
- parent_experiment_id :
- historical
- parent_mip_era :
- CMIP6
- parent_source_id :
- IPSL-CM6A-LR
- parent_time_units :
- days since 1850-01-01 00:00:00
- parent_variant_label :
- r1i1p1f1
- physics_index :
- 1
- product :
- model-output
- realization_index :
- 1
- realm :
- ocean
- source :
- IPSL-CM6A-LR (2017): atmos: LMDZ (NPv6, N96; 144 x 143 longitude/latitude; 79 levels; top level 40000 m) land: ORCHIDEE (v2.0, Water/Carbon/Energy mode) ocean: NEMO-OPA (eORCA1.3, tripolar primarily 1deg; 362 x 332 longitude/latitude; 75 levels; top grid cell 0-2 m) ocnBgchem: NEMO-PISCES seaIce: NEMO-LIM3
- source_id :
- IPSL-CM6A-LR
- source_type :
- AOGCM BGC
- sub_experiment :
- none
- sub_experiment_id :
- none
- table_id :
- Omon
- title :
- IPSL-CM6A-LR model output prepared for CMIP6 / ScenarioMIP ssp585
- tracking_id :
- hdl:21.14100/eb01c677-634a-4e5c-8e0f-7984423c7136
- variable_id :
- tos
- variant_info :
- Each member starts from the corresponding member of its parent experiment. Information provided by this attribute may in some cases be flawed. Users can find more comprehensive and up-to-date documentation via the further_info_url global attribute.
- variant_label :
- r1i1p1f1
- status :
- 2019-10-25;created;by nhn2@columbia.edu
- netcdf_tracking_ids :
- hdl:21.14100/eb01c677-634a-4e5c-8e0f-7984423c7136
- version_id :
- v20190903
- intake_esm_vars :
- ['tos']
- intake_esm_attrs:activity_id :
- ScenarioMIP
- intake_esm_attrs:institution_id :
- IPSL
- intake_esm_attrs:source_id :
- IPSL-CM6A-LR
- intake_esm_attrs:experiment_id :
- ssp585
- intake_esm_attrs:member_id :
- r1i1p1f1
- intake_esm_attrs:table_id :
- Omon
- intake_esm_attrs:variable_id :
- tos
- intake_esm_attrs:grid_label :
- gn
- intake_esm_attrs:zstore :
- gs://cmip6/CMIP6/ScenarioMIP/IPSL/IPSL-CM6A-LR/ssp585/r1i1p1f1/Omon/tos/gn/v20190903/
- intake_esm_attrs:version :
- 20190903
- intake_esm_attrs:_data_format_ :
- zarr
- intake_esm_dataset_key :
- ScenarioMIP.IPSL.IPSL-CM6A-LR.ssp585.r1i1p1f1.Omon.tos.gn.gs://cmip6/CMIP6/ScenarioMIP/IPSL/IPSL-CM6A-LR/ssp585/r1i1p1f1/Omon/tos/gn/v20190903/.20190903
<xarray.DatasetView> Dimensions: (member_id: 1, y: 332, x: 362, dcpp_init_year: 1, time: 1032, vertex: 4, bnds: 2) Coordinates: (12/13) lat (y, x) float32 dask.array<chunksize=(332, 362), meta=np.ndarray> lon (y, x) float32 dask.array<chunksize=(332, 362), meta=np.ndarray> * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 lat_verticies (y, x, vertex) float32 dask.array<chunksize=(332, 362, 4), meta=np.ndarray> lon_verticies (y, x, vertex) float32 dask.array<chunksize=(332, 362, 4), meta=np.ndarray> time_bounds (time, bnds) object dask.array<chunksize=(1032, 2), meta=np.ndarray> ... ... * x (x) int64 0 1 2 3 4 5 6 7 ... 355 356 357 358 359 360 361 lon_bounds (bnds, y, x) float32 dask.array<chunksize=(1, 332, 362), meta=np.ndarray> lat_bounds (bnds, y, x) float32 dask.array<chunksize=(1, 332, 362), meta=np.ndarray> * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan areacello (member_id, dcpp_init_year, y, x) float32 dask.array<chunksize=(1, 1, 332, 362), meta=np.ndarray> Dimensions without coordinates: vertex, bnds Data variables: area (member_id, y, x) float32 dask.array<chunksize=(1, 332, 362), meta=np.ndarray> tos (member_id, dcpp_init_year, time, y, x) float32 dask.array<chunksize=(1, 1, 120, 332, 362), meta=np.ndarray> Attributes: (12/67) CMIP6_CV_version: cv=6.2.3.5-2-g63b123e Conventions: CF-1.7 CMIP-6.2 EXPID: ssp585 activity_id: ScenarioMIP branch_method: standard branch_time_in_child: 0.0 ... ... intake_esm_attrs:variable_id: tos intake_esm_attrs:grid_label: gn intake_esm_attrs:zstore: gs://cmip6/CMIP6/ScenarioMIP/IPSL/IPSL-... intake_esm_attrs:version: 20190903 intake_esm_attrs:_data_format_: zarr intake_esm_dataset_key: ScenarioMIP.IPSL.IPSL-CM6A-LR.ssp585.r1...ssp585- member_id: 1
- y: 332
- x: 362
- dcpp_init_year: 1
- time: 1980
- vertex: 4
- bnds: 2
- lat(y, x)float32dask.array<chunksize=(332, 362), meta=np.ndarray>
- bounds :
- bounds_nav_lat
- long_name :
- Latitude
- standard_name :
- latitude
- units :
- degrees_north
Array Chunk Bytes 469.47 kiB 469.47 kiB Shape (332, 362) (332, 362) Dask graph 1 chunks in 5 graph layers Data type float32 numpy.ndarray - lon(y, x)float32dask.array<chunksize=(332, 362), meta=np.ndarray>
- bounds :
- bounds_nav_lon
- long_name :
- Longitude
- standard_name :
- longitude
- units :
- degrees_east
Array Chunk Bytes 469.47 kiB 469.47 kiB Shape (332, 362) (332, 362) Dask graph 1 chunks in 8 graph layers Data type float32 numpy.ndarray - time(time)object1850-01-16 12:00:00 ... 2014-12-...
- axis :
- T
- bounds :
- time_bounds
- long_name :
- Time axis
- standard_name :
- time
- time_origin :
- 1850-01-01 00:00:00
array([cftime.DatetimeGregorian(1850, 1, 16, 12, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(1850, 2, 15, 0, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(1850, 3, 16, 12, 0, 0, 0, has_year_zero=False), ..., cftime.DatetimeGregorian(2014, 10, 16, 12, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2014, 11, 16, 0, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2014, 12, 16, 12, 0, 0, 0, has_year_zero=False)], dtype=object) - lat_verticies(y, x, vertex)float32dask.array<chunksize=(332, 362, 4), meta=np.ndarray>
Array Chunk Bytes 1.83 MiB 1.83 MiB Shape (332, 362, 4) (332, 362, 4) Dask graph 1 chunks in 3 graph layers Data type float32 numpy.ndarray - lon_verticies(y, x, vertex)float32dask.array<chunksize=(332, 362, 4), meta=np.ndarray>
Array Chunk Bytes 1.83 MiB 1.83 MiB Shape (332, 362, 4) (332, 362, 4) Dask graph 1 chunks in 6 graph layers Data type float32 numpy.ndarray - time_bounds(time, bnds)objectdask.array<chunksize=(1980, 2), meta=np.ndarray>
Array Chunk Bytes 30.94 kiB 30.94 kiB Shape (1980, 2) (1980, 2) Dask graph 1 chunks in 2 graph layers Data type object numpy.ndarray - y(y)int640 1 2 3 4 5 ... 327 328 329 330 331
array([ 0, 1, 2, ..., 329, 330, 331])
- x(x)int640 1 2 3 4 5 ... 357 358 359 360 361
array([ 0, 1, 2, ..., 359, 360, 361])
- lon_bounds(bnds, y, x)float32dask.array<chunksize=(1, 332, 362), meta=np.ndarray>
Array Chunk Bytes 0.92 MiB 469.47 kiB Shape (2, 332, 362) (1, 332, 362) Dask graph 2 chunks in 15 graph layers Data type float32 numpy.ndarray - lat_bounds(bnds, y, x)float32dask.array<chunksize=(1, 332, 362), meta=np.ndarray>
Array Chunk Bytes 0.92 MiB 469.47 kiB Shape (2, 332, 362) (1, 332, 362) Dask graph 2 chunks in 12 graph layers Data type float32 numpy.ndarray - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- areacello(member_id, dcpp_init_year, y, x)float32dask.array<chunksize=(1, 1, 332, 362), meta=np.ndarray>
- cell_measures :
- area: area
- cell_methods :
- area: sum
- description :
- Cell areas for any grid used to report ocean variables and variables which are requested as used on the model ocean grid (e.g. hfsso, which is a downward heat flux from the atmosphere interpolated onto the ocean grid). These cell areas should be defined to enable exact calculation of global integrals (e.g., of vertical fluxes of energy at the surface and top of the atmosphere).
- history :
- none
- long_name :
- Grid-Cell Area
- online_operation :
- once
- standard_name :
- cell_area
- units :
- m²
- original_key :
- CMIP.IPSL.IPSL-CM6A-LR.historical.r1i1p1f1.Ofx.areacello.gn.gs://cmip6/CMIP6/CMIP/IPSL/IPSL-CM6A-LR/historical/r1i1p1f1/Ofx/areacello/gn/v20180803/.20180803
- parsed_with :
- xmip/postprocessing/_parse_metric
Array Chunk Bytes 469.47 kiB 469.47 kiB Shape (1, 1, 332, 362) (1, 1, 332, 362) Dask graph 1 chunks in 3 graph layers Data type float32 numpy.ndarray
- area(member_id, y, x)float32dask.array<chunksize=(1, 332, 362), meta=np.ndarray>
- standard_name :
- cell_area
- units :
- m²
Array Chunk Bytes 469.47 kiB 469.47 kiB Shape (1, 332, 362) (1, 332, 362) Dask graph 1 chunks in 3 graph layers Data type float32 numpy.ndarray - tos(member_id, dcpp_init_year, time, y, x)float32dask.array<chunksize=(1, 1, 252, 332, 362), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- description :
- This may differ from "surface temperature" in regions of sea ice or floating ice shelves. For models using conservative temperature as the prognostic field, they should report the top ocean layer as surface potential temperature, which is the same as surface in situ temperature.
- history :
- none
- interval_operation :
- 2700 s
- interval_write :
- 1 month
- long_name :
- Sea Surface Temperature
- online_operation :
- average
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 907.76 MiB 115.53 MiB Shape (1, 1, 1980, 332, 362) (1, 1, 252, 332, 362) Dask graph 8 chunks in 3 graph layers Data type float32 numpy.ndarray
- CMIP6_CV_version :
- cv=6.2.3.5-2-g63b123e
- Conventions :
- CF-1.7 CMIP-6.2
- EXPID :
- historical
- NCO :
- "4.6.0"
- activity_id :
- CMIP
- branch_method :
- standard
- branch_time_in_child :
- 0.0
- branch_time_in_parent :
- 21914.0
- contact :
- ipsl-cmip6@listes.ipsl.fr
- creation_date :
- 2018-07-11T07:36:14Z
- data_specs_version :
- 01.00.21
- description :
- CMIP6 historical
- dr2xml_md5sum :
- f1e40c1fc5d8281f865f72fbf4e38f9d
- dr2xml_version :
- 1.11
- experiment :
- all-forcing simulation of the recent past
- experiment_id :
- historical
- external_variables :
- areacello
- forcing_index :
- 1
- frequency :
- mon
- further_info_url :
- https://furtherinfo.es-doc.org/CMIP6.IPSL.IPSL-CM6A-LR.historical.none.r1i1p1f1
- grid :
- native ocean tri-polar grid with 105 k ocean cells
- grid_label :
- gn
- history :
- Sat Dec 1 12:15:54 2018: ncatted -O -a realization_index,global,m,i,1 /ccc/work/cont003/cmip6/cmip6/onhold/CM61-LR-histEXT-03.1910/files+ext/tos_Omon_IPSL-CM6A-LR_historical_r1i1p1f1_gn_185001-201412.nc Sat Dec 1 12:09:05 2018: ncatted -O -a realization_index,global,m,i,1 /ccc/work/cont003/cmip6/cmip6/onhold/CM61-LR-hist-03.1910/files/tos_Omon_IPSL-CM6A-LR_historical_r1i1p1f1_gn_185001-201412.nc Sat Dec 1 10:58:36 2018: ncatted -O -a realization_index,global,m,i,1 /ccc/work/cont003/gencmip6/p86caub/IGCM_OUT/IPSLCM6/PROD/historical/CM61-LR-hist-03.1910/CMIP6/OCE/tos_Omon_IPSL-CM6A-LR_historical_r1i1p1f1_gn_185001-201412.nc Fri Nov 30 16:47:56 2018: ncatted -O -a realization_index,global,m,s,1 /ccc/work/cont003/gencmip6/p86caub/IGCM_OUT/IPSLCM6/PROD/historical/CM61-LR-hist-03.1910/CMIP6/OCE/tos_Omon_IPSL-CM6A-LR_historical_r1i1p1f1_gn_185001-201412.nc Thu Nov 29 16:47:45 2018: ncatted -O -a variant_label,global,m,c,r1i1p1f1 /ccc/work/cont003/gencmip6/p86caub/IGCM_OUT/IPSLCM6/PROD/historical/CM61-LR-hist-03.1910/CMIP6/OCE/tos_Omon_IPSL-CM6A-LR_historical_r3i1p1f1_gn_185001-201412.nc Thu Nov 29 16:47:45 2018: ncatted -O -a further_info_url,global,m,c,https://furtherinfo.es-doc.org/CMIP6.IPSL.IPSL-CM6A-LR.historical.none.r1i1p1f1 /ccc/work/cont003/gencmip6/p86caub/IGCM_OUT/IPSLCM6/PROD/historical/CM61-LR-hist-03.1910/CMIP6/OCE/tos_Omon_IPSL-CM6A-LR_historical_r3i1p1f1_gn_185001-201412.nc Thu Nov 29 16:47:45 2018: ncatted -O -a name,global,m,c,/ccc/work/cont003/gencmip6/p86caub/IGCM_OUT/IPSLCM6/PROD/historical/CM61-LR-hist-03.1910/CMIP6/OCE/tos_Omon_IPSL-CM6A-LR_historical_r1i1p1f1_gn_%start_date%-%end_date% /ccc/work/cont003/gencmip6/p86caub/IGCM_OUT/IPSLCM6/PROD/historical/CM61-LR-hist-03.1910/CMIP6/OCE/tos_Omon_IPSL-CM6A-LR_historical_r3i1p1f1_gn_185001-201412.nc Mon Sep 3 14:53:26 2018: ncatted -O -a parent_variant_label,global,m,c,r1i1p1f1 tos_Omon_IPSL-CM6A-LR_historical_r3i1p1f1_gn_185001-201412.nc Mon Aug 6 17:58:17 2018: ncatted -O -a coordinates,area,o,c,nav_lon nav_lat /ccc/work/cont003/gencmip6/p86caub/IGCM_OUT/IPSLCM6/PROD/historical/CM61-LR-hist-03.1910/CMIP6/OCE/tos_Omon_IPSL-CM6A-LR_historical_r3i1p1f1_gn_185001-201412.nc none
- initialization_index :
- 1
- institution :
- Institut Pierre Simon Laplace, Paris 75252, France
- institution_id :
- IPSL
- license :
- CMIP6 model data produced by IPSL is licensed under a Creative Commons Attribution-NonCommercial-ShareAlike 4.0 International License (https://creativecommons.org/licenses). Consult https://pcmdi.llnl.gov/CMIP6/TermsOfUse for terms of use governing CMIP6 output, including citation requirements and proper acknowledgment. Further information about this data, including some limitations, can be found via the further_info_url (recorded as a global attribute in this file) and at https://cmc.ipsl.fr/. The data producers and data providers make no warranty, either express or implied, including, but not limited to, warranties of merchantability and fitness for a particular purpose. All liabilities arising from the supply of the information (including any liability arising in negligence) are excluded to the fullest extent permitted by law.
- mip_era :
- CMIP6
- model_version :
- 6.1.5
- name :
- /ccc/work/cont003/gencmip6/p86caub/IGCM_OUT/IPSLCM6/PROD/historical/CM61-LR-hist-03.1910/CMIP6/OCE/tos_Omon_IPSL-CM6A-LR_historical_r1i1p1f1_gn_%start_date%-%end_date%
- nominal_resolution :
- 100 km
- parent_activity_id :
- CMIP
- parent_experiment_id :
- piControl
- parent_mip_era :
- CMIP6
- parent_source_id :
- IPSL-CM6A-LR
- parent_time_units :
- days since 1850-01-01 00:00:00
- parent_variant_label :
- r1i1p1f1
- physics_index :
- 1
- product :
- model-output
- realization_index :
- 1
- realm :
- ocean
- source :
- IPSL-CM6A-LR (2017): atmos: LMDZ (NPv6, N96; 144 x 143 longitude/latitude; 79 levels; top level 40000 m) land: ORCHIDEE (v2.0, Water/Carbon/Energy mode) ocean: NEMO-OPA (eORCA1.3, tripolar primarily 1deg; 362 x 332 longitude/latitude; 75 levels; top grid cell 0-2 m) ocnBgchem: NEMO-PISCES seaIce: NEMO-LIM3
- source_id :
- IPSL-CM6A-LR
- source_type :
- AOGCM BGC
- sub_experiment :
- none
- sub_experiment_id :
- none
- table_id :
- Omon
- title :
- IPSL-CM6A-LR model output prepared for CMIP6 / CMIP historical
- tracking_id :
- hdl:21.14100/01f4d96a-9054-4974-b8dd-4f91e73989d2
- variable_id :
- tos
- variant_label :
- r1i1p1f1
- status :
- 2019-11-10;created;by nhn2@columbia.edu
- netcdf_tracking_ids :
- hdl:21.14100/01f4d96a-9054-4974-b8dd-4f91e73989d2
- version_id :
- v20180803
- intake_esm_vars :
- ['tos']
- intake_esm_attrs:activity_id :
- CMIP
- intake_esm_attrs:institution_id :
- IPSL
- intake_esm_attrs:source_id :
- IPSL-CM6A-LR
- intake_esm_attrs:experiment_id :
- historical
- intake_esm_attrs:member_id :
- r1i1p1f1
- intake_esm_attrs:table_id :
- Omon
- intake_esm_attrs:variable_id :
- tos
- intake_esm_attrs:grid_label :
- gn
- intake_esm_attrs:zstore :
- gs://cmip6/CMIP6/CMIP/IPSL/IPSL-CM6A-LR/historical/r1i1p1f1/Omon/tos/gn/v20180803/
- intake_esm_attrs:version :
- 20180803
- intake_esm_attrs:_data_format_ :
- zarr
- intake_esm_dataset_key :
- CMIP.IPSL.IPSL-CM6A-LR.historical.r1i1p1f1.Omon.tos.gn.gs://cmip6/CMIP6/CMIP/IPSL/IPSL-CM6A-LR/historical/r1i1p1f1/Omon/tos/gn/v20180803/.20180803
<xarray.DatasetView> Dimensions: (member_id: 1, y: 332, x: 362, dcpp_init_year: 1, time: 1980, vertex: 4, bnds: 2) Coordinates: (12/13) lat (y, x) float32 dask.array<chunksize=(332, 362), meta=np.ndarray> lon (y, x) float32 dask.array<chunksize=(332, 362), meta=np.ndarray> * time (time) object 1850-01-16 12:00:00 ... 2014-12-16 12:00:00 lat_verticies (y, x, vertex) float32 dask.array<chunksize=(332, 362, 4), meta=np.ndarray> lon_verticies (y, x, vertex) float32 dask.array<chunksize=(332, 362, 4), meta=np.ndarray> time_bounds (time, bnds) object dask.array<chunksize=(1980, 2), meta=np.ndarray> ... ... * x (x) int64 0 1 2 3 4 5 6 7 ... 355 356 357 358 359 360 361 lon_bounds (bnds, y, x) float32 dask.array<chunksize=(1, 332, 362), meta=np.ndarray> lat_bounds (bnds, y, x) float32 dask.array<chunksize=(1, 332, 362), meta=np.ndarray> * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan areacello (member_id, dcpp_init_year, y, x) float32 dask.array<chunksize=(1, 1, 332, 362), meta=np.ndarray> Dimensions without coordinates: vertex, bnds Data variables: area (member_id, y, x) float32 dask.array<chunksize=(1, 332, 362), meta=np.ndarray> tos (member_id, dcpp_init_year, time, y, x) float32 dask.array<chunksize=(1, 1, 252, 332, 362), meta=np.ndarray> Attributes: (12/67) CMIP6_CV_version: cv=6.2.3.5-2-g63b123e Conventions: CF-1.7 CMIP-6.2 EXPID: historical NCO: "4.6.0" activity_id: CMIP branch_method: standard ... ... intake_esm_attrs:variable_id: tos intake_esm_attrs:grid_label: gn intake_esm_attrs:zstore: gs://cmip6/CMIP6/CMIP/IPSL/IPSL-CM6A-LR... intake_esm_attrs:version: 20180803 intake_esm_attrs:_data_format_: zarr intake_esm_dataset_key: CMIP.IPSL.IPSL-CM6A-LR.historical.r1i1p...historical- member_id: 1
- y: 332
- x: 362
- dcpp_init_year: 1
- time: 1032
- vertex: 4
- bnds: 2
- lat(y, x)float32dask.array<chunksize=(332, 362), meta=np.ndarray>
- bounds :
- bounds_nav_lat
- long_name :
- Latitude
- standard_name :
- latitude
- units :
- degrees_north
Array Chunk Bytes 469.47 kiB 469.47 kiB Shape (332, 362) (332, 362) Dask graph 1 chunks in 5 graph layers Data type float32 numpy.ndarray - lon(y, x)float32dask.array<chunksize=(332, 362), meta=np.ndarray>
- bounds :
- bounds_nav_lon
- long_name :
- Longitude
- standard_name :
- longitude
- units :
- degrees_east
Array Chunk Bytes 469.47 kiB 469.47 kiB Shape (332, 362) (332, 362) Dask graph 1 chunks in 8 graph layers Data type float32 numpy.ndarray - time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bounds
- long_name :
- Time axis
- standard_name :
- time
- time_origin :
- 2015-01-01 00:00:00
array([cftime.DatetimeGregorian(2015, 1, 16, 12, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2015, 2, 15, 0, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2015, 3, 16, 12, 0, 0, 0, has_year_zero=False), ..., cftime.DatetimeGregorian(2100, 10, 16, 12, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2100, 11, 16, 0, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2100, 12, 16, 12, 0, 0, 0, has_year_zero=False)], dtype=object) - lat_verticies(y, x, vertex)float32dask.array<chunksize=(332, 362, 4), meta=np.ndarray>
Array Chunk Bytes 1.83 MiB 1.83 MiB Shape (332, 362, 4) (332, 362, 4) Dask graph 1 chunks in 3 graph layers Data type float32 numpy.ndarray - lon_verticies(y, x, vertex)float32dask.array<chunksize=(332, 362, 4), meta=np.ndarray>
Array Chunk Bytes 1.83 MiB 1.83 MiB Shape (332, 362, 4) (332, 362, 4) Dask graph 1 chunks in 6 graph layers Data type float32 numpy.ndarray - time_bounds(time, bnds)objectdask.array<chunksize=(1032, 2), meta=np.ndarray>
Array Chunk Bytes 16.12 kiB 16.12 kiB Shape (1032, 2) (1032, 2) Dask graph 1 chunks in 2 graph layers Data type object numpy.ndarray - y(y)int640 1 2 3 4 5 ... 327 328 329 330 331
array([ 0, 1, 2, ..., 329, 330, 331])
- x(x)int640 1 2 3 4 5 ... 357 358 359 360 361
array([ 0, 1, 2, ..., 359, 360, 361])
- lon_bounds(bnds, y, x)float32dask.array<chunksize=(1, 332, 362), meta=np.ndarray>
Array Chunk Bytes 0.92 MiB 469.47 kiB Shape (2, 332, 362) (1, 332, 362) Dask graph 2 chunks in 15 graph layers Data type float32 numpy.ndarray - lat_bounds(bnds, y, x)float32dask.array<chunksize=(1, 332, 362), meta=np.ndarray>
Array Chunk Bytes 0.92 MiB 469.47 kiB Shape (2, 332, 362) (1, 332, 362) Dask graph 2 chunks in 12 graph layers Data type float32 numpy.ndarray - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- areacello(member_id, dcpp_init_year, y, x)float32dask.array<chunksize=(1, 1, 332, 362), meta=np.ndarray>
- cell_measures :
- area: area
- cell_methods :
- area: sum
- description :
- Cell areas for any grid used to report ocean variables and variables which are requested as used on the model ocean grid (e.g. hfsso, which is a downward heat flux from the atmosphere interpolated onto the ocean grid). These cell areas should be defined to enable exact calculation of global integrals (e.g., of vertical fluxes of energy at the surface and top of the atmosphere).
- history :
- none
- long_name :
- Grid-Cell Area
- online_operation :
- once
- standard_name :
- cell_area
- units :
- m²
- original_key :
- ScenarioMIP.IPSL.IPSL-CM6A-LR.ssp370.r1i1p1f1.Ofx.areacello.gn.gs://cmip6/CMIP6/ScenarioMIP/IPSL/IPSL-CM6A-LR/ssp370/r1i1p1f1/Ofx/areacello/gn/v20190119/.20190119
- parsed_with :
- xmip/postprocessing/_parse_metric
Array Chunk Bytes 469.47 kiB 469.47 kiB Shape (1, 1, 332, 362) (1, 1, 332, 362) Dask graph 1 chunks in 3 graph layers Data type float32 numpy.ndarray
- area(member_id, y, x)float32dask.array<chunksize=(1, 332, 362), meta=np.ndarray>
- standard_name :
- cell_area
- units :
- m²
Array Chunk Bytes 469.47 kiB 469.47 kiB Shape (1, 332, 362) (1, 332, 362) Dask graph 1 chunks in 3 graph layers Data type float32 numpy.ndarray - tos(member_id, dcpp_init_year, time, y, x)float32dask.array<chunksize=(1, 1, 250, 332, 362), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- description :
- This may differ from "surface temperature" in regions of sea ice or floating ice shelves. For models using conservative temperature as the prognostic field, they should report the top ocean layer as surface potential temperature, which is the same as surface in situ temperature.
- history :
- none
- interval_operation :
- 2700 s
- interval_write :
- 1 month
- long_name :
- Sea Surface Temperature
- online_operation :
- average
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 473.14 MiB 114.62 MiB Shape (1, 1, 1032, 332, 362) (1, 1, 250, 332, 362) Dask graph 5 chunks in 3 graph layers Data type float32 numpy.ndarray
- CMIP6_CV_version :
- cv=6.2.3.5-2-g63b123e
- Conventions :
- CF-1.7 CMIP-6.2
- EXPID :
- ssp370
- activity_id :
- ScenarioMIP AerChemMIP
- branch_method :
- standard
- branch_time_in_child :
- 0.0
- branch_time_in_parent :
- 60265.0
- contact :
- ipsl-cmip6@listes.ipsl.fr
- creation_date :
- 2018-12-18T20:37:37Z
- data_specs_version :
- 01.00.28
- description :
- Future scenario with high radiative forcing by the end of century. Reaches about 7.0 W/m2 by 2100; fills gap in RCP forcing pathways between 6.0 and 8.5 W/m2. Concentration-driven
- dr2xml_md5sum :
- c2dce418e78ca835be1e2ff817c2c403
- dr2xml_version :
- 1.16
- experiment :
- gap-filling scenario reaching 7.0 based on SSP3
- experiment_id :
- ssp370
- external_variables :
- areacello
- forcing_index :
- 1
- frequency :
- mon
- further_info_url :
- https://furtherinfo.es-doc.org/CMIP6.IPSL.IPSL-CM6A-LR.ssp370.none.r1i1p1f1
- grid :
- native ocean tri-polar grid with 105 k ocean cells
- grid_label :
- gn
- history :
- none
- initialization_index :
- 1
- institution :
- Institut Pierre Simon Laplace, Paris 75252, France
- institution_id :
- IPSL
- license :
- CMIP6 model data produced by IPSL is licensed under a Creative Commons Attribution-NonCommercial-ShareAlike 4.0 International License (https://creativecommons.org/licenses). Consult https://pcmdi.llnl.gov/CMIP6/TermsOfUse for terms of use governing CMIP6 output, including citation requirements and proper acknowledgment. Further information about this data, including some limitations, can be found via the further_info_url (recorded as a global attribute in this file) and at https://cmc.ipsl.fr/. The data producers and data providers make no warranty, either express or implied, including, but not limited to, warranties of merchantability and fitness for a particular purpose. All liabilities arising from the supply of the information (including any liability arising in negligence) are excluded to the fullest extent permitted by law.
- mip_era :
- CMIP6
- model_version :
- 6.1.8
- name :
- /ccc/work/cont003/gencmip6/p86caub/IGCM_OUT/IPSLCM6/PROD/ssp370/CM61-LR-scen-ssp370/CMIP6/OCE/tos_Omon_IPSL-CM6A-LR_ssp370_r1i1p1f1_gn_%start_date%-%end_date%
- nominal_resolution :
- 100 km
- parent_activity_id :
- CMIP
- parent_experiment_id :
- historical
- parent_mip_era :
- CMIP6
- parent_source_id :
- IPSL-CM6A-LR
- parent_time_units :
- days since 1850-01-01 00:00:00
- parent_variant_label :
- r1i1p1f1
- physics_index :
- 1
- product :
- model-output
- realization_index :
- 1
- realm :
- ocean
- source :
- IPSL-CM6A-LR (2017): atmos: LMDZ (NPv6, N96; 144 x 143 longitude/latitude; 79 levels; top level 40000 m) land: ORCHIDEE (v2.0, Water/Carbon/Energy mode) ocean: NEMO-OPA (eORCA1.3, tripolar primarily 1deg; 362 x 332 longitude/latitude; 75 levels; top grid cell 0-2 m) ocnBgchem: NEMO-PISCES seaIce: NEMO-LIM3
- source_id :
- IPSL-CM6A-LR
- source_type :
- AOGCM BGC
- sub_experiment :
- none
- sub_experiment_id :
- none
- table_id :
- Omon
- title :
- IPSL-CM6A-LR model output prepared for CMIP6 / ScenarioMIP AerChemMIP ssp370
- tracking_id :
- hdl:21.14100/469aced0-c00e-4848-97bc-d3ee46cb52d9
- variable_id :
- tos
- variant_info :
- Each member starts from the corresponding member of its parent experiment. Information provided by this attribute may in some cases be flawed. Users can find more comprehensive and up-to-date documentation via the further_info_url global attribute.
- variant_label :
- r1i1p1f1
- status :
- 2019-11-14;created;by nhn2@columbia.edu
- netcdf_tracking_ids :
- hdl:21.14100/469aced0-c00e-4848-97bc-d3ee46cb52d9
- version_id :
- v20190119
- intake_esm_vars :
- ['tos']
- intake_esm_attrs:activity_id :
- ScenarioMIP
- intake_esm_attrs:institution_id :
- IPSL
- intake_esm_attrs:source_id :
- IPSL-CM6A-LR
- intake_esm_attrs:experiment_id :
- ssp370
- intake_esm_attrs:member_id :
- r1i1p1f1
- intake_esm_attrs:table_id :
- Omon
- intake_esm_attrs:variable_id :
- tos
- intake_esm_attrs:grid_label :
- gn
- intake_esm_attrs:zstore :
- gs://cmip6/CMIP6/ScenarioMIP/IPSL/IPSL-CM6A-LR/ssp370/r1i1p1f1/Omon/tos/gn/v20190119/
- intake_esm_attrs:version :
- 20190119
- intake_esm_attrs:_data_format_ :
- zarr
- intake_esm_dataset_key :
- ScenarioMIP.IPSL.IPSL-CM6A-LR.ssp370.r1i1p1f1.Omon.tos.gn.gs://cmip6/CMIP6/ScenarioMIP/IPSL/IPSL-CM6A-LR/ssp370/r1i1p1f1/Omon/tos/gn/v20190119/.20190119
<xarray.DatasetView> Dimensions: (member_id: 1, y: 332, x: 362, dcpp_init_year: 1, time: 1032, vertex: 4, bnds: 2) Coordinates: (12/13) lat (y, x) float32 dask.array<chunksize=(332, 362), meta=np.ndarray> lon (y, x) float32 dask.array<chunksize=(332, 362), meta=np.ndarray> * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 lat_verticies (y, x, vertex) float32 dask.array<chunksize=(332, 362, 4), meta=np.ndarray> lon_verticies (y, x, vertex) float32 dask.array<chunksize=(332, 362, 4), meta=np.ndarray> time_bounds (time, bnds) object dask.array<chunksize=(1032, 2), meta=np.ndarray> ... ... * x (x) int64 0 1 2 3 4 5 6 7 ... 355 356 357 358 359 360 361 lon_bounds (bnds, y, x) float32 dask.array<chunksize=(1, 332, 362), meta=np.ndarray> lat_bounds (bnds, y, x) float32 dask.array<chunksize=(1, 332, 362), meta=np.ndarray> * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan areacello (member_id, dcpp_init_year, y, x) float32 dask.array<chunksize=(1, 1, 332, 362), meta=np.ndarray> Dimensions without coordinates: vertex, bnds Data variables: area (member_id, y, x) float32 dask.array<chunksize=(1, 332, 362), meta=np.ndarray> tos (member_id, dcpp_init_year, time, y, x) float32 dask.array<chunksize=(1, 1, 250, 332, 362), meta=np.ndarray> Attributes: (12/67) CMIP6_CV_version: cv=6.2.3.5-2-g63b123e Conventions: CF-1.7 CMIP-6.2 EXPID: ssp370 activity_id: ScenarioMIP AerChemMIP branch_method: standard branch_time_in_child: 0.0 ... ... intake_esm_attrs:variable_id: tos intake_esm_attrs:grid_label: gn intake_esm_attrs:zstore: gs://cmip6/CMIP6/ScenarioMIP/IPSL/IPSL-... intake_esm_attrs:version: 20190119 intake_esm_attrs:_data_format_: zarr intake_esm_dataset_key: ScenarioMIP.IPSL.IPSL-CM6A-LR.ssp370.r1...ssp370- member_id: 1
- y: 332
- x: 362
- dcpp_init_year: 1
- time: 1032
- vertex: 4
- bnds: 2
- lat(y, x)float32dask.array<chunksize=(332, 362), meta=np.ndarray>
- bounds :
- bounds_nav_lat
- long_name :
- Latitude
- standard_name :
- latitude
- units :
- degrees_north
Array Chunk Bytes 469.47 kiB 469.47 kiB Shape (332, 362) (332, 362) Dask graph 1 chunks in 5 graph layers Data type float32 numpy.ndarray - lon(y, x)float32dask.array<chunksize=(332, 362), meta=np.ndarray>
- bounds :
- bounds_nav_lon
- long_name :
- Longitude
- standard_name :
- longitude
- units :
- degrees_east
Array Chunk Bytes 469.47 kiB 469.47 kiB Shape (332, 362) (332, 362) Dask graph 1 chunks in 8 graph layers Data type float32 numpy.ndarray - time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bounds
- long_name :
- Time axis
- standard_name :
- time
- time_origin :
- 2015-01-01 00:00:00
array([cftime.DatetimeGregorian(2015, 1, 16, 12, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2015, 2, 15, 0, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2015, 3, 16, 12, 0, 0, 0, has_year_zero=False), ..., cftime.DatetimeGregorian(2100, 10, 16, 12, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2100, 11, 16, 0, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2100, 12, 16, 12, 0, 0, 0, has_year_zero=False)], dtype=object) - lat_verticies(y, x, vertex)float32dask.array<chunksize=(332, 362, 4), meta=np.ndarray>
Array Chunk Bytes 1.83 MiB 1.83 MiB Shape (332, 362, 4) (332, 362, 4) Dask graph 1 chunks in 3 graph layers Data type float32 numpy.ndarray - lon_verticies(y, x, vertex)float32dask.array<chunksize=(332, 362, 4), meta=np.ndarray>
Array Chunk Bytes 1.83 MiB 1.83 MiB Shape (332, 362, 4) (332, 362, 4) Dask graph 1 chunks in 6 graph layers Data type float32 numpy.ndarray - time_bounds(time, bnds)objectdask.array<chunksize=(1032, 2), meta=np.ndarray>
Array Chunk Bytes 16.12 kiB 16.12 kiB Shape (1032, 2) (1032, 2) Dask graph 1 chunks in 2 graph layers Data type object numpy.ndarray - y(y)int640 1 2 3 4 5 ... 327 328 329 330 331
array([ 0, 1, 2, ..., 329, 330, 331])
- x(x)int640 1 2 3 4 5 ... 357 358 359 360 361
array([ 0, 1, 2, ..., 359, 360, 361])
- lon_bounds(bnds, y, x)float32dask.array<chunksize=(1, 332, 362), meta=np.ndarray>
Array Chunk Bytes 0.92 MiB 469.47 kiB Shape (2, 332, 362) (1, 332, 362) Dask graph 2 chunks in 15 graph layers Data type float32 numpy.ndarray - lat_bounds(bnds, y, x)float32dask.array<chunksize=(1, 332, 362), meta=np.ndarray>
Array Chunk Bytes 0.92 MiB 469.47 kiB Shape (2, 332, 362) (1, 332, 362) Dask graph 2 chunks in 12 graph layers Data type float32 numpy.ndarray - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- areacello(member_id, dcpp_init_year, y, x)float32dask.array<chunksize=(1, 1, 332, 362), meta=np.ndarray>
- cell_measures :
- area: area
- cell_methods :
- area: sum
- description :
- Cell areas for any grid used to report ocean variables and variables which are requested as used on the model ocean grid (e.g. hfsso, which is a downward heat flux from the atmosphere interpolated onto the ocean grid). These cell areas should be defined to enable exact calculation of global integrals (e.g., of vertical fluxes of energy at the surface and top of the atmosphere).
- history :
- none
- long_name :
- Grid-Cell Area
- online_operation :
- once
- standard_name :
- cell_area
- units :
- m²
- original_key :
- ScenarioMIP.IPSL.IPSL-CM6A-LR.ssp245.r1i1p1f1.Ofx.areacello.gn.gs://cmip6/CMIP6/ScenarioMIP/IPSL/IPSL-CM6A-LR/ssp245/r1i1p1f1/Ofx/areacello/gn/v20190119/.20190119
- parsed_with :
- xmip/postprocessing/_parse_metric
Array Chunk Bytes 469.47 kiB 469.47 kiB Shape (1, 1, 332, 362) (1, 1, 332, 362) Dask graph 1 chunks in 3 graph layers Data type float32 numpy.ndarray
- area(member_id, y, x)float32dask.array<chunksize=(1, 332, 362), meta=np.ndarray>
- standard_name :
- cell_area
- units :
- m²
Array Chunk Bytes 469.47 kiB 469.47 kiB Shape (1, 332, 362) (1, 332, 362) Dask graph 1 chunks in 3 graph layers Data type float32 numpy.ndarray - tos(member_id, dcpp_init_year, time, y, x)float32dask.array<chunksize=(1, 1, 120, 332, 362), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- description :
- This may differ from "surface temperature" in regions of sea ice or floating ice shelves. For models using conservative temperature as the prognostic field, they should report the top ocean layer as surface potential temperature, which is the same as surface in situ temperature.
- history :
- none
- interval_operation :
- 2700 s
- interval_write :
- 1 month
- long_name :
- Sea Surface Temperature
- online_operation :
- average
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 473.14 MiB 55.02 MiB Shape (1, 1, 1032, 332, 362) (1, 1, 120, 332, 362) Dask graph 9 chunks in 3 graph layers Data type float32 numpy.ndarray
- CMIP6_CV_version :
- cv=6.2.3.5-2-g63b123e
- Conventions :
- CF-1.7 CMIP-6.2
- EXPID :
- ssp245
- activity_id :
- ScenarioMIP
- branch_method :
- standard
- branch_time_in_child :
- 0.0
- branch_time_in_parent :
- 60265.0
- contact :
- ipsl-cmip6@listes.ipsl.fr
- creation_date :
- 2018-12-05T17:25:51Z
- data_specs_version :
- 01.00.28
- description :
- Future scenario with medium radiative forcing by the end of century. Following approximately RCP4.5 global forcing pathway but with new forcing based on SSP2. Concentration-driven
- dr2xml_md5sum :
- c2dce418e78ca835be1e2ff817c2c403
- dr2xml_version :
- 1.16
- experiment :
- update of RCP4.5 based on SSP2
- experiment_id :
- ssp245
- external_variables :
- areacello
- forcing_index :
- 1
- frequency :
- mon
- further_info_url :
- https://furtherinfo.es-doc.org/CMIP6.IPSL.IPSL-CM6A-LR.ssp245.none.r1i1p1f1
- grid :
- native ocean tri-polar grid with 105 k ocean cells
- grid_label :
- gn
- history :
- none
- initialization_index :
- 1
- institution :
- Institut Pierre Simon Laplace, Paris 75252, France
- institution_id :
- IPSL
- license :
- CMIP6 model data produced by IPSL is licensed under a Creative Commons Attribution-NonCommercial-ShareAlike 4.0 International License (https://creativecommons.org/licenses). Consult https://pcmdi.llnl.gov/CMIP6/TermsOfUse for terms of use governing CMIP6 output, including citation requirements and proper acknowledgment. Further information about this data, including some limitations, can be found via the further_info_url (recorded as a global attribute in this file) and at https://cmc.ipsl.fr/. The data producers and data providers make no warranty, either express or implied, including, but not limited to, warranties of merchantability and fitness for a particular purpose. All liabilities arising from the supply of the information (including any liability arising in negligence) are excluded to the fullest extent permitted by law.
- mip_era :
- CMIP6
- model_version :
- 6.1.8
- name :
- /ccc/work/cont003/gencmip6/lurtont/IGCM_OUT/IPSLCM6/PROD/ssp245/CM61-LR-scen-ssp245/CMIP6/OCE/tos_Omon_IPSL-CM6A-LR_ssp245_r1i1p1f1_gn_%start_date%-%end_date%
- nominal_resolution :
- 100 km
- parent_activity_id :
- CMIP
- parent_experiment_id :
- historical
- parent_mip_era :
- CMIP6
- parent_source_id :
- IPSL-CM6A-LR
- parent_time_units :
- days since 1850-01-01 00:00:00
- parent_variant_label :
- r1i1p1f1
- physics_index :
- 1
- product :
- model-output
- realization_index :
- 1
- realm :
- ocean
- source :
- IPSL-CM6A-LR (2017): atmos: LMDZ (NPv6, N96; 144 x 143 longitude/latitude; 79 levels; top level 40000 m) land: ORCHIDEE (v2.0, Water/Carbon/Energy mode) ocean: NEMO-OPA (eORCA1.3, tripolar primarily 1deg; 362 x 332 longitude/latitude; 75 levels; top grid cell 0-2 m) ocnBgchem: NEMO-PISCES seaIce: NEMO-LIM3
- source_id :
- IPSL-CM6A-LR
- source_type :
- AOGCM BGC
- sub_experiment :
- none
- sub_experiment_id :
- none
- table_id :
- Omon
- title :
- IPSL-CM6A-LR model output prepared for CMIP6 / ScenarioMIP ssp245
- tracking_id :
- hdl:21.14100/d0cf25a0-2c33-4730-bffa-de0fadcbed68
- variable_id :
- tos
- variant_info :
- Each member starts from the corresponding member of its parent experiment. Information provided by this attribute may in some cases be flawed. Users can find more comprehensive and up-to-date documentation via the further_info_url global attribute.
- variant_label :
- r1i1p1f1
- status :
- 2019-10-25;created;by nhn2@columbia.edu
- netcdf_tracking_ids :
- hdl:21.14100/d0cf25a0-2c33-4730-bffa-de0fadcbed68
- version_id :
- v20190119
- intake_esm_vars :
- ['tos']
- intake_esm_attrs:activity_id :
- ScenarioMIP
- intake_esm_attrs:institution_id :
- IPSL
- intake_esm_attrs:source_id :
- IPSL-CM6A-LR
- intake_esm_attrs:experiment_id :
- ssp245
- intake_esm_attrs:member_id :
- r1i1p1f1
- intake_esm_attrs:table_id :
- Omon
- intake_esm_attrs:variable_id :
- tos
- intake_esm_attrs:grid_label :
- gn
- intake_esm_attrs:zstore :
- gs://cmip6/CMIP6/ScenarioMIP/IPSL/IPSL-CM6A-LR/ssp245/r1i1p1f1/Omon/tos/gn/v20190119/
- intake_esm_attrs:version :
- 20190119
- intake_esm_attrs:_data_format_ :
- zarr
- intake_esm_dataset_key :
- ScenarioMIP.IPSL.IPSL-CM6A-LR.ssp245.r1i1p1f1.Omon.tos.gn.gs://cmip6/CMIP6/ScenarioMIP/IPSL/IPSL-CM6A-LR/ssp245/r1i1p1f1/Omon/tos/gn/v20190119/.20190119
<xarray.DatasetView> Dimensions: (member_id: 1, y: 332, x: 362, dcpp_init_year: 1, time: 1032, vertex: 4, bnds: 2) Coordinates: (12/13) lat (y, x) float32 dask.array<chunksize=(332, 362), meta=np.ndarray> lon (y, x) float32 dask.array<chunksize=(332, 362), meta=np.ndarray> * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 lat_verticies (y, x, vertex) float32 dask.array<chunksize=(332, 362, 4), meta=np.ndarray> lon_verticies (y, x, vertex) float32 dask.array<chunksize=(332, 362, 4), meta=np.ndarray> time_bounds (time, bnds) object dask.array<chunksize=(1032, 2), meta=np.ndarray> ... ... * x (x) int64 0 1 2 3 4 5 6 7 ... 355 356 357 358 359 360 361 lon_bounds (bnds, y, x) float32 dask.array<chunksize=(1, 332, 362), meta=np.ndarray> lat_bounds (bnds, y, x) float32 dask.array<chunksize=(1, 332, 362), meta=np.ndarray> * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan areacello (member_id, dcpp_init_year, y, x) float32 dask.array<chunksize=(1, 1, 332, 362), meta=np.ndarray> Dimensions without coordinates: vertex, bnds Data variables: area (member_id, y, x) float32 dask.array<chunksize=(1, 332, 362), meta=np.ndarray> tos (member_id, dcpp_init_year, time, y, x) float32 dask.array<chunksize=(1, 1, 120, 332, 362), meta=np.ndarray> Attributes: (12/67) CMIP6_CV_version: cv=6.2.3.5-2-g63b123e Conventions: CF-1.7 CMIP-6.2 EXPID: ssp245 activity_id: ScenarioMIP branch_method: standard branch_time_in_child: 0.0 ... ... intake_esm_attrs:variable_id: tos intake_esm_attrs:grid_label: gn intake_esm_attrs:zstore: gs://cmip6/CMIP6/ScenarioMIP/IPSL/IPSL-... intake_esm_attrs:version: 20190119 intake_esm_attrs:_data_format_: zarr intake_esm_dataset_key: ScenarioMIP.IPSL.IPSL-CM6A-LR.ssp245.r1...ssp245
<xarray.DatasetView> Dimensions: () Data variables: *empty*IPSL-CM6A-LR- member_id: 1
- dcpp_init_year: 1
- time: 1032
- y: 220
- x: 256
- vertex: 4
- bnds: 2
- lat(y, x)float64dask.array<chunksize=(220, 256), meta=np.ndarray>
- bounds :
- vertices_latitude
- long_name :
- latitude
- standard_name :
- latitude
- units :
- degrees_north
Array Chunk Bytes 440.00 kiB 440.00 kiB Shape (220, 256) (220, 256) Dask graph 1 chunks in 5 graph layers Data type float64 numpy.ndarray - lon(y, x)float64dask.array<chunksize=(220, 256), meta=np.ndarray>
- bounds :
- vertices_longitude
- long_name :
- longitude
- standard_name :
- longitude
- units :
- degrees_east
Array Chunk Bytes 440.00 kiB 440.00 kiB Shape (220, 256) (220, 256) Dask graph 1 chunks in 8 graph layers Data type float64 numpy.ndarray - time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bnds
- long_name :
- time
- standard_name :
- time
array([cftime.DatetimeProlepticGregorian(2015, 1, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2015, 2, 15, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2015, 3, 16, 12, 0, 0, 0, has_year_zero=True), ..., cftime.DatetimeProlepticGregorian(2100, 10, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2100, 11, 16, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2100, 12, 16, 12, 0, 0, 0, has_year_zero=True)], dtype=object) - lat_verticies(y, x, vertex)float64dask.array<chunksize=(220, 256, 4), meta=np.ndarray>
- units :
- degrees_north
Array Chunk Bytes 1.72 MiB 1.72 MiB Shape (220, 256, 4) (220, 256, 4) Dask graph 1 chunks in 3 graph layers Data type float64 numpy.ndarray - lon_verticies(y, x, vertex)float64dask.array<chunksize=(220, 256, 4), meta=np.ndarray>
- units :
- degrees_east
Array Chunk Bytes 1.72 MiB 1.72 MiB Shape (220, 256, 4) (220, 256, 4) Dask graph 1 chunks in 6 graph layers Data type float64 numpy.ndarray - time_bounds(time, bnds)objectdask.array<chunksize=(1032, 2), meta=np.ndarray>
Array Chunk Bytes 16.12 kiB 16.12 kiB Shape (1032, 2) (1032, 2) Dask graph 1 chunks in 2 graph layers Data type object numpy.ndarray - y(y)int640 1 2 3 4 5 ... 215 216 217 218 219
array([ 0, 1, 2, ..., 217, 218, 219])
- x(x)int640 1 2 3 4 5 ... 251 252 253 254 255
array([ 0, 1, 2, ..., 253, 254, 255])
- lon_bounds(bnds, y, x)float64dask.array<chunksize=(1, 220, 256), meta=np.ndarray>
- units :
- degrees_east
Array Chunk Bytes 880.00 kiB 440.00 kiB Shape (2, 220, 256) (1, 220, 256) Dask graph 2 chunks in 15 graph layers Data type float64 numpy.ndarray - lat_bounds(bnds, y, x)float64dask.array<chunksize=(1, 220, 256), meta=np.ndarray>
- units :
- degrees_north
Array Chunk Bytes 880.00 kiB 440.00 kiB Shape (2, 220, 256) (1, 220, 256) Dask graph 2 chunks in 12 graph layers Data type float64 numpy.ndarray - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- areacello(member_id, dcpp_init_year, y, x)float32dask.array<chunksize=(1, 1, 220, 256), meta=np.ndarray>
- cell_methods :
- area: sum
- comment :
- Horizontal area of ocean grid cells
- history :
- 2019-10-29T16:23:28Z altered by CMOR: replaced missing value flag (-9e+33) and corresponding data with standard missing value (1e+20).
- long_name :
- Grid-Cell Area for Ocean Variables
- original_name :
- areacello
- standard_name :
- cell_area
- units :
- m²
- original_key :
- ScenarioMIP.MPI-M.MPI-ESM1-2-LR.ssp126.r1i1p1f1.Ofx.areacello.gn.gs://cmip6/CMIP6/ScenarioMIP/MPI-M/MPI-ESM1-2-LR/ssp126/r1i1p1f1/Ofx/areacello/gn/v20190710/.20190710
- parsed_with :
- xmip/postprocessing/_parse_metric
Array Chunk Bytes 220.00 kiB 220.00 kiB Shape (1, 1, 220, 256) (1, 1, 220, 256) Dask graph 1 chunks in 3 graph layers Data type float32 numpy.ndarray
- tos(member_id, dcpp_init_year, time, y, x)float32dask.array<chunksize=(1, 1, 394, 220, 256), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- comment :
- Temperature of upper boundary of the liquid ocean, including temperatures below sea-ice and floating ice shelves.
- long_name :
- Sea Surface Temperature
- original_name :
- tos
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 221.72 MiB 84.65 MiB Shape (1, 1, 1032, 220, 256) (1, 1, 394, 220, 256) Dask graph 3 chunks in 3 graph layers Data type float32 numpy.ndarray
- Conventions :
- CF-1.7 CMIP-6.2
- activity_id :
- ScenarioMIP
- branch_method :
- standard
- branch_time_in_child :
- 60265.0
- branch_time_in_parent :
- 60265.0
- cmor_version :
- 3.5.0
- contact :
- cmip6-mpi-esm@dkrz.de
- creation_date :
- 2019-10-29T16:23:28Z
- data_specs_version :
- 01.00.30
- experiment :
- update of RCP2.6 based on SSP1
- experiment_id :
- ssp126
- external_variables :
- areacello
- forcing_index :
- 1
- frequency :
- mon
- further_info_url :
- https://furtherinfo.es-doc.org/CMIP6.MPI-M.MPI-ESM1-2-LR.ssp126.none.r1i1p1f1
- grid :
- gn
- grid_label :
- gn
- history :
- 2019-10-29T16:23:28Z ; CMOR rewrote data to be consistent with CMIP6, CF-1.7 CMIP-6.2 and CF standards.
- initialization_index :
- 1
- institution :
- Max Planck Institute for Meteorology, Hamburg 20146, Germany
- institution_id :
- MPI-M
- license :
- CMIP6 model data produced by MPI-M is licensed under a Creative Commons Attribution ShareAlike 4.0 International License (https://creativecommons.org/licenses). Consult https://pcmdi.llnl.gov/CMIP6/TermsOfUse for terms of use governing CMIP6 output, including citation requirements and proper acknowledgment. Further information about this data, including some limitations, can be found via the further_info_url (recorded as a global attribute in this file) and. The data producers and data providers make no warranty, either express or implied, including, but not limited to, warranties of merchantability and fitness for a particular purpose. All liabilities arising from the supply of the information (including any liability arising in negligence) are excluded to the fullest extent permitted by law.
- mip_era :
- CMIP6
- nominal_resolution :
- 250 km
- parent_activity_id :
- CMIP
- parent_experiment_id :
- historical
- parent_mip_era :
- CMIP6
- parent_source_id :
- MPI-ESM1-2-LR
- parent_time_units :
- days since 1850-1-1 00:00:00
- parent_variant_label :
- r1i1p1f1
- physics_index :
- 1
- product :
- model-output
- project_id :
- CMIP6
- realization_index :
- 1
- realm :
- ocean
- references :
- MPI-ESM: Mauritsen, T. et al. (2019), Developments in the MPI‐M Earth System Model version 1.2 (MPI‐ESM1.2) and Its Response to Increasing CO2, J. Adv. Model. Earth Syst.,11, 998-1038, doi:10.1029/2018MS001400, Mueller, W.A. et al. (2018): A high‐resolution version of the Max Planck Institute Earth System Model MPI‐ESM1.2‐HR. J. Adv. Model. EarthSyst.,10,1383–1413, doi:10.1029/2017MS001217
- source :
- MPI-ESM1.2-LR (2017): aerosol: none, prescribed MACv2-SP atmos: ECHAM6.3 (spectral T63; 192 x 96 longitude/latitude; 47 levels; top level 0.01 hPa) atmosChem: none land: JSBACH3.20 landIce: none/prescribed ocean: MPIOM1.63 (bipolar GR1.5, approximately 1.5deg; 256 x 220 longitude/latitude; 40 levels; top grid cell 0-12 m) ocnBgchem: HAMOCC6 seaIce: unnamed (thermodynamic (Semtner zero-layer) dynamic (Hibler 79) sea ice model)
- source_id :
- MPI-ESM1-2-LR
- source_type :
- AOGCM
- status :
- 2020-03-21;created; by gcs.cmip6.ldeo@gmail.com
- sub_experiment :
- none
- sub_experiment_id :
- none
- table_id :
- Omon
- table_info :
- Creation Date:(09 May 2019) MD5:e6ef8ececc8f338646ebfb3aeed36bfc
- title :
- MPI-ESM1-2-LR output prepared for CMIP6
- tracking_id :
- hdl:21.14100/1128a79c-b5c7-44d4-9e96-6e39ca21402c hdl:21.14100/c1344808-5dcf-4a92-adf4-4c7c2fbd4c32 hdl:21.14100/677125b6-828a-4e16-ae19-0b77a70365ff hdl:21.14100/7dc107ca-a429-4d6a-9796-356b65983f88 hdl:21.14100/f94f87e6-49b1-4117-9ac3-3866658fd116
- variable_id :
- tos
- variant_label :
- r1i1p1f1
- netcdf_tracking_ids :
- hdl:21.14100/1128a79c-b5c7-44d4-9e96-6e39ca21402c hdl:21.14100/c1344808-5dcf-4a92-adf4-4c7c2fbd4c32 hdl:21.14100/677125b6-828a-4e16-ae19-0b77a70365ff hdl:21.14100/7dc107ca-a429-4d6a-9796-356b65983f88 hdl:21.14100/f94f87e6-49b1-4117-9ac3-3866658fd116
- version_id :
- v20190710
- intake_esm_vars :
- ['tos']
- intake_esm_attrs:activity_id :
- ScenarioMIP
- intake_esm_attrs:institution_id :
- MPI-M
- intake_esm_attrs:source_id :
- MPI-ESM1-2-LR
- intake_esm_attrs:experiment_id :
- ssp126
- intake_esm_attrs:member_id :
- r1i1p1f1
- intake_esm_attrs:table_id :
- Omon
- intake_esm_attrs:variable_id :
- tos
- intake_esm_attrs:grid_label :
- gn
- intake_esm_attrs:zstore :
- gs://cmip6/CMIP6/ScenarioMIP/MPI-M/MPI-ESM1-2-LR/ssp126/r1i1p1f1/Omon/tos/gn/v20190710/
- intake_esm_attrs:version :
- 20190710
- intake_esm_attrs:_data_format_ :
- zarr
- intake_esm_dataset_key :
- ScenarioMIP.MPI-M.MPI-ESM1-2-LR.ssp126.r1i1p1f1.Omon.tos.gn.gs://cmip6/CMIP6/ScenarioMIP/MPI-M/MPI-ESM1-2-LR/ssp126/r1i1p1f1/Omon/tos/gn/v20190710/.20190710
<xarray.DatasetView> Dimensions: (member_id: 1, dcpp_init_year: 1, time: 1032, y: 220, x: 256, vertex: 4, bnds: 2) Coordinates: (12/13) lat (y, x) float64 dask.array<chunksize=(220, 256), meta=np.ndarray> lon (y, x) float64 dask.array<chunksize=(220, 256), meta=np.ndarray> * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 lat_verticies (y, x, vertex) float64 dask.array<chunksize=(220, 256, 4), meta=np.ndarray> lon_verticies (y, x, vertex) float64 dask.array<chunksize=(220, 256, 4), meta=np.ndarray> time_bounds (time, bnds) object dask.array<chunksize=(1032, 2), meta=np.ndarray> ... ... * x (x) int64 0 1 2 3 4 5 6 7 ... 249 250 251 252 253 254 255 lon_bounds (bnds, y, x) float64 dask.array<chunksize=(1, 220, 256), meta=np.ndarray> lat_bounds (bnds, y, x) float64 dask.array<chunksize=(1, 220, 256), meta=np.ndarray> * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan areacello (member_id, dcpp_init_year, y, x) float32 dask.array<chunksize=(1, 1, 220, 256), meta=np.ndarray> Dimensions without coordinates: vertex, bnds Data variables: tos (member_id, dcpp_init_year, time, y, x) float32 dask.array<chunksize=(1, 1, 394, 220, 256), meta=np.ndarray> Attributes: (12/63) Conventions: CF-1.7 CMIP-6.2 activity_id: ScenarioMIP branch_method: standard branch_time_in_child: 60265.0 branch_time_in_parent: 60265.0 cmor_version: 3.5.0 ... ... intake_esm_attrs:variable_id: tos intake_esm_attrs:grid_label: gn intake_esm_attrs:zstore: gs://cmip6/CMIP6/ScenarioMIP/MPI-M/MPI-... intake_esm_attrs:version: 20190710 intake_esm_attrs:_data_format_: zarr intake_esm_dataset_key: ScenarioMIP.MPI-M.MPI-ESM1-2-LR.ssp126....ssp126- member_id: 1
- dcpp_init_year: 1
- time: 1032
- y: 220
- x: 256
- vertex: 4
- bnds: 2
- lat(y, x)float64dask.array<chunksize=(220, 256), meta=np.ndarray>
- bounds :
- vertices_latitude
- long_name :
- latitude
- standard_name :
- latitude
- units :
- degrees_north
Array Chunk Bytes 440.00 kiB 440.00 kiB Shape (220, 256) (220, 256) Dask graph 1 chunks in 5 graph layers Data type float64 numpy.ndarray - lon(y, x)float64dask.array<chunksize=(220, 256), meta=np.ndarray>
- bounds :
- vertices_longitude
- long_name :
- longitude
- standard_name :
- longitude
- units :
- degrees_east
Array Chunk Bytes 440.00 kiB 440.00 kiB Shape (220, 256) (220, 256) Dask graph 1 chunks in 8 graph layers Data type float64 numpy.ndarray - time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bnds
- long_name :
- time
- standard_name :
- time
array([cftime.DatetimeProlepticGregorian(2015, 1, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2015, 2, 15, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2015, 3, 16, 12, 0, 0, 0, has_year_zero=True), ..., cftime.DatetimeProlepticGregorian(2100, 10, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2100, 11, 16, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2100, 12, 16, 12, 0, 0, 0, has_year_zero=True)], dtype=object) - lat_verticies(y, x, vertex)float64dask.array<chunksize=(220, 256, 4), meta=np.ndarray>
- units :
- degrees_north
Array Chunk Bytes 1.72 MiB 1.72 MiB Shape (220, 256, 4) (220, 256, 4) Dask graph 1 chunks in 3 graph layers Data type float64 numpy.ndarray - lon_verticies(y, x, vertex)float64dask.array<chunksize=(220, 256, 4), meta=np.ndarray>
- units :
- degrees_east
Array Chunk Bytes 1.72 MiB 1.72 MiB Shape (220, 256, 4) (220, 256, 4) Dask graph 1 chunks in 6 graph layers Data type float64 numpy.ndarray - time_bounds(time, bnds)objectdask.array<chunksize=(1032, 2), meta=np.ndarray>
Array Chunk Bytes 16.12 kiB 16.12 kiB Shape (1032, 2) (1032, 2) Dask graph 1 chunks in 2 graph layers Data type object numpy.ndarray - y(y)int640 1 2 3 4 5 ... 215 216 217 218 219
array([ 0, 1, 2, ..., 217, 218, 219])
- x(x)int640 1 2 3 4 5 ... 251 252 253 254 255
array([ 0, 1, 2, ..., 253, 254, 255])
- lon_bounds(bnds, y, x)float64dask.array<chunksize=(1, 220, 256), meta=np.ndarray>
- units :
- degrees_east
Array Chunk Bytes 880.00 kiB 440.00 kiB Shape (2, 220, 256) (1, 220, 256) Dask graph 2 chunks in 15 graph layers Data type float64 numpy.ndarray - lat_bounds(bnds, y, x)float64dask.array<chunksize=(1, 220, 256), meta=np.ndarray>
- units :
- degrees_north
Array Chunk Bytes 880.00 kiB 440.00 kiB Shape (2, 220, 256) (1, 220, 256) Dask graph 2 chunks in 12 graph layers Data type float64 numpy.ndarray - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- areacello(member_id, dcpp_init_year, y, x)float32dask.array<chunksize=(1, 1, 220, 256), meta=np.ndarray>
- cell_methods :
- area: sum
- comment :
- Horizontal area of ocean grid cells
- history :
- 2019-11-05T12:36:43Z altered by CMOR: replaced missing value flag (-9e+33) and corresponding data with standard missing value (1e+20).
- long_name :
- Grid-Cell Area for Ocean Variables
- original_name :
- areacello
- standard_name :
- cell_area
- units :
- m²
- original_key :
- ScenarioMIP.MPI-M.MPI-ESM1-2-LR.ssp245.r1i1p1f1.Ofx.areacello.gn.gs://cmip6/CMIP6/ScenarioMIP/MPI-M/MPI-ESM1-2-LR/ssp245/r1i1p1f1/Ofx/areacello/gn/v20190710/.20190710
- parsed_with :
- xmip/postprocessing/_parse_metric
Array Chunk Bytes 220.00 kiB 220.00 kiB Shape (1, 1, 220, 256) (1, 1, 220, 256) Dask graph 1 chunks in 3 graph layers Data type float32 numpy.ndarray
- tos(member_id, dcpp_init_year, time, y, x)float32dask.array<chunksize=(1, 1, 394, 220, 256), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- comment :
- Temperature of upper boundary of the liquid ocean, including temperatures below sea-ice and floating ice shelves.
- long_name :
- Sea Surface Temperature
- original_name :
- tos
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 221.72 MiB 84.65 MiB Shape (1, 1, 1032, 220, 256) (1, 1, 394, 220, 256) Dask graph 3 chunks in 3 graph layers Data type float32 numpy.ndarray
- Conventions :
- CF-1.7 CMIP-6.2
- activity_id :
- ScenarioMIP
- branch_method :
- standard
- branch_time_in_child :
- 60265.0
- branch_time_in_parent :
- 60265.0
- cmor_version :
- 3.5.0
- contact :
- cmip6-mpi-esm@dkrz.de
- creation_date :
- 2019-11-05T12:36:43Z
- data_specs_version :
- 01.00.30
- experiment :
- update of RCP4.5 based on SSP2
- experiment_id :
- ssp245
- external_variables :
- areacello
- forcing_index :
- 1
- frequency :
- mon
- further_info_url :
- https://furtherinfo.es-doc.org/CMIP6.MPI-M.MPI-ESM1-2-LR.ssp245.none.r1i1p1f1
- grid :
- gn
- grid_label :
- gn
- history :
- 2019-11-05T12:36:43Z ; CMOR rewrote data to be consistent with CMIP6, CF-1.7 CMIP-6.2 and CF standards.
- initialization_index :
- 1
- institution :
- Max Planck Institute for Meteorology, Hamburg 20146, Germany
- institution_id :
- MPI-M
- license :
- CMIP6 model data produced by MPI-M is licensed under a Creative Commons Attribution ShareAlike 4.0 International License (https://creativecommons.org/licenses). Consult https://pcmdi.llnl.gov/CMIP6/TermsOfUse for terms of use governing CMIP6 output, including citation requirements and proper acknowledgment. Further information about this data, including some limitations, can be found via the further_info_url (recorded as a global attribute in this file) and. The data producers and data providers make no warranty, either express or implied, including, but not limited to, warranties of merchantability and fitness for a particular purpose. All liabilities arising from the supply of the information (including any liability arising in negligence) are excluded to the fullest extent permitted by law.
- mip_era :
- CMIP6
- nominal_resolution :
- 250 km
- parent_activity_id :
- CMIP
- parent_experiment_id :
- historical
- parent_mip_era :
- CMIP6
- parent_source_id :
- MPI-ESM1-2-LR
- parent_time_units :
- days since 1850-1-1 00:00:00
- parent_variant_label :
- r1i1p1f1
- physics_index :
- 1
- product :
- model-output
- project_id :
- CMIP6
- realization_index :
- 1
- realm :
- ocean
- references :
- MPI-ESM: Mauritsen, T. et al. (2019), Developments in the MPI‐M Earth System Model version 1.2 (MPI‐ESM1.2) and Its Response to Increasing CO2, J. Adv. Model. Earth Syst.,11, 998-1038, doi:10.1029/2018MS001400, Mueller, W.A. et al. (2018): A high‐resolution version of the Max Planck Institute Earth System Model MPI‐ESM1.2‐HR. J. Adv. Model. EarthSyst.,10,1383–1413, doi:10.1029/2017MS001217
- source :
- MPI-ESM1.2-LR (2017): aerosol: none, prescribed MACv2-SP atmos: ECHAM6.3 (spectral T63; 192 x 96 longitude/latitude; 47 levels; top level 0.01 hPa) atmosChem: none land: JSBACH3.20 landIce: none/prescribed ocean: MPIOM1.63 (bipolar GR1.5, approximately 1.5deg; 256 x 220 longitude/latitude; 40 levels; top grid cell 0-12 m) ocnBgchem: HAMOCC6 seaIce: unnamed (thermodynamic (Semtner zero-layer) dynamic (Hibler 79) sea ice model)
- source_id :
- MPI-ESM1-2-LR
- source_type :
- AOGCM
- status :
- 2020-03-21;created; by gcs.cmip6.ldeo@gmail.com
- sub_experiment :
- none
- sub_experiment_id :
- none
- table_id :
- Omon
- table_info :
- Creation Date:(09 May 2019) MD5:e6ef8ececc8f338646ebfb3aeed36bfc
- title :
- MPI-ESM1-2-LR output prepared for CMIP6
- tracking_id :
- hdl:21.14100/7c9cdb39-c5d8-4ac7-bd82-da75b91390b1 hdl:21.14100/126253ca-3103-40b6-b375-ee4508548010 hdl:21.14100/0088eda7-e845-418a-8de3-1943d82a394b hdl:21.14100/c5812c6c-6301-40fd-ae20-c5b1477df2b0 hdl:21.14100/a98a86c4-946e-4ad3-b706-19f295b5df9c
- variable_id :
- tos
- variant_label :
- r1i1p1f1
- netcdf_tracking_ids :
- hdl:21.14100/7c9cdb39-c5d8-4ac7-bd82-da75b91390b1 hdl:21.14100/126253ca-3103-40b6-b375-ee4508548010 hdl:21.14100/0088eda7-e845-418a-8de3-1943d82a394b hdl:21.14100/c5812c6c-6301-40fd-ae20-c5b1477df2b0 hdl:21.14100/a98a86c4-946e-4ad3-b706-19f295b5df9c
- version_id :
- v20190710
- intake_esm_vars :
- ['tos']
- intake_esm_attrs:activity_id :
- ScenarioMIP
- intake_esm_attrs:institution_id :
- MPI-M
- intake_esm_attrs:source_id :
- MPI-ESM1-2-LR
- intake_esm_attrs:experiment_id :
- ssp245
- intake_esm_attrs:member_id :
- r1i1p1f1
- intake_esm_attrs:table_id :
- Omon
- intake_esm_attrs:variable_id :
- tos
- intake_esm_attrs:grid_label :
- gn
- intake_esm_attrs:zstore :
- gs://cmip6/CMIP6/ScenarioMIP/MPI-M/MPI-ESM1-2-LR/ssp245/r1i1p1f1/Omon/tos/gn/v20190710/
- intake_esm_attrs:version :
- 20190710
- intake_esm_attrs:_data_format_ :
- zarr
- intake_esm_dataset_key :
- ScenarioMIP.MPI-M.MPI-ESM1-2-LR.ssp245.r1i1p1f1.Omon.tos.gn.gs://cmip6/CMIP6/ScenarioMIP/MPI-M/MPI-ESM1-2-LR/ssp245/r1i1p1f1/Omon/tos/gn/v20190710/.20190710
<xarray.DatasetView> Dimensions: (member_id: 1, dcpp_init_year: 1, time: 1032, y: 220, x: 256, vertex: 4, bnds: 2) Coordinates: (12/13) lat (y, x) float64 dask.array<chunksize=(220, 256), meta=np.ndarray> lon (y, x) float64 dask.array<chunksize=(220, 256), meta=np.ndarray> * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 lat_verticies (y, x, vertex) float64 dask.array<chunksize=(220, 256, 4), meta=np.ndarray> lon_verticies (y, x, vertex) float64 dask.array<chunksize=(220, 256, 4), meta=np.ndarray> time_bounds (time, bnds) object dask.array<chunksize=(1032, 2), meta=np.ndarray> ... ... * x (x) int64 0 1 2 3 4 5 6 7 ... 249 250 251 252 253 254 255 lon_bounds (bnds, y, x) float64 dask.array<chunksize=(1, 220, 256), meta=np.ndarray> lat_bounds (bnds, y, x) float64 dask.array<chunksize=(1, 220, 256), meta=np.ndarray> * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan areacello (member_id, dcpp_init_year, y, x) float32 dask.array<chunksize=(1, 1, 220, 256), meta=np.ndarray> Dimensions without coordinates: vertex, bnds Data variables: tos (member_id, dcpp_init_year, time, y, x) float32 dask.array<chunksize=(1, 1, 394, 220, 256), meta=np.ndarray> Attributes: (12/63) Conventions: CF-1.7 CMIP-6.2 activity_id: ScenarioMIP branch_method: standard branch_time_in_child: 60265.0 branch_time_in_parent: 60265.0 cmor_version: 3.5.0 ... ... intake_esm_attrs:variable_id: tos intake_esm_attrs:grid_label: gn intake_esm_attrs:zstore: gs://cmip6/CMIP6/ScenarioMIP/MPI-M/MPI-... intake_esm_attrs:version: 20190710 intake_esm_attrs:_data_format_: zarr intake_esm_dataset_key: ScenarioMIP.MPI-M.MPI-ESM1-2-LR.ssp245....ssp245- member_id: 1
- dcpp_init_year: 1
- time: 1032
- y: 220
- x: 256
- vertex: 4
- bnds: 2
- lat(y, x)float64dask.array<chunksize=(220, 256), meta=np.ndarray>
- bounds :
- vertices_latitude
- long_name :
- latitude
- standard_name :
- latitude
- units :
- degrees_north
Array Chunk Bytes 440.00 kiB 440.00 kiB Shape (220, 256) (220, 256) Dask graph 1 chunks in 5 graph layers Data type float64 numpy.ndarray - lon(y, x)float64dask.array<chunksize=(220, 256), meta=np.ndarray>
- bounds :
- vertices_longitude
- long_name :
- longitude
- standard_name :
- longitude
- units :
- degrees_east
Array Chunk Bytes 440.00 kiB 440.00 kiB Shape (220, 256) (220, 256) Dask graph 1 chunks in 8 graph layers Data type float64 numpy.ndarray - time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bnds
- long_name :
- time
- standard_name :
- time
array([cftime.DatetimeProlepticGregorian(2015, 1, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2015, 2, 15, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2015, 3, 16, 12, 0, 0, 0, has_year_zero=True), ..., cftime.DatetimeProlepticGregorian(2100, 10, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2100, 11, 16, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2100, 12, 16, 12, 0, 0, 0, has_year_zero=True)], dtype=object) - lat_verticies(y, x, vertex)float64dask.array<chunksize=(220, 256, 4), meta=np.ndarray>
- units :
- degrees_north
Array Chunk Bytes 1.72 MiB 1.72 MiB Shape (220, 256, 4) (220, 256, 4) Dask graph 1 chunks in 3 graph layers Data type float64 numpy.ndarray - lon_verticies(y, x, vertex)float64dask.array<chunksize=(220, 256, 4), meta=np.ndarray>
- units :
- degrees_east
Array Chunk Bytes 1.72 MiB 1.72 MiB Shape (220, 256, 4) (220, 256, 4) Dask graph 1 chunks in 6 graph layers Data type float64 numpy.ndarray - time_bounds(time, bnds)objectdask.array<chunksize=(1032, 2), meta=np.ndarray>
Array Chunk Bytes 16.12 kiB 16.12 kiB Shape (1032, 2) (1032, 2) Dask graph 1 chunks in 2 graph layers Data type object numpy.ndarray - y(y)int640 1 2 3 4 5 ... 215 216 217 218 219
array([ 0, 1, 2, ..., 217, 218, 219])
- x(x)int640 1 2 3 4 5 ... 251 252 253 254 255
array([ 0, 1, 2, ..., 253, 254, 255])
- lon_bounds(bnds, y, x)float64dask.array<chunksize=(1, 220, 256), meta=np.ndarray>
- units :
- degrees_east
Array Chunk Bytes 880.00 kiB 440.00 kiB Shape (2, 220, 256) (1, 220, 256) Dask graph 2 chunks in 15 graph layers Data type float64 numpy.ndarray - lat_bounds(bnds, y, x)float64dask.array<chunksize=(1, 220, 256), meta=np.ndarray>
- units :
- degrees_north
Array Chunk Bytes 880.00 kiB 440.00 kiB Shape (2, 220, 256) (1, 220, 256) Dask graph 2 chunks in 12 graph layers Data type float64 numpy.ndarray - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- areacello(member_id, dcpp_init_year, y, x)float32dask.array<chunksize=(1, 1, 220, 256), meta=np.ndarray>
- cell_methods :
- area: sum
- comment :
- Horizontal area of ocean grid cells
- history :
- 2019-11-04T13:44:11Z altered by CMOR: replaced missing value flag (-9e+33) and corresponding data with standard missing value (1e+20).
- long_name :
- Grid-Cell Area for Ocean Variables
- original_name :
- areacello
- standard_name :
- cell_area
- units :
- m²
- original_key :
- ScenarioMIP.MPI-M.MPI-ESM1-2-LR.ssp370.r1i1p1f1.Ofx.areacello.gn.gs://cmip6/CMIP6/ScenarioMIP/MPI-M/MPI-ESM1-2-LR/ssp370/r1i1p1f1/Ofx/areacello/gn/v20190710/.20190710
- parsed_with :
- xmip/postprocessing/_parse_metric
Array Chunk Bytes 220.00 kiB 220.00 kiB Shape (1, 1, 220, 256) (1, 1, 220, 256) Dask graph 1 chunks in 3 graph layers Data type float32 numpy.ndarray
- tos(member_id, dcpp_init_year, time, y, x)float32dask.array<chunksize=(1, 1, 394, 220, 256), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- comment :
- Temperature of upper boundary of the liquid ocean, including temperatures below sea-ice and floating ice shelves.
- history :
- 2019-11-04T13:44:14Z altered by CMOR: replaced missing value flag (-9e+33) and corresponding data with standard missing value (1e+20).
- long_name :
- Sea Surface Temperature
- original_name :
- tos
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 221.72 MiB 84.65 MiB Shape (1, 1, 1032, 220, 256) (1, 1, 394, 220, 256) Dask graph 3 chunks in 3 graph layers Data type float32 numpy.ndarray
- Conventions :
- CF-1.7 CMIP-6.2
- activity_id :
- ScenarioMIP AerChemMIP
- branch_method :
- standard
- branch_time_in_child :
- 60265.0
- branch_time_in_parent :
- 60265.0
- cmor_version :
- 3.5.0
- contact :
- cmip6-mpi-esm@dkrz.de
- creation_date :
- 2019-11-04T13:44:14Z
- data_specs_version :
- 01.00.30
- experiment :
- gap-filling scenario reaching 7.0 based on SSP3
- experiment_id :
- ssp370
- external_variables :
- areacello
- forcing_index :
- 1
- frequency :
- mon
- further_info_url :
- https://furtherinfo.es-doc.org/CMIP6.MPI-M.MPI-ESM1-2-LR.ssp370.none.r1i1p1f1
- grid :
- gn
- grid_label :
- gn
- history :
- 2019-11-04T13:44:14Z ; CMOR rewrote data to be consistent with CMIP6, CF-1.7 CMIP-6.2 and CF standards.
- initialization_index :
- 1
- institution :
- Max Planck Institute for Meteorology, Hamburg 20146, Germany
- institution_id :
- MPI-M
- license :
- CMIP6 model data produced by MPI-M is licensed under a Creative Commons Attribution ShareAlike 4.0 International License (https://creativecommons.org/licenses). Consult https://pcmdi.llnl.gov/CMIP6/TermsOfUse for terms of use governing CMIP6 output, including citation requirements and proper acknowledgment. Further information about this data, including some limitations, can be found via the further_info_url (recorded as a global attribute in this file) and. The data producers and data providers make no warranty, either express or implied, including, but not limited to, warranties of merchantability and fitness for a particular purpose. All liabilities arising from the supply of the information (including any liability arising in negligence) are excluded to the fullest extent permitted by law.
- mip_era :
- CMIP6
- nominal_resolution :
- 250 km
- parent_activity_id :
- CMIP
- parent_experiment_id :
- historical
- parent_mip_era :
- CMIP6
- parent_source_id :
- MPI-ESM1-2-LR
- parent_time_units :
- days since 1850-1-1 00:00:00
- parent_variant_label :
- r1i1p1f1
- physics_index :
- 1
- product :
- model-output
- project_id :
- CMIP6
- realization_index :
- 1
- realm :
- ocean
- references :
- MPI-ESM: Mauritsen, T. et al. (2019), Developments in the MPI‐M Earth System Model version 1.2 (MPI‐ESM1.2) and Its Response to Increasing CO2, J. Adv. Model. Earth Syst.,11, 998-1038, doi:10.1029/2018MS001400, Mueller, W.A. et al. (2018): A high‐resolution version of the Max Planck Institute Earth System Model MPI‐ESM1.2‐HR. J. Adv. Model. EarthSyst.,10,1383–1413, doi:10.1029/2017MS001217
- source :
- MPI-ESM1.2-LR (2017): aerosol: none, prescribed MACv2-SP atmos: ECHAM6.3 (spectral T63; 192 x 96 longitude/latitude; 47 levels; top level 0.01 hPa) atmosChem: none land: JSBACH3.20 landIce: none/prescribed ocean: MPIOM1.63 (bipolar GR1.5, approximately 1.5deg; 256 x 220 longitude/latitude; 40 levels; top grid cell 0-12 m) ocnBgchem: HAMOCC6 seaIce: unnamed (thermodynamic (Semtner zero-layer) dynamic (Hibler 79) sea ice model)
- source_id :
- MPI-ESM1-2-LR
- source_type :
- AOGCM
- status :
- 2020-05-12;created; by gcs.cmip6.ldeo@gmail.com
- sub_experiment :
- none
- sub_experiment_id :
- none
- table_id :
- Omon
- table_info :
- Creation Date:(09 May 2019) MD5:e6ef8ececc8f338646ebfb3aeed36bfc
- title :
- MPI-ESM1-2-LR output prepared for CMIP6
- tracking_id :
- hdl:21.14100/8040d8f2-6bc4-407f-9d31-2930601dea1d hdl:21.14100/e1cbace4-2290-4adb-943d-e8997a25546b hdl:21.14100/5e2c1f30-1446-4a49-b27c-4112cc9e7c95 hdl:21.14100/652666ac-1d93-4102-8a04-bea4c34696c6 hdl:21.14100/71f92ba2-15b4-4fc7-bc07-37d3f4d0a58e
- variable_id :
- tos
- variant_label :
- r1i1p1f1
- netcdf_tracking_ids :
- hdl:21.14100/8040d8f2-6bc4-407f-9d31-2930601dea1d hdl:21.14100/e1cbace4-2290-4adb-943d-e8997a25546b hdl:21.14100/5e2c1f30-1446-4a49-b27c-4112cc9e7c95 hdl:21.14100/652666ac-1d93-4102-8a04-bea4c34696c6 hdl:21.14100/71f92ba2-15b4-4fc7-bc07-37d3f4d0a58e
- version_id :
- v20190710
- intake_esm_vars :
- ['tos']
- intake_esm_attrs:activity_id :
- ScenarioMIP
- intake_esm_attrs:institution_id :
- MPI-M
- intake_esm_attrs:source_id :
- MPI-ESM1-2-LR
- intake_esm_attrs:experiment_id :
- ssp370
- intake_esm_attrs:member_id :
- r1i1p1f1
- intake_esm_attrs:table_id :
- Omon
- intake_esm_attrs:variable_id :
- tos
- intake_esm_attrs:grid_label :
- gn
- intake_esm_attrs:zstore :
- gs://cmip6/CMIP6/ScenarioMIP/MPI-M/MPI-ESM1-2-LR/ssp370/r1i1p1f1/Omon/tos/gn/v20190710/
- intake_esm_attrs:version :
- 20190710
- intake_esm_attrs:_data_format_ :
- zarr
- intake_esm_dataset_key :
- ScenarioMIP.MPI-M.MPI-ESM1-2-LR.ssp370.r1i1p1f1.Omon.tos.gn.gs://cmip6/CMIP6/ScenarioMIP/MPI-M/MPI-ESM1-2-LR/ssp370/r1i1p1f1/Omon/tos/gn/v20190710/.20190710
<xarray.DatasetView> Dimensions: (member_id: 1, dcpp_init_year: 1, time: 1032, y: 220, x: 256, vertex: 4, bnds: 2) Coordinates: (12/13) lat (y, x) float64 dask.array<chunksize=(220, 256), meta=np.ndarray> lon (y, x) float64 dask.array<chunksize=(220, 256), meta=np.ndarray> * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 lat_verticies (y, x, vertex) float64 dask.array<chunksize=(220, 256, 4), meta=np.ndarray> lon_verticies (y, x, vertex) float64 dask.array<chunksize=(220, 256, 4), meta=np.ndarray> time_bounds (time, bnds) object dask.array<chunksize=(1032, 2), meta=np.ndarray> ... ... * x (x) int64 0 1 2 3 4 5 6 7 ... 249 250 251 252 253 254 255 lon_bounds (bnds, y, x) float64 dask.array<chunksize=(1, 220, 256), meta=np.ndarray> lat_bounds (bnds, y, x) float64 dask.array<chunksize=(1, 220, 256), meta=np.ndarray> * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan areacello (member_id, dcpp_init_year, y, x) float32 dask.array<chunksize=(1, 1, 220, 256), meta=np.ndarray> Dimensions without coordinates: vertex, bnds Data variables: tos (member_id, dcpp_init_year, time, y, x) float32 dask.array<chunksize=(1, 1, 394, 220, 256), meta=np.ndarray> Attributes: (12/63) Conventions: CF-1.7 CMIP-6.2 activity_id: ScenarioMIP AerChemMIP branch_method: standard branch_time_in_child: 60265.0 branch_time_in_parent: 60265.0 cmor_version: 3.5.0 ... ... intake_esm_attrs:variable_id: tos intake_esm_attrs:grid_label: gn intake_esm_attrs:zstore: gs://cmip6/CMIP6/ScenarioMIP/MPI-M/MPI-... intake_esm_attrs:version: 20190710 intake_esm_attrs:_data_format_: zarr intake_esm_dataset_key: ScenarioMIP.MPI-M.MPI-ESM1-2-LR.ssp370....ssp370- member_id: 1
- dcpp_init_year: 1
- time: 1032
- y: 220
- x: 256
- vertex: 4
- bnds: 2
- lat(y, x)float64dask.array<chunksize=(220, 256), meta=np.ndarray>
- bounds :
- vertices_latitude
- long_name :
- latitude
- standard_name :
- latitude
- units :
- degrees_north
Array Chunk Bytes 440.00 kiB 440.00 kiB Shape (220, 256) (220, 256) Dask graph 1 chunks in 5 graph layers Data type float64 numpy.ndarray - lon(y, x)float64dask.array<chunksize=(220, 256), meta=np.ndarray>
- bounds :
- vertices_longitude
- long_name :
- longitude
- standard_name :
- longitude
- units :
- degrees_east
Array Chunk Bytes 440.00 kiB 440.00 kiB Shape (220, 256) (220, 256) Dask graph 1 chunks in 8 graph layers Data type float64 numpy.ndarray - time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bnds
- long_name :
- time
- standard_name :
- time
array([cftime.DatetimeProlepticGregorian(2015, 1, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2015, 2, 15, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2015, 3, 16, 12, 0, 0, 0, has_year_zero=True), ..., cftime.DatetimeProlepticGregorian(2100, 10, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2100, 11, 16, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2100, 12, 16, 12, 0, 0, 0, has_year_zero=True)], dtype=object) - lat_verticies(y, x, vertex)float64dask.array<chunksize=(220, 256, 4), meta=np.ndarray>
- units :
- degrees_north
Array Chunk Bytes 1.72 MiB 1.72 MiB Shape (220, 256, 4) (220, 256, 4) Dask graph 1 chunks in 3 graph layers Data type float64 numpy.ndarray - lon_verticies(y, x, vertex)float64dask.array<chunksize=(220, 256, 4), meta=np.ndarray>
- units :
- degrees_east
Array Chunk Bytes 1.72 MiB 1.72 MiB Shape (220, 256, 4) (220, 256, 4) Dask graph 1 chunks in 6 graph layers Data type float64 numpy.ndarray - time_bounds(time, bnds)objectdask.array<chunksize=(1032, 2), meta=np.ndarray>
Array Chunk Bytes 16.12 kiB 16.12 kiB Shape (1032, 2) (1032, 2) Dask graph 1 chunks in 2 graph layers Data type object numpy.ndarray - y(y)int640 1 2 3 4 5 ... 215 216 217 218 219
array([ 0, 1, 2, ..., 217, 218, 219])
- x(x)int640 1 2 3 4 5 ... 251 252 253 254 255
array([ 0, 1, 2, ..., 253, 254, 255])
- lon_bounds(bnds, y, x)float64dask.array<chunksize=(1, 220, 256), meta=np.ndarray>
- units :
- degrees_east
Array Chunk Bytes 880.00 kiB 440.00 kiB Shape (2, 220, 256) (1, 220, 256) Dask graph 2 chunks in 15 graph layers Data type float64 numpy.ndarray - lat_bounds(bnds, y, x)float64dask.array<chunksize=(1, 220, 256), meta=np.ndarray>
- units :
- degrees_north
Array Chunk Bytes 880.00 kiB 440.00 kiB Shape (2, 220, 256) (1, 220, 256) Dask graph 2 chunks in 12 graph layers Data type float64 numpy.ndarray - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- areacello(member_id, dcpp_init_year, y, x)float32dask.array<chunksize=(1, 1, 220, 256), meta=np.ndarray>
- cell_methods :
- area: sum
- comment :
- Horizontal area of ocean grid cells
- history :
- 2019-11-11T15:58:25Z altered by CMOR: replaced missing value flag (-9e+33) and corresponding data with standard missing value (1e+20).
- long_name :
- Grid-Cell Area for Ocean Variables
- original_name :
- areacello
- standard_name :
- cell_area
- units :
- m²
- original_key :
- ScenarioMIP.MPI-M.MPI-ESM1-2-LR.ssp585.r1i1p1f1.Ofx.areacello.gn.gs://cmip6/CMIP6/ScenarioMIP/MPI-M/MPI-ESM1-2-LR/ssp585/r1i1p1f1/Ofx/areacello/gn/v20190710/.20190710
- parsed_with :
- xmip/postprocessing/_parse_metric
Array Chunk Bytes 220.00 kiB 220.00 kiB Shape (1, 1, 220, 256) (1, 1, 220, 256) Dask graph 1 chunks in 3 graph layers Data type float32 numpy.ndarray
- tos(member_id, dcpp_init_year, time, y, x)float32dask.array<chunksize=(1, 1, 394, 220, 256), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- comment :
- Temperature of upper boundary of the liquid ocean, including temperatures below sea-ice and floating ice shelves.
- long_name :
- Sea Surface Temperature
- original_name :
- tos
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 221.72 MiB 84.65 MiB Shape (1, 1, 1032, 220, 256) (1, 1, 394, 220, 256) Dask graph 3 chunks in 3 graph layers Data type float32 numpy.ndarray
- Conventions :
- CF-1.7 CMIP-6.2
- activity_id :
- ScenarioMIP
- branch_method :
- standard
- branch_time_in_child :
- 60265.0
- branch_time_in_parent :
- 60265.0
- cmor_version :
- 3.5.0
- contact :
- cmip6-mpi-esm@dkrz.de
- creation_date :
- 2019-11-11T15:58:26Z
- data_specs_version :
- 01.00.30
- experiment :
- update of RCP8.5 based on SSP5
- experiment_id :
- ssp585
- external_variables :
- areacello
- forcing_index :
- 1
- frequency :
- mon
- further_info_url :
- https://furtherinfo.es-doc.org/CMIP6.MPI-M.MPI-ESM1-2-LR.ssp585.none.r1i1p1f1
- grid :
- gn
- grid_label :
- gn
- history :
- 2019-11-11T15:58:26Z ; CMOR rewrote data to be consistent with CMIP6, CF-1.7 CMIP-6.2 and CF standards.
- initialization_index :
- 1
- institution :
- Max Planck Institute for Meteorology, Hamburg 20146, Germany
- institution_id :
- MPI-M
- license :
- CMIP6 model data produced by MPI-M is licensed under a Creative Commons Attribution ShareAlike 4.0 International License (https://creativecommons.org/licenses). Consult https://pcmdi.llnl.gov/CMIP6/TermsOfUse for terms of use governing CMIP6 output, including citation requirements and proper acknowledgment. Further information about this data, including some limitations, can be found via the further_info_url (recorded as a global attribute in this file) and. The data producers and data providers make no warranty, either express or implied, including, but not limited to, warranties of merchantability and fitness for a particular purpose. All liabilities arising from the supply of the information (including any liability arising in negligence) are excluded to the fullest extent permitted by law.
- mip_era :
- CMIP6
- nominal_resolution :
- 250 km
- parent_activity_id :
- CMIP
- parent_experiment_id :
- historical
- parent_mip_era :
- CMIP6
- parent_source_id :
- MPI-ESM1-2-LR
- parent_time_units :
- days since 1850-1-1 00:00:00
- parent_variant_label :
- r1i1p1f1
- physics_index :
- 1
- product :
- model-output
- project_id :
- CMIP6
- realization_index :
- 1
- realm :
- ocean
- references :
- MPI-ESM: Mauritsen, T. et al. (2019), Developments in the MPI‐M Earth System Model version 1.2 (MPI‐ESM1.2) and Its Response to Increasing CO2, J. Adv. Model. Earth Syst.,11, 998-1038, doi:10.1029/2018MS001400, Mueller, W.A. et al. (2018): A high‐resolution version of the Max Planck Institute Earth System Model MPI‐ESM1.2‐HR. J. Adv. Model. EarthSyst.,10,1383–1413, doi:10.1029/2017MS001217
- source :
- MPI-ESM1.2-LR (2017): aerosol: none, prescribed MACv2-SP atmos: ECHAM6.3 (spectral T63; 192 x 96 longitude/latitude; 47 levels; top level 0.01 hPa) atmosChem: none land: JSBACH3.20 landIce: none/prescribed ocean: MPIOM1.63 (bipolar GR1.5, approximately 1.5deg; 256 x 220 longitude/latitude; 40 levels; top grid cell 0-12 m) ocnBgchem: HAMOCC6 seaIce: unnamed (thermodynamic (Semtner zero-layer) dynamic (Hibler 79) sea ice model)
- source_id :
- MPI-ESM1-2-LR
- source_type :
- AOGCM
- status :
- 2020-03-21;created; by gcs.cmip6.ldeo@gmail.com
- sub_experiment :
- none
- sub_experiment_id :
- none
- table_id :
- Omon
- table_info :
- Creation Date:(09 May 2019) MD5:e6ef8ececc8f338646ebfb3aeed36bfc
- title :
- MPI-ESM1-2-LR output prepared for CMIP6
- tracking_id :
- hdl:21.14100/e9fc6810-e058-4ff3-91ff-e98783e10e6b hdl:21.14100/23b00fee-f847-45c7-9505-e14671fac95e hdl:21.14100/9acf643c-7d91-4922-9922-4ce59bd72b8a hdl:21.14100/3bd4924b-c201-454f-8dfb-48c70183198d hdl:21.14100/182b6a5a-c535-4d4e-b9ce-65f86c291af5
- variable_id :
- tos
- variant_label :
- r1i1p1f1
- netcdf_tracking_ids :
- hdl:21.14100/e9fc6810-e058-4ff3-91ff-e98783e10e6b hdl:21.14100/23b00fee-f847-45c7-9505-e14671fac95e hdl:21.14100/9acf643c-7d91-4922-9922-4ce59bd72b8a hdl:21.14100/3bd4924b-c201-454f-8dfb-48c70183198d hdl:21.14100/182b6a5a-c535-4d4e-b9ce-65f86c291af5
- version_id :
- v20190710
- intake_esm_vars :
- ['tos']
- intake_esm_attrs:activity_id :
- ScenarioMIP
- intake_esm_attrs:institution_id :
- MPI-M
- intake_esm_attrs:source_id :
- MPI-ESM1-2-LR
- intake_esm_attrs:experiment_id :
- ssp585
- intake_esm_attrs:member_id :
- r1i1p1f1
- intake_esm_attrs:table_id :
- Omon
- intake_esm_attrs:variable_id :
- tos
- intake_esm_attrs:grid_label :
- gn
- intake_esm_attrs:zstore :
- gs://cmip6/CMIP6/ScenarioMIP/MPI-M/MPI-ESM1-2-LR/ssp585/r1i1p1f1/Omon/tos/gn/v20190710/
- intake_esm_attrs:version :
- 20190710
- intake_esm_attrs:_data_format_ :
- zarr
- intake_esm_dataset_key :
- ScenarioMIP.MPI-M.MPI-ESM1-2-LR.ssp585.r1i1p1f1.Omon.tos.gn.gs://cmip6/CMIP6/ScenarioMIP/MPI-M/MPI-ESM1-2-LR/ssp585/r1i1p1f1/Omon/tos/gn/v20190710/.20190710
<xarray.DatasetView> Dimensions: (member_id: 1, dcpp_init_year: 1, time: 1032, y: 220, x: 256, vertex: 4, bnds: 2) Coordinates: (12/13) lat (y, x) float64 dask.array<chunksize=(220, 256), meta=np.ndarray> lon (y, x) float64 dask.array<chunksize=(220, 256), meta=np.ndarray> * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 lat_verticies (y, x, vertex) float64 dask.array<chunksize=(220, 256, 4), meta=np.ndarray> lon_verticies (y, x, vertex) float64 dask.array<chunksize=(220, 256, 4), meta=np.ndarray> time_bounds (time, bnds) object dask.array<chunksize=(1032, 2), meta=np.ndarray> ... ... * x (x) int64 0 1 2 3 4 5 6 7 ... 249 250 251 252 253 254 255 lon_bounds (bnds, y, x) float64 dask.array<chunksize=(1, 220, 256), meta=np.ndarray> lat_bounds (bnds, y, x) float64 dask.array<chunksize=(1, 220, 256), meta=np.ndarray> * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan areacello (member_id, dcpp_init_year, y, x) float32 dask.array<chunksize=(1, 1, 220, 256), meta=np.ndarray> Dimensions without coordinates: vertex, bnds Data variables: tos (member_id, dcpp_init_year, time, y, x) float32 dask.array<chunksize=(1, 1, 394, 220, 256), meta=np.ndarray> Attributes: (12/63) Conventions: CF-1.7 CMIP-6.2 activity_id: ScenarioMIP branch_method: standard branch_time_in_child: 60265.0 branch_time_in_parent: 60265.0 cmor_version: 3.5.0 ... ... intake_esm_attrs:variable_id: tos intake_esm_attrs:grid_label: gn intake_esm_attrs:zstore: gs://cmip6/CMIP6/ScenarioMIP/MPI-M/MPI-... intake_esm_attrs:version: 20190710 intake_esm_attrs:_data_format_: zarr intake_esm_dataset_key: ScenarioMIP.MPI-M.MPI-ESM1-2-LR.ssp585....ssp585- member_id: 1
- dcpp_init_year: 1
- time: 1980
- y: 220
- x: 256
- vertex: 4
- bnds: 2
- lat(y, x)float64dask.array<chunksize=(220, 256), meta=np.ndarray>
- bounds :
- vertices_latitude
- long_name :
- latitude
- standard_name :
- latitude
- units :
- degrees_north
Array Chunk Bytes 440.00 kiB 440.00 kiB Shape (220, 256) (220, 256) Dask graph 1 chunks in 5 graph layers Data type float64 numpy.ndarray - lon(y, x)float64dask.array<chunksize=(220, 256), meta=np.ndarray>
- bounds :
- vertices_longitude
- long_name :
- longitude
- standard_name :
- longitude
- units :
- degrees_east
Array Chunk Bytes 440.00 kiB 440.00 kiB Shape (220, 256) (220, 256) Dask graph 1 chunks in 8 graph layers Data type float64 numpy.ndarray - time(time)object1850-01-16 12:00:00 ... 2014-12-...
- axis :
- T
- bounds :
- time_bnds
- long_name :
- time
- standard_name :
- time
array([cftime.DatetimeProlepticGregorian(1850, 1, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(1850, 2, 15, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(1850, 3, 16, 12, 0, 0, 0, has_year_zero=True), ..., cftime.DatetimeProlepticGregorian(2014, 10, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2014, 11, 16, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2014, 12, 16, 12, 0, 0, 0, has_year_zero=True)], dtype=object) - lat_verticies(y, x, vertex)float64dask.array<chunksize=(220, 256, 4), meta=np.ndarray>
- units :
- degrees_north
Array Chunk Bytes 1.72 MiB 1.72 MiB Shape (220, 256, 4) (220, 256, 4) Dask graph 1 chunks in 3 graph layers Data type float64 numpy.ndarray - lon_verticies(y, x, vertex)float64dask.array<chunksize=(220, 256, 4), meta=np.ndarray>
- units :
- degrees_east
Array Chunk Bytes 1.72 MiB 1.72 MiB Shape (220, 256, 4) (220, 256, 4) Dask graph 1 chunks in 6 graph layers Data type float64 numpy.ndarray - time_bounds(time, bnds)objectdask.array<chunksize=(1980, 2), meta=np.ndarray>
Array Chunk Bytes 30.94 kiB 30.94 kiB Shape (1980, 2) (1980, 2) Dask graph 1 chunks in 2 graph layers Data type object numpy.ndarray - y(y)int640 1 2 3 4 5 ... 215 216 217 218 219
array([ 0, 1, 2, ..., 217, 218, 219])
- x(x)int640 1 2 3 4 5 ... 251 252 253 254 255
array([ 0, 1, 2, ..., 253, 254, 255])
- lon_bounds(bnds, y, x)float64dask.array<chunksize=(1, 220, 256), meta=np.ndarray>
- units :
- degrees_east
Array Chunk Bytes 880.00 kiB 440.00 kiB Shape (2, 220, 256) (1, 220, 256) Dask graph 2 chunks in 15 graph layers Data type float64 numpy.ndarray - lat_bounds(bnds, y, x)float64dask.array<chunksize=(1, 220, 256), meta=np.ndarray>
- units :
- degrees_north
Array Chunk Bytes 880.00 kiB 440.00 kiB Shape (2, 220, 256) (1, 220, 256) Dask graph 2 chunks in 12 graph layers Data type float64 numpy.ndarray - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- areacello(member_id, dcpp_init_year, y, x)float32dask.array<chunksize=(1, 1, 220, 256), meta=np.ndarray>
- cell_methods :
- area: sum
- comment :
- Horizontal area of ocean grid cells
- history :
- 2019-09-11T14:21:40Z altered by CMOR: replaced missing value flag (-9e+33) and corresponding data with standard missing value (1e+20).
- long_name :
- Grid-Cell Area for Ocean Variables
- original_name :
- areacello
- standard_name :
- cell_area
- units :
- m²
- original_key :
- CMIP.MPI-M.MPI-ESM1-2-LR.historical.r1i1p1f1.Ofx.areacello.gn.gs://cmip6/CMIP6/CMIP/MPI-M/MPI-ESM1-2-LR/historical/r1i1p1f1/Ofx/areacello/gn/v20190710/.20190710
- parsed_with :
- xmip/postprocessing/_parse_metric
Array Chunk Bytes 220.00 kiB 220.00 kiB Shape (1, 1, 220, 256) (1, 1, 220, 256) Dask graph 1 chunks in 3 graph layers Data type float32 numpy.ndarray
- tos(member_id, dcpp_init_year, time, y, x)float32dask.array<chunksize=(1, 1, 397, 220, 256), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- comment :
- Temperature of upper boundary of the liquid ocean, including temperatures below sea-ice and floating ice shelves.
- history :
- 2019-09-11T14:21:40Z altered by CMOR: replaced missing value flag (-9e+33) and corresponding data with standard missing value (1e+20).
- long_name :
- Sea Surface Temperature
- original_name :
- tos
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 425.39 MiB 85.29 MiB Shape (1, 1, 1980, 220, 256) (1, 1, 397, 220, 256) Dask graph 5 chunks in 3 graph layers Data type float32 numpy.ndarray
- Conventions :
- CF-1.7 CMIP-6.2
- activity_id :
- CMIP
- branch_method :
- standard
- branch_time_in_child :
- 0.0
- branch_time_in_parent :
- 0.0
- cmor_version :
- 3.5.0
- contact :
- cmip6-mpi-esm@dkrz.de
- creation_date :
- 2019-09-11T14:21:40Z
- data_specs_version :
- 01.00.30
- experiment :
- all-forcing simulation of the recent past
- experiment_id :
- historical
- external_variables :
- areacello
- forcing_index :
- 1
- frequency :
- mon
- further_info_url :
- https://furtherinfo.es-doc.org/CMIP6.MPI-M.MPI-ESM1-2-LR.historical.none.r1i1p1f1
- grid :
- gn
- grid_label :
- gn
- history :
- 2019-09-11T14:21:40Z ; CMOR rewrote data to be consistent with CMIP6, CF-1.7 CMIP-6.2 and CF standards.
- initialization_index :
- 1
- institution :
- Max Planck Institute for Meteorology, Hamburg 20146, Germany
- institution_id :
- MPI-M
- license :
- CMIP6 model data produced by MPI-M is licensed under a Creative Commons Attribution ShareAlike 4.0 International License (https://creativecommons.org/licenses). Consult https://pcmdi.llnl.gov/CMIP6/TermsOfUse for terms of use governing CMIP6 output, including citation requirements and proper acknowledgment. Further information about this data, including some limitations, can be found via the further_info_url (recorded as a global attribute in this file) and. The data producers and data providers make no warranty, either express or implied, including, but not limited to, warranties of merchantability and fitness for a particular purpose. All liabilities arising from the supply of the information (including any liability arising in negligence) are excluded to the fullest extent permitted by law.
- mip_era :
- CMIP6
- nominal_resolution :
- 250 km
- parent_activity_id :
- CMIP
- parent_experiment_id :
- piControl
- parent_mip_era :
- CMIP6
- parent_source_id :
- MPI-ESM1-2-LR
- parent_time_units :
- days since 1850-1-1 00:00:00
- parent_variant_label :
- r1i1p1f1
- physics_index :
- 1
- product :
- model-output
- project_id :
- CMIP6
- realization_index :
- 1
- realm :
- ocean
- references :
- MPI-ESM: Mauritsen, T. et al. (2019), Developments in the MPI‐M Earth System Model version 1.2 (MPI‐ESM1.2) and Its Response to Increasing CO2, J. Adv. Model. Earth Syst.,11, 998-1038, doi:10.1029/2018MS001400, Mueller, W.A. et al. (2018): A high‐resolution version of the Max Planck Institute Earth System Model MPI‐ESM1.2‐HR. J. Adv. Model. EarthSyst.,10,1383–1413, doi:10.1029/2017MS001217
- source :
- MPI-ESM1.2-LR (2017): aerosol: none, prescribed MACv2-SP atmos: ECHAM6.3 (spectral T63; 192 x 96 longitude/latitude; 47 levels; top level 0.01 hPa) atmosChem: none land: JSBACH3.20 landIce: none/prescribed ocean: MPIOM1.63 (bipolar GR1.5, approximately 1.5deg; 256 x 220 longitude/latitude; 40 levels; top grid cell 0-12 m) ocnBgchem: HAMOCC6 seaIce: unnamed (thermodynamic (Semtner zero-layer) dynamic (Hibler 79) sea ice model)
- source_id :
- MPI-ESM1-2-LR
- source_type :
- AOGCM
- status :
- 2020-05-09;created; by gcs.cmip6.ldeo@gmail.com
- sub_experiment :
- none
- sub_experiment_id :
- none
- table_id :
- Omon
- table_info :
- Creation Date:(09 May 2019) MD5:e6ef8ececc8f338646ebfb3aeed36bfc
- title :
- MPI-ESM1-2-LR output prepared for CMIP6
- tracking_id :
- hdl:21.14100/40326101-1a73-46bb-a1d8-edb6f6a4da7c hdl:21.14100/26ca0da6-9d9c-4ae1-9369-ea76f3fa2f29 hdl:21.14100/ef01818e-7926-48da-a1c2-9b227d5eb72a hdl:21.14100/c81e4f2a-7891-4b37-826e-1c4d5ae72c78 hdl:21.14100/bd7cd450-90c9-4448-b801-0fa880cf35e6 hdl:21.14100/ab1b5214-c015-4c91-98cd-409d0f95bbf2 hdl:21.14100/8e3f623d-41f3-4578-8d58-37b982950db8 hdl:21.14100/1bab583c-849a-43e3-9d61-f3b8b5758650 hdl:21.14100/b11b872a-7ab3-4ca1-8ec7-1f6936b5f354
- variable_id :
- tos
- variant_label :
- r1i1p1f1
- netcdf_tracking_ids :
- hdl:21.14100/40326101-1a73-46bb-a1d8-edb6f6a4da7c hdl:21.14100/26ca0da6-9d9c-4ae1-9369-ea76f3fa2f29 hdl:21.14100/ef01818e-7926-48da-a1c2-9b227d5eb72a hdl:21.14100/c81e4f2a-7891-4b37-826e-1c4d5ae72c78 hdl:21.14100/bd7cd450-90c9-4448-b801-0fa880cf35e6 hdl:21.14100/ab1b5214-c015-4c91-98cd-409d0f95bbf2 hdl:21.14100/8e3f623d-41f3-4578-8d58-37b982950db8 hdl:21.14100/1bab583c-849a-43e3-9d61-f3b8b5758650 hdl:21.14100/b11b872a-7ab3-4ca1-8ec7-1f6936b5f354
- version_id :
- v20190710
- intake_esm_vars :
- ['tos']
- intake_esm_attrs:activity_id :
- CMIP
- intake_esm_attrs:institution_id :
- MPI-M
- intake_esm_attrs:source_id :
- MPI-ESM1-2-LR
- intake_esm_attrs:experiment_id :
- historical
- intake_esm_attrs:member_id :
- r1i1p1f1
- intake_esm_attrs:table_id :
- Omon
- intake_esm_attrs:variable_id :
- tos
- intake_esm_attrs:grid_label :
- gn
- intake_esm_attrs:zstore :
- gs://cmip6/CMIP6/CMIP/MPI-M/MPI-ESM1-2-LR/historical/r1i1p1f1/Omon/tos/gn/v20190710/
- intake_esm_attrs:version :
- 20190710
- intake_esm_attrs:_data_format_ :
- zarr
- intake_esm_dataset_key :
- CMIP.MPI-M.MPI-ESM1-2-LR.historical.r1i1p1f1.Omon.tos.gn.gs://cmip6/CMIP6/CMIP/MPI-M/MPI-ESM1-2-LR/historical/r1i1p1f1/Omon/tos/gn/v20190710/.20190710
<xarray.DatasetView> Dimensions: (member_id: 1, dcpp_init_year: 1, time: 1980, y: 220, x: 256, vertex: 4, bnds: 2) Coordinates: (12/13) lat (y, x) float64 dask.array<chunksize=(220, 256), meta=np.ndarray> lon (y, x) float64 dask.array<chunksize=(220, 256), meta=np.ndarray> * time (time) object 1850-01-16 12:00:00 ... 2014-12-16 12:00:00 lat_verticies (y, x, vertex) float64 dask.array<chunksize=(220, 256, 4), meta=np.ndarray> lon_verticies (y, x, vertex) float64 dask.array<chunksize=(220, 256, 4), meta=np.ndarray> time_bounds (time, bnds) object dask.array<chunksize=(1980, 2), meta=np.ndarray> ... ... * x (x) int64 0 1 2 3 4 5 6 7 ... 249 250 251 252 253 254 255 lon_bounds (bnds, y, x) float64 dask.array<chunksize=(1, 220, 256), meta=np.ndarray> lat_bounds (bnds, y, x) float64 dask.array<chunksize=(1, 220, 256), meta=np.ndarray> * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan areacello (member_id, dcpp_init_year, y, x) float32 dask.array<chunksize=(1, 1, 220, 256), meta=np.ndarray> Dimensions without coordinates: vertex, bnds Data variables: tos (member_id, dcpp_init_year, time, y, x) float32 dask.array<chunksize=(1, 1, 397, 220, 256), meta=np.ndarray> Attributes: (12/63) Conventions: CF-1.7 CMIP-6.2 activity_id: CMIP branch_method: standard branch_time_in_child: 0.0 branch_time_in_parent: 0.0 cmor_version: 3.5.0 ... ... intake_esm_attrs:variable_id: tos intake_esm_attrs:grid_label: gn intake_esm_attrs:zstore: gs://cmip6/CMIP6/CMIP/MPI-M/MPI-ESM1-2-... intake_esm_attrs:version: 20190710 intake_esm_attrs:_data_format_: zarr intake_esm_dataset_key: CMIP.MPI-M.MPI-ESM1-2-LR.historical.r1i...historical
<xarray.DatasetView> Dimensions: () Data variables: *empty*MPI-ESM1-2-LR- member_id: 1
- dcpp_init_year: 1
- time: 1032
- x: 292
- y: 362
- vertex: 4
- bnds: 2
- lat(x, y)float64dask.array<chunksize=(292, 362), meta=np.ndarray>
- bounds :
- vertices_latitude
- long_name :
- latitude
- standard_name :
- latitude
- units :
- degrees_north
Array Chunk Bytes 825.81 kiB 825.81 kiB Shape (292, 362) (292, 362) Dask graph 1 chunks in 5 graph layers Data type float64 numpy.ndarray - lon(x, y)float64dask.array<chunksize=(292, 362), meta=np.ndarray>
- bounds :
- vertices_longitude
- long_name :
- longitude
- standard_name :
- longitude
- units :
- degrees_east
Array Chunk Bytes 825.81 kiB 825.81 kiB Shape (292, 362) (292, 362) Dask graph 1 chunks in 8 graph layers Data type float64 numpy.ndarray - time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bnds
- long_name :
- time
- standard_name :
- time
array([cftime.DatetimeNoLeap(2015, 1, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2015, 2, 15, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2015, 3, 16, 12, 0, 0, 0, has_year_zero=True), ..., cftime.DatetimeNoLeap(2100, 10, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2100, 11, 16, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2100, 12, 16, 12, 0, 0, 0, has_year_zero=True)], dtype=object) - lat_verticies(x, y, vertex)float64dask.array<chunksize=(292, 362, 4), meta=np.ndarray>
- units :
- degrees_north
Array Chunk Bytes 3.23 MiB 3.23 MiB Shape (292, 362, 4) (292, 362, 4) Dask graph 1 chunks in 3 graph layers Data type float64 numpy.ndarray - lon_verticies(x, y, vertex)float64dask.array<chunksize=(292, 362, 4), meta=np.ndarray>
- units :
- degrees_east
Array Chunk Bytes 3.23 MiB 3.23 MiB Shape (292, 362, 4) (292, 362, 4) Dask graph 1 chunks in 6 graph layers Data type float64 numpy.ndarray - time_bounds(time, bnds)objectdask.array<chunksize=(1032, 2), meta=np.ndarray>
Array Chunk Bytes 16.12 kiB 16.12 kiB Shape (1032, 2) (1032, 2) Dask graph 1 chunks in 2 graph layers Data type object numpy.ndarray - x(x)int640 1 2 3 4 5 ... 287 288 289 290 291
array([ 0, 1, 2, ..., 289, 290, 291])
- y(y)int640 1 2 3 4 5 ... 357 358 359 360 361
array([ 0, 1, 2, ..., 359, 360, 361])
- lon_bounds(bnds, x, y)float64dask.array<chunksize=(1, 292, 362), meta=np.ndarray>
- units :
- degrees_east
Array Chunk Bytes 1.61 MiB 825.81 kiB Shape (2, 292, 362) (1, 292, 362) Dask graph 2 chunks in 15 graph layers Data type float64 numpy.ndarray - lat_bounds(bnds, x, y)float64dask.array<chunksize=(1, 292, 362), meta=np.ndarray>
- units :
- degrees_north
Array Chunk Bytes 1.61 MiB 825.81 kiB Shape (2, 292, 362) (1, 292, 362) Dask graph 2 chunks in 12 graph layers Data type float64 numpy.ndarray - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- areacello(member_id, dcpp_init_year, x, y)float32dask.array<chunksize=(1, 1, 292, 362), meta=np.ndarray>
- cell_methods :
- area: sum
- comment :
- Horizontal area of ocean grid cells
- history :
- 2021-01-25T13:28:30Z altered by CMOR: Converted type from 'd' to 'f'.
- long_name :
- Grid-Cell Area for Ocean Variables
- original_name :
- e1t, e2t
- standard_name :
- cell_area
- units :
- m²
- original_key :
- ScenarioMIP.CMCC.CMCC-ESM2.ssp126.r1i1p1f1.Ofx.areacello.gn.gs://cmip6/CMIP6/ScenarioMIP/CMCC/CMCC-ESM2/ssp126/r1i1p1f1/Ofx/areacello/gn/v20210126/.20210126
- parsed_with :
- xmip/postprocessing/_parse_metric
Array Chunk Bytes 412.91 kiB 412.91 kiB Shape (1, 1, 292, 362) (1, 1, 292, 362) Dask graph 1 chunks in 3 graph layers Data type float32 numpy.ndarray
- tos(member_id, dcpp_init_year, time, x, y)float32dask.array<chunksize=(1, 1, 253, 292, 362), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- comment :
- Temperature of upper boundary of the liquid ocean, including temperatures below sea-ice and floating ice shelves.
- long_name :
- Sea Surface Temperature
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 416.13 MiB 102.02 MiB Shape (1, 1, 1032, 292, 362) (1, 1, 253, 292, 362) Dask graph 5 chunks in 3 graph layers Data type float32 numpy.ndarray
- Conventions :
- CF-1.7 CMIP-6.2
- activity_id :
- ScenarioMIP
- branch_method :
- standard
- branch_time_in_child :
- 60225.0
- branch_time_in_parent :
- 60225.0
- cmor_version :
- 3.6.0
- comment :
- none
- contact :
- T. Lovato
- creation_date :
- 2021-01-25T13:28:44Z
- data_specs_version :
- 01.00.31
- experiment :
- update of RCP2.6 based on SSP1
- experiment_id :
- ssp126
- external_variables :
- areacello
- forcing_index :
- 1
- frequency :
- mon
- further_info_url :
- https://furtherinfo.es-doc.org/CMIP6.CMCC.CMCC-ESM2.ssp126.none.r1i1p1f1
- grid :
- native ocean curvilinear grid
- grid_label :
- gn
- history :
- 2021-01-25T13:28:44Z ;rewrote data to be consistent with ScenarioMIP for variable tos found in table Omon.; none
- initialization_index :
- 1
- institution :
- Fondazione Centro Euro-Mediterraneo sui Cambiamenti Climatici, Lecce 73100, Italy
- institution_id :
- CMCC
- license :
- CMIP6 model data produced by CMCC is licensed under a Creative Commons Attribution ShareAlike 4.0 International License (https://creativecommons.org/licenses). Consult https://pcmdi.llnl.gov/CMIP6/TermsOfUse for terms of use governing CMIP6 output, including citation requirements and proper acknowledgment. Further information about this data, including some limitations, can be found via the further_info_url (recorded as a global attribute in this file) and at https:///pcmdi.llnl.gov/. The data producers and data providers make no warranty, either express or implied, including, but not limited to, warranties of merchantability and fitness for a particular purpose. All liabilities arising from the supply of the information (including any liability arising in negligence) are excluded to the fullest extent permitted by law.
- mip_era :
- CMIP6
- netcdf_tracking_ids :
- hdl:21.14100/b0e80d51-362b-4bcb-8046-d2ed66a64815
- nominal_resolution :
- 100 km
- parent_activity_id :
- CMIP
- parent_experiment_id :
- historical
- parent_mip_era :
- CMIP6
- parent_source_id :
- CMCC-ESM2
- parent_time_units :
- days since 1850-01-01
- parent_variant_label :
- r1i1p1f1
- physics_index :
- 1
- product :
- model-output
- realization_index :
- 1
- realm :
- ocean
- references :
- none
- run_variant :
- 1st realization
- source :
- CMCC-ESM2 (2017): aerosol: MAM3 atmos: CAM5.3 (1deg; 288 x 192 longitude/latitude; 30 levels; top at ~2 hPa) atmosChem: none land: CLM4.5 (BGC mode) landIce: none ocean: NEMO3.6 (ORCA1 tripolar primarly 1 deg lat/lon with meridional refinement down to 1/3 degree in the tropics; 362 x 292 longitude/latitude; 50 vertical levels; top grid cell 0-1 m) ocnBgchem: BFM5.1 seaIce: CICE4.0
- source_id :
- CMCC-ESM2
- source_type :
- AOGCM BGC
- status :
- 2021-04-07;created; by gcs.cmip6.ldeo@gmail.com
- sub_experiment :
- none
- sub_experiment_id :
- none
- table_id :
- Omon
- table_info :
- Creation Date:(05 February 2020) MD5:6a248fd76c55aa6d6f7b3cc6866b5faf
- title :
- CMCC-ESM2 output prepared for CMIP6
- tracking_id :
- hdl:21.14100/b0e80d51-362b-4bcb-8046-d2ed66a64815
- variable_id :
- tos
- variant_label :
- r1i1p1f1
- version_id :
- v20210126
- intake_esm_vars :
- ['tos']
- intake_esm_attrs:activity_id :
- ScenarioMIP
- intake_esm_attrs:institution_id :
- CMCC
- intake_esm_attrs:source_id :
- CMCC-ESM2
- intake_esm_attrs:experiment_id :
- ssp126
- intake_esm_attrs:member_id :
- r1i1p1f1
- intake_esm_attrs:table_id :
- Omon
- intake_esm_attrs:variable_id :
- tos
- intake_esm_attrs:grid_label :
- gn
- intake_esm_attrs:zstore :
- gs://cmip6/CMIP6/ScenarioMIP/CMCC/CMCC-ESM2/ssp126/r1i1p1f1/Omon/tos/gn/v20210126/
- intake_esm_attrs:version :
- 20210126
- intake_esm_attrs:_data_format_ :
- zarr
- intake_esm_dataset_key :
- ScenarioMIP.CMCC.CMCC-ESM2.ssp126.r1i1p1f1.Omon.tos.gn.gs://cmip6/CMIP6/ScenarioMIP/CMCC/CMCC-ESM2/ssp126/r1i1p1f1/Omon/tos/gn/v20210126/.20210126
<xarray.DatasetView> Dimensions: (member_id: 1, dcpp_init_year: 1, time: 1032, x: 292, y: 362, vertex: 4, bnds: 2) Coordinates: (12/13) lat (x, y) float64 dask.array<chunksize=(292, 362), meta=np.ndarray> lon (x, y) float64 dask.array<chunksize=(292, 362), meta=np.ndarray> * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 lat_verticies (x, y, vertex) float64 dask.array<chunksize=(292, 362, 4), meta=np.ndarray> lon_verticies (x, y, vertex) float64 dask.array<chunksize=(292, 362, 4), meta=np.ndarray> time_bounds (time, bnds) object dask.array<chunksize=(1032, 2), meta=np.ndarray> ... ... * y (y) int64 0 1 2 3 4 5 6 7 ... 355 356 357 358 359 360 361 lon_bounds (bnds, x, y) float64 dask.array<chunksize=(1, 292, 362), meta=np.ndarray> lat_bounds (bnds, x, y) float64 dask.array<chunksize=(1, 292, 362), meta=np.ndarray> * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan areacello (member_id, dcpp_init_year, x, y) float32 dask.array<chunksize=(1, 1, 292, 362), meta=np.ndarray> Dimensions without coordinates: vertex, bnds Data variables: tos (member_id, dcpp_init_year, time, x, y) float32 dask.array<chunksize=(1, 1, 253, 292, 362), meta=np.ndarray> Attributes: (12/64) Conventions: CF-1.7 CMIP-6.2 activity_id: ScenarioMIP branch_method: standard branch_time_in_child: 60225.0 branch_time_in_parent: 60225.0 cmor_version: 3.6.0 ... ... intake_esm_attrs:variable_id: tos intake_esm_attrs:grid_label: gn intake_esm_attrs:zstore: gs://cmip6/CMIP6/ScenarioMIP/CMCC/CMCC-... intake_esm_attrs:version: 20210126 intake_esm_attrs:_data_format_: zarr intake_esm_dataset_key: ScenarioMIP.CMCC.CMCC-ESM2.ssp126.r1i1p...ssp126- member_id: 1
- dcpp_init_year: 1
- time: 1032
- x: 292
- y: 362
- vertex: 4
- bnds: 2
- lat(x, y)float64dask.array<chunksize=(292, 362), meta=np.ndarray>
- bounds :
- vertices_latitude
- long_name :
- latitude
- standard_name :
- latitude
- units :
- degrees_north
Array Chunk Bytes 825.81 kiB 825.81 kiB Shape (292, 362) (292, 362) Dask graph 1 chunks in 5 graph layers Data type float64 numpy.ndarray - lon(x, y)float64dask.array<chunksize=(292, 362), meta=np.ndarray>
- bounds :
- vertices_longitude
- long_name :
- longitude
- standard_name :
- longitude
- units :
- degrees_east
Array Chunk Bytes 825.81 kiB 825.81 kiB Shape (292, 362) (292, 362) Dask graph 1 chunks in 8 graph layers Data type float64 numpy.ndarray - time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bnds
- long_name :
- time
- standard_name :
- time
array([cftime.DatetimeNoLeap(2015, 1, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2015, 2, 15, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2015, 3, 16, 12, 0, 0, 0, has_year_zero=True), ..., cftime.DatetimeNoLeap(2100, 10, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2100, 11, 16, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2100, 12, 16, 12, 0, 0, 0, has_year_zero=True)], dtype=object) - lat_verticies(x, y, vertex)float64dask.array<chunksize=(292, 362, 4), meta=np.ndarray>
- units :
- degrees_north
Array Chunk Bytes 3.23 MiB 3.23 MiB Shape (292, 362, 4) (292, 362, 4) Dask graph 1 chunks in 3 graph layers Data type float64 numpy.ndarray - lon_verticies(x, y, vertex)float64dask.array<chunksize=(292, 362, 4), meta=np.ndarray>
- units :
- degrees_east
Array Chunk Bytes 3.23 MiB 3.23 MiB Shape (292, 362, 4) (292, 362, 4) Dask graph 1 chunks in 6 graph layers Data type float64 numpy.ndarray - time_bounds(time, bnds)objectdask.array<chunksize=(1032, 2), meta=np.ndarray>
Array Chunk Bytes 16.12 kiB 16.12 kiB Shape (1032, 2) (1032, 2) Dask graph 1 chunks in 2 graph layers Data type object numpy.ndarray - x(x)int640 1 2 3 4 5 ... 287 288 289 290 291
array([ 0, 1, 2, ..., 289, 290, 291])
- y(y)int640 1 2 3 4 5 ... 357 358 359 360 361
array([ 0, 1, 2, ..., 359, 360, 361])
- lon_bounds(bnds, x, y)float64dask.array<chunksize=(1, 292, 362), meta=np.ndarray>
- units :
- degrees_east
Array Chunk Bytes 1.61 MiB 825.81 kiB Shape (2, 292, 362) (1, 292, 362) Dask graph 2 chunks in 15 graph layers Data type float64 numpy.ndarray - lat_bounds(bnds, x, y)float64dask.array<chunksize=(1, 292, 362), meta=np.ndarray>
- units :
- degrees_north
Array Chunk Bytes 1.61 MiB 825.81 kiB Shape (2, 292, 362) (1, 292, 362) Dask graph 2 chunks in 12 graph layers Data type float64 numpy.ndarray - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- areacello(member_id, dcpp_init_year, x, y)float32dask.array<chunksize=(1, 1, 292, 362), meta=np.ndarray>
- cell_methods :
- area: sum
- comment :
- Horizontal area of ocean grid cells
- history :
- 2021-01-25T13:28:30Z altered by CMOR: Converted type from 'd' to 'f'.
- long_name :
- Grid-Cell Area for Ocean Variables
- original_name :
- e1t, e2t
- standard_name :
- cell_area
- units :
- m²
- original_key :
- ScenarioMIP.CMCC.CMCC-ESM2.ssp126.r1i1p1f1.Ofx.areacello.gn.gs://cmip6/CMIP6/ScenarioMIP/CMCC/CMCC-ESM2/ssp126/r1i1p1f1/Ofx/areacello/gn/v20210126/.20210126
- parsed_with :
- xmip/postprocessing/_parse_metric
Array Chunk Bytes 412.91 kiB 412.91 kiB Shape (1, 1, 292, 362) (1, 1, 292, 362) Dask graph 1 chunks in 3 graph layers Data type float32 numpy.ndarray
- tos(member_id, dcpp_init_year, time, x, y)float32dask.array<chunksize=(1, 1, 252, 292, 362), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- comment :
- Temperature of upper boundary of the liquid ocean, including temperatures below sea-ice and floating ice shelves.
- long_name :
- Sea Surface Temperature
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 416.13 MiB 101.61 MiB Shape (1, 1, 1032, 292, 362) (1, 1, 252, 292, 362) Dask graph 5 chunks in 3 graph layers Data type float32 numpy.ndarray
- Conventions :
- CF-1.7 CMIP-6.2
- activity_id :
- ScenarioMIP AerChemMIP
- branch_method :
- standard
- branch_time_in_child :
- 60225.0
- branch_time_in_parent :
- 60225.0
- cmor_version :
- 3.6.0
- comment :
- none
- contact :
- T. Lovato
- creation_date :
- 2021-02-01T16:02:26Z
- data_specs_version :
- 01.00.31
- experiment :
- gap-filling scenario reaching 7.0 based on SSP3
- experiment_id :
- ssp370
- external_variables :
- areacello
- forcing_index :
- 1
- frequency :
- mon
- further_info_url :
- https://furtherinfo.es-doc.org/CMIP6.CMCC.CMCC-ESM2.ssp370.none.r1i1p1f1
- grid :
- native ocean curvilinear grid
- grid_label :
- gn
- history :
- 2021-02-01T16:02:26Z ;rewrote data to be consistent with ScenarioMIP for variable tos found in table Omon.; none
- initialization_index :
- 1
- institution :
- Fondazione Centro Euro-Mediterraneo sui Cambiamenti Climatici, Lecce 73100, Italy
- institution_id :
- CMCC
- license :
- CMIP6 model data produced by CMCC is licensed under a Creative Commons Attribution ShareAlike 4.0 International License (https://creativecommons.org/licenses). Consult https://pcmdi.llnl.gov/CMIP6/TermsOfUse for terms of use governing CMIP6 output, including citation requirements and proper acknowledgment. Further information about this data, including some limitations, can be found via the further_info_url (recorded as a global attribute in this file) and at https:///pcmdi.llnl.gov/. The data producers and data providers make no warranty, either express or implied, including, but not limited to, warranties of merchantability and fitness for a particular purpose. All liabilities arising from the supply of the information (including any liability arising in negligence) are excluded to the fullest extent permitted by law.
- mip_era :
- CMIP6
- netcdf_tracking_ids :
- hdl:21.14100/dc7a1e92-a64f-4be9-909f-ee5378b2e286
- nominal_resolution :
- 100 km
- parent_activity_id :
- CMIP
- parent_experiment_id :
- historical
- parent_mip_era :
- CMIP6
- parent_source_id :
- CMCC-ESM2
- parent_time_units :
- days since 1850-01-01
- parent_variant_label :
- r1i1p1f1
- physics_index :
- 1
- product :
- model-output
- realization_index :
- 1
- realm :
- ocean
- references :
- none
- run_variant :
- 1st realization
- source :
- CMCC-ESM2 (2017): aerosol: MAM3 atmos: CAM5.3 (1deg; 288 x 192 longitude/latitude; 30 levels; top at ~2 hPa) atmosChem: none land: CLM4.5 (BGC mode) landIce: none ocean: NEMO3.6 (ORCA1 tripolar primarly 1 deg lat/lon with meridional refinement down to 1/3 degree in the tropics; 362 x 292 longitude/latitude; 50 vertical levels; top grid cell 0-1 m) ocnBgchem: BFM5.1 seaIce: CICE4.0
- source_id :
- CMCC-ESM2
- source_type :
- AOGCM BGC
- status :
- 2021-04-07;created; by gcs.cmip6.ldeo@gmail.com
- sub_experiment :
- none
- sub_experiment_id :
- none
- table_id :
- Omon
- table_info :
- Creation Date:(05 February 2020) MD5:6a248fd76c55aa6d6f7b3cc6866b5faf
- title :
- CMCC-ESM2 output prepared for CMIP6
- tracking_id :
- hdl:21.14100/dc7a1e92-a64f-4be9-909f-ee5378b2e286
- variable_id :
- tos
- variant_label :
- r1i1p1f1
- version_id :
- v20210202
- intake_esm_vars :
- ['tos']
- intake_esm_attrs:activity_id :
- ScenarioMIP
- intake_esm_attrs:institution_id :
- CMCC
- intake_esm_attrs:source_id :
- CMCC-ESM2
- intake_esm_attrs:experiment_id :
- ssp370
- intake_esm_attrs:member_id :
- r1i1p1f1
- intake_esm_attrs:table_id :
- Omon
- intake_esm_attrs:variable_id :
- tos
- intake_esm_attrs:grid_label :
- gn
- intake_esm_attrs:zstore :
- gs://cmip6/CMIP6/ScenarioMIP/CMCC/CMCC-ESM2/ssp370/r1i1p1f1/Omon/tos/gn/v20210202/
- intake_esm_attrs:version :
- 20210202
- intake_esm_attrs:_data_format_ :
- zarr
- intake_esm_dataset_key :
- ScenarioMIP.CMCC.CMCC-ESM2.ssp370.r1i1p1f1.Omon.tos.gn.gs://cmip6/CMIP6/ScenarioMIP/CMCC/CMCC-ESM2/ssp370/r1i1p1f1/Omon/tos/gn/v20210202/.20210202
<xarray.DatasetView> Dimensions: (member_id: 1, dcpp_init_year: 1, time: 1032, x: 292, y: 362, vertex: 4, bnds: 2) Coordinates: (12/13) lat (x, y) float64 dask.array<chunksize=(292, 362), meta=np.ndarray> lon (x, y) float64 dask.array<chunksize=(292, 362), meta=np.ndarray> * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 lat_verticies (x, y, vertex) float64 dask.array<chunksize=(292, 362, 4), meta=np.ndarray> lon_verticies (x, y, vertex) float64 dask.array<chunksize=(292, 362, 4), meta=np.ndarray> time_bounds (time, bnds) object dask.array<chunksize=(1032, 2), meta=np.ndarray> ... ... * y (y) int64 0 1 2 3 4 5 6 7 ... 355 356 357 358 359 360 361 lon_bounds (bnds, x, y) float64 dask.array<chunksize=(1, 292, 362), meta=np.ndarray> lat_bounds (bnds, x, y) float64 dask.array<chunksize=(1, 292, 362), meta=np.ndarray> * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan areacello (member_id, dcpp_init_year, x, y) float32 dask.array<chunksize=(1, 1, 292, 362), meta=np.ndarray> Dimensions without coordinates: vertex, bnds Data variables: tos (member_id, dcpp_init_year, time, x, y) float32 dask.array<chunksize=(1, 1, 252, 292, 362), meta=np.ndarray> Attributes: (12/64) Conventions: CF-1.7 CMIP-6.2 activity_id: ScenarioMIP AerChemMIP branch_method: standard branch_time_in_child: 60225.0 branch_time_in_parent: 60225.0 cmor_version: 3.6.0 ... ... intake_esm_attrs:variable_id: tos intake_esm_attrs:grid_label: gn intake_esm_attrs:zstore: gs://cmip6/CMIP6/ScenarioMIP/CMCC/CMCC-... intake_esm_attrs:version: 20210202 intake_esm_attrs:_data_format_: zarr intake_esm_dataset_key: ScenarioMIP.CMCC.CMCC-ESM2.ssp370.r1i1p...ssp370- member_id: 1
- dcpp_init_year: 1
- time: 1032
- x: 292
- y: 362
- vertex: 4
- bnds: 2
- lat(x, y)float64dask.array<chunksize=(292, 362), meta=np.ndarray>
- bounds :
- vertices_latitude
- long_name :
- latitude
- standard_name :
- latitude
- units :
- degrees_north
Array Chunk Bytes 825.81 kiB 825.81 kiB Shape (292, 362) (292, 362) Dask graph 1 chunks in 5 graph layers Data type float64 numpy.ndarray - lon(x, y)float64dask.array<chunksize=(292, 362), meta=np.ndarray>
- bounds :
- vertices_longitude
- long_name :
- longitude
- standard_name :
- longitude
- units :
- degrees_east
Array Chunk Bytes 825.81 kiB 825.81 kiB Shape (292, 362) (292, 362) Dask graph 1 chunks in 8 graph layers Data type float64 numpy.ndarray - time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bnds
- long_name :
- time
- standard_name :
- time
array([cftime.DatetimeNoLeap(2015, 1, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2015, 2, 15, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2015, 3, 16, 12, 0, 0, 0, has_year_zero=True), ..., cftime.DatetimeNoLeap(2100, 10, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2100, 11, 16, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2100, 12, 16, 12, 0, 0, 0, has_year_zero=True)], dtype=object) - lat_verticies(x, y, vertex)float64dask.array<chunksize=(292, 362, 4), meta=np.ndarray>
- units :
- degrees_north
Array Chunk Bytes 3.23 MiB 3.23 MiB Shape (292, 362, 4) (292, 362, 4) Dask graph 1 chunks in 3 graph layers Data type float64 numpy.ndarray - lon_verticies(x, y, vertex)float64dask.array<chunksize=(292, 362, 4), meta=np.ndarray>
- units :
- degrees_east
Array Chunk Bytes 3.23 MiB 3.23 MiB Shape (292, 362, 4) (292, 362, 4) Dask graph 1 chunks in 6 graph layers Data type float64 numpy.ndarray - time_bounds(time, bnds)objectdask.array<chunksize=(1032, 2), meta=np.ndarray>
Array Chunk Bytes 16.12 kiB 16.12 kiB Shape (1032, 2) (1032, 2) Dask graph 1 chunks in 2 graph layers Data type object numpy.ndarray - x(x)int640 1 2 3 4 5 ... 287 288 289 290 291
array([ 0, 1, 2, ..., 289, 290, 291])
- y(y)int640 1 2 3 4 5 ... 357 358 359 360 361
array([ 0, 1, 2, ..., 359, 360, 361])
- lon_bounds(bnds, x, y)float64dask.array<chunksize=(1, 292, 362), meta=np.ndarray>
- units :
- degrees_east
Array Chunk Bytes 1.61 MiB 825.81 kiB Shape (2, 292, 362) (1, 292, 362) Dask graph 2 chunks in 15 graph layers Data type float64 numpy.ndarray - lat_bounds(bnds, x, y)float64dask.array<chunksize=(1, 292, 362), meta=np.ndarray>
- units :
- degrees_north
Array Chunk Bytes 1.61 MiB 825.81 kiB Shape (2, 292, 362) (1, 292, 362) Dask graph 2 chunks in 12 graph layers Data type float64 numpy.ndarray - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- areacello(member_id, dcpp_init_year, x, y)float32dask.array<chunksize=(1, 1, 292, 362), meta=np.ndarray>
- cell_methods :
- area: sum
- comment :
- Horizontal area of ocean grid cells
- history :
- 2021-01-28T20:03:39Z altered by CMOR: Converted type from 'd' to 'f'.
- long_name :
- Grid-Cell Area for Ocean Variables
- original_name :
- e1t, e2t
- standard_name :
- cell_area
- units :
- m²
- original_key :
- ScenarioMIP.CMCC.CMCC-ESM2.ssp245.r1i1p1f1.Ofx.areacello.gn.gs://cmip6/CMIP6/ScenarioMIP/CMCC/CMCC-ESM2/ssp245/r1i1p1f1/Ofx/areacello/gn/v20210129/.20210129
- parsed_with :
- xmip/postprocessing/_parse_metric
Array Chunk Bytes 412.91 kiB 412.91 kiB Shape (1, 1, 292, 362) (1, 1, 292, 362) Dask graph 1 chunks in 3 graph layers Data type float32 numpy.ndarray
- tos(member_id, dcpp_init_year, time, x, y)float32dask.array<chunksize=(1, 1, 252, 292, 362), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- comment :
- Temperature of upper boundary of the liquid ocean, including temperatures below sea-ice and floating ice shelves.
- long_name :
- Sea Surface Temperature
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 416.13 MiB 101.61 MiB Shape (1, 1, 1032, 292, 362) (1, 1, 252, 292, 362) Dask graph 5 chunks in 3 graph layers Data type float32 numpy.ndarray
- Conventions :
- CF-1.7 CMIP-6.2
- activity_id :
- ScenarioMIP
- branch_method :
- standard
- branch_time_in_child :
- 60225.0
- branch_time_in_parent :
- 60225.0
- cmor_version :
- 3.6.0
- comment :
- none
- contact :
- T. Lovato
- creation_date :
- 2021-01-28T20:03:54Z
- data_specs_version :
- 01.00.31
- experiment :
- update of RCP4.5 based on SSP2
- experiment_id :
- ssp245
- external_variables :
- areacello
- forcing_index :
- 1
- frequency :
- mon
- further_info_url :
- https://furtherinfo.es-doc.org/CMIP6.CMCC.CMCC-ESM2.ssp245.none.r1i1p1f1
- grid :
- native ocean curvilinear grid
- grid_label :
- gn
- history :
- 2021-01-28T20:03:54Z ;rewrote data to be consistent with ScenarioMIP for variable tos found in table Omon.; none
- initialization_index :
- 1
- institution :
- Fondazione Centro Euro-Mediterraneo sui Cambiamenti Climatici, Lecce 73100, Italy
- institution_id :
- CMCC
- license :
- CMIP6 model data produced by CMCC is licensed under a Creative Commons Attribution ShareAlike 4.0 International License (https://creativecommons.org/licenses). Consult https://pcmdi.llnl.gov/CMIP6/TermsOfUse for terms of use governing CMIP6 output, including citation requirements and proper acknowledgment. Further information about this data, including some limitations, can be found via the further_info_url (recorded as a global attribute in this file) and at https:///pcmdi.llnl.gov/. The data producers and data providers make no warranty, either express or implied, including, but not limited to, warranties of merchantability and fitness for a particular purpose. All liabilities arising from the supply of the information (including any liability arising in negligence) are excluded to the fullest extent permitted by law.
- mip_era :
- CMIP6
- netcdf_tracking_ids :
- hdl:21.14100/d8b14c9d-c790-4123-88eb-401cee5d4103
- nominal_resolution :
- 100 km
- parent_activity_id :
- CMIP
- parent_experiment_id :
- historical
- parent_mip_era :
- CMIP6
- parent_source_id :
- CMCC-ESM2
- parent_time_units :
- days since 1850-01-01
- parent_variant_label :
- r1i1p1f1
- physics_index :
- 1
- product :
- model-output
- realization_index :
- 1
- realm :
- ocean
- references :
- none
- run_variant :
- 1st realization
- source :
- CMCC-ESM2 (2017): aerosol: MAM3 atmos: CAM5.3 (1deg; 288 x 192 longitude/latitude; 30 levels; top at ~2 hPa) atmosChem: none land: CLM4.5 (BGC mode) landIce: none ocean: NEMO3.6 (ORCA1 tripolar primarly 1 deg lat/lon with meridional refinement down to 1/3 degree in the tropics; 362 x 292 longitude/latitude; 50 vertical levels; top grid cell 0-1 m) ocnBgchem: BFM5.1 seaIce: CICE4.0
- source_id :
- CMCC-ESM2
- source_type :
- AOGCM BGC
- status :
- 2021-02-22;created; by gcs.cmip6.ldeo@gmail.com
- sub_experiment :
- none
- sub_experiment_id :
- none
- table_id :
- Omon
- table_info :
- Creation Date:(05 February 2020) MD5:6a248fd76c55aa6d6f7b3cc6866b5faf
- title :
- CMCC-ESM2 output prepared for CMIP6
- tracking_id :
- hdl:21.14100/d8b14c9d-c790-4123-88eb-401cee5d4103
- variable_id :
- tos
- variant_label :
- r1i1p1f1
- version_id :
- v20210129
- intake_esm_vars :
- ['tos']
- intake_esm_attrs:activity_id :
- ScenarioMIP
- intake_esm_attrs:institution_id :
- CMCC
- intake_esm_attrs:source_id :
- CMCC-ESM2
- intake_esm_attrs:experiment_id :
- ssp245
- intake_esm_attrs:member_id :
- r1i1p1f1
- intake_esm_attrs:table_id :
- Omon
- intake_esm_attrs:variable_id :
- tos
- intake_esm_attrs:grid_label :
- gn
- intake_esm_attrs:zstore :
- gs://cmip6/CMIP6/ScenarioMIP/CMCC/CMCC-ESM2/ssp245/r1i1p1f1/Omon/tos/gn/v20210129/
- intake_esm_attrs:version :
- 20210129
- intake_esm_attrs:_data_format_ :
- zarr
- intake_esm_dataset_key :
- ScenarioMIP.CMCC.CMCC-ESM2.ssp245.r1i1p1f1.Omon.tos.gn.gs://cmip6/CMIP6/ScenarioMIP/CMCC/CMCC-ESM2/ssp245/r1i1p1f1/Omon/tos/gn/v20210129/.20210129
<xarray.DatasetView> Dimensions: (member_id: 1, dcpp_init_year: 1, time: 1032, x: 292, y: 362, vertex: 4, bnds: 2) Coordinates: (12/13) lat (x, y) float64 dask.array<chunksize=(292, 362), meta=np.ndarray> lon (x, y) float64 dask.array<chunksize=(292, 362), meta=np.ndarray> * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 lat_verticies (x, y, vertex) float64 dask.array<chunksize=(292, 362, 4), meta=np.ndarray> lon_verticies (x, y, vertex) float64 dask.array<chunksize=(292, 362, 4), meta=np.ndarray> time_bounds (time, bnds) object dask.array<chunksize=(1032, 2), meta=np.ndarray> ... ... * y (y) int64 0 1 2 3 4 5 6 7 ... 355 356 357 358 359 360 361 lon_bounds (bnds, x, y) float64 dask.array<chunksize=(1, 292, 362), meta=np.ndarray> lat_bounds (bnds, x, y) float64 dask.array<chunksize=(1, 292, 362), meta=np.ndarray> * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan areacello (member_id, dcpp_init_year, x, y) float32 dask.array<chunksize=(1, 1, 292, 362), meta=np.ndarray> Dimensions without coordinates: vertex, bnds Data variables: tos (member_id, dcpp_init_year, time, x, y) float32 dask.array<chunksize=(1, 1, 252, 292, 362), meta=np.ndarray> Attributes: (12/64) Conventions: CF-1.7 CMIP-6.2 activity_id: ScenarioMIP branch_method: standard branch_time_in_child: 60225.0 branch_time_in_parent: 60225.0 cmor_version: 3.6.0 ... ... intake_esm_attrs:variable_id: tos intake_esm_attrs:grid_label: gn intake_esm_attrs:zstore: gs://cmip6/CMIP6/ScenarioMIP/CMCC/CMCC-... intake_esm_attrs:version: 20210129 intake_esm_attrs:_data_format_: zarr intake_esm_dataset_key: ScenarioMIP.CMCC.CMCC-ESM2.ssp245.r1i1p...ssp245- member_id: 1
- dcpp_init_year: 1
- time: 1032
- x: 292
- y: 362
- vertex: 4
- bnds: 2
- lat(x, y)float64dask.array<chunksize=(292, 362), meta=np.ndarray>
- bounds :
- vertices_latitude
- long_name :
- latitude
- standard_name :
- latitude
- units :
- degrees_north
Array Chunk Bytes 825.81 kiB 825.81 kiB Shape (292, 362) (292, 362) Dask graph 1 chunks in 5 graph layers Data type float64 numpy.ndarray - lon(x, y)float64dask.array<chunksize=(292, 362), meta=np.ndarray>
- bounds :
- vertices_longitude
- long_name :
- longitude
- standard_name :
- longitude
- units :
- degrees_east
Array Chunk Bytes 825.81 kiB 825.81 kiB Shape (292, 362) (292, 362) Dask graph 1 chunks in 8 graph layers Data type float64 numpy.ndarray - time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bnds
- long_name :
- time
- standard_name :
- time
array([cftime.DatetimeNoLeap(2015, 1, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2015, 2, 15, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2015, 3, 16, 12, 0, 0, 0, has_year_zero=True), ..., cftime.DatetimeNoLeap(2100, 10, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2100, 11, 16, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2100, 12, 16, 12, 0, 0, 0, has_year_zero=True)], dtype=object) - lat_verticies(x, y, vertex)float64dask.array<chunksize=(292, 362, 4), meta=np.ndarray>
- units :
- degrees_north
Array Chunk Bytes 3.23 MiB 3.23 MiB Shape (292, 362, 4) (292, 362, 4) Dask graph 1 chunks in 3 graph layers Data type float64 numpy.ndarray - lon_verticies(x, y, vertex)float64dask.array<chunksize=(292, 362, 4), meta=np.ndarray>
- units :
- degrees_east
Array Chunk Bytes 3.23 MiB 3.23 MiB Shape (292, 362, 4) (292, 362, 4) Dask graph 1 chunks in 6 graph layers Data type float64 numpy.ndarray - time_bounds(time, bnds)objectdask.array<chunksize=(1032, 2), meta=np.ndarray>
Array Chunk Bytes 16.12 kiB 16.12 kiB Shape (1032, 2) (1032, 2) Dask graph 1 chunks in 2 graph layers Data type object numpy.ndarray - x(x)int640 1 2 3 4 5 ... 287 288 289 290 291
array([ 0, 1, 2, ..., 289, 290, 291])
- y(y)int640 1 2 3 4 5 ... 357 358 359 360 361
array([ 0, 1, 2, ..., 359, 360, 361])
- lon_bounds(bnds, x, y)float64dask.array<chunksize=(1, 292, 362), meta=np.ndarray>
- units :
- degrees_east
Array Chunk Bytes 1.61 MiB 825.81 kiB Shape (2, 292, 362) (1, 292, 362) Dask graph 2 chunks in 15 graph layers Data type float64 numpy.ndarray - lat_bounds(bnds, x, y)float64dask.array<chunksize=(1, 292, 362), meta=np.ndarray>
- units :
- degrees_north
Array Chunk Bytes 1.61 MiB 825.81 kiB Shape (2, 292, 362) (1, 292, 362) Dask graph 2 chunks in 12 graph layers Data type float64 numpy.ndarray - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- areacello(member_id, dcpp_init_year, x, y)float32dask.array<chunksize=(1, 1, 292, 362), meta=np.ndarray>
- cell_methods :
- area: sum
- comment :
- Horizontal area of ocean grid cells
- history :
- 2021-01-20T12:47:16Z altered by CMOR: Converted type from 'd' to 'f'.
- long_name :
- Grid-Cell Area for Ocean Variables
- original_name :
- e1t, e2t
- standard_name :
- cell_area
- units :
- m²
- original_key :
- ScenarioMIP.CMCC.CMCC-ESM2.ssp585.r1i1p1f1.Ofx.areacello.gn.gs://cmip6/CMIP6/ScenarioMIP/CMCC/CMCC-ESM2/ssp585/r1i1p1f1/Ofx/areacello/gn/v20210126/.20210126
- parsed_with :
- xmip/postprocessing/_parse_metric
Array Chunk Bytes 412.91 kiB 412.91 kiB Shape (1, 1, 292, 362) (1, 1, 292, 362) Dask graph 1 chunks in 3 graph layers Data type float32 numpy.ndarray
- tos(member_id, dcpp_init_year, time, x, y)float32dask.array<chunksize=(1, 1, 253, 292, 362), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- comment :
- Temperature of upper boundary of the liquid ocean, including temperatures below sea-ice and floating ice shelves.
- long_name :
- Sea Surface Temperature
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 416.13 MiB 102.02 MiB Shape (1, 1, 1032, 292, 362) (1, 1, 253, 292, 362) Dask graph 5 chunks in 3 graph layers Data type float32 numpy.ndarray
- Conventions :
- CF-1.7 CMIP-6.2
- activity_id :
- ScenarioMIP
- branch_method :
- standard
- branch_time_in_child :
- 60225.0
- branch_time_in_parent :
- 60225.0
- cmor_version :
- 3.6.0
- comment :
- none
- contact :
- T. Lovato
- creation_date :
- 2021-01-20T12:47:59Z
- data_specs_version :
- 01.00.31
- experiment :
- update of RCP8.5 based on SSP5
- experiment_id :
- ssp585
- external_variables :
- areacello
- forcing_index :
- 1
- frequency :
- mon
- further_info_url :
- https://furtherinfo.es-doc.org/CMIP6.CMCC.CMCC-ESM2.ssp585.none.r1i1p1f1
- grid :
- native ocean curvilinear grid
- grid_label :
- gn
- history :
- 2021-01-20T12:47:59Z ;rewrote data to be consistent with ScenarioMIP for variable tos found in table Omon.; none
- initialization_index :
- 1
- institution :
- Fondazione Centro Euro-Mediterraneo sui Cambiamenti Climatici, Lecce 73100, Italy
- institution_id :
- CMCC
- license :
- CMIP6 model data produced by CMCC is licensed under a Creative Commons Attribution ShareAlike 4.0 International License (https://creativecommons.org/licenses). Consult https://pcmdi.llnl.gov/CMIP6/TermsOfUse for terms of use governing CMIP6 output, including citation requirements and proper acknowledgment. Further information about this data, including some limitations, can be found via the further_info_url (recorded as a global attribute in this file) and at https:///pcmdi.llnl.gov/. The data producers and data providers make no warranty, either express or implied, including, but not limited to, warranties of merchantability and fitness for a particular purpose. All liabilities arising from the supply of the information (including any liability arising in negligence) are excluded to the fullest extent permitted by law.
- mip_era :
- CMIP6
- netcdf_tracking_ids :
- hdl:21.14100/ddf593f1-127a-480c-884f-c8c5cb0588a2
- nominal_resolution :
- 100 km
- parent_activity_id :
- CMIP
- parent_experiment_id :
- historical
- parent_mip_era :
- CMIP6
- parent_source_id :
- CMCC-ESM2
- parent_time_units :
- days since 1850-01-01
- parent_variant_label :
- r1i1p1f1
- physics_index :
- 1
- product :
- model-output
- realization_index :
- 1
- realm :
- ocean
- references :
- none
- run_variant :
- 1st realization
- source :
- CMCC-ESM2 (2017): aerosol: MAM3 atmos: CAM5.3 (1deg; 288 x 192 longitude/latitude; 30 levels; top at ~2 hPa) atmosChem: none land: CLM4.5 (BGC mode) landIce: none ocean: NEMO3.6 (ORCA1 tripolar primarly 1 deg lat/lon with meridional refinement down to 1/3 degree in the tropics; 362 x 292 longitude/latitude; 50 vertical levels; top grid cell 0-1 m) ocnBgchem: BFM5.1 seaIce: CICE4.0
- source_id :
- CMCC-ESM2
- source_type :
- AOGCM BGC
- status :
- 2021-02-22;created; by gcs.cmip6.ldeo@gmail.com
- sub_experiment :
- none
- sub_experiment_id :
- none
- table_id :
- Omon
- table_info :
- Creation Date:(05 February 2020) MD5:6a248fd76c55aa6d6f7b3cc6866b5faf
- title :
- CMCC-ESM2 output prepared for CMIP6
- tracking_id :
- hdl:21.14100/ddf593f1-127a-480c-884f-c8c5cb0588a2
- variable_id :
- tos
- variant_label :
- r1i1p1f1
- version_id :
- v20210126
- intake_esm_vars :
- ['tos']
- intake_esm_attrs:activity_id :
- ScenarioMIP
- intake_esm_attrs:institution_id :
- CMCC
- intake_esm_attrs:source_id :
- CMCC-ESM2
- intake_esm_attrs:experiment_id :
- ssp585
- intake_esm_attrs:member_id :
- r1i1p1f1
- intake_esm_attrs:table_id :
- Omon
- intake_esm_attrs:variable_id :
- tos
- intake_esm_attrs:grid_label :
- gn
- intake_esm_attrs:zstore :
- gs://cmip6/CMIP6/ScenarioMIP/CMCC/CMCC-ESM2/ssp585/r1i1p1f1/Omon/tos/gn/v20210126/
- intake_esm_attrs:version :
- 20210126
- intake_esm_attrs:_data_format_ :
- zarr
- intake_esm_dataset_key :
- ScenarioMIP.CMCC.CMCC-ESM2.ssp585.r1i1p1f1.Omon.tos.gn.gs://cmip6/CMIP6/ScenarioMIP/CMCC/CMCC-ESM2/ssp585/r1i1p1f1/Omon/tos/gn/v20210126/.20210126
<xarray.DatasetView> Dimensions: (member_id: 1, dcpp_init_year: 1, time: 1032, x: 292, y: 362, vertex: 4, bnds: 2) Coordinates: (12/13) lat (x, y) float64 dask.array<chunksize=(292, 362), meta=np.ndarray> lon (x, y) float64 dask.array<chunksize=(292, 362), meta=np.ndarray> * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 lat_verticies (x, y, vertex) float64 dask.array<chunksize=(292, 362, 4), meta=np.ndarray> lon_verticies (x, y, vertex) float64 dask.array<chunksize=(292, 362, 4), meta=np.ndarray> time_bounds (time, bnds) object dask.array<chunksize=(1032, 2), meta=np.ndarray> ... ... * y (y) int64 0 1 2 3 4 5 6 7 ... 355 356 357 358 359 360 361 lon_bounds (bnds, x, y) float64 dask.array<chunksize=(1, 292, 362), meta=np.ndarray> lat_bounds (bnds, x, y) float64 dask.array<chunksize=(1, 292, 362), meta=np.ndarray> * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan areacello (member_id, dcpp_init_year, x, y) float32 dask.array<chunksize=(1, 1, 292, 362), meta=np.ndarray> Dimensions without coordinates: vertex, bnds Data variables: tos (member_id, dcpp_init_year, time, x, y) float32 dask.array<chunksize=(1, 1, 253, 292, 362), meta=np.ndarray> Attributes: (12/64) Conventions: CF-1.7 CMIP-6.2 activity_id: ScenarioMIP branch_method: standard branch_time_in_child: 60225.0 branch_time_in_parent: 60225.0 cmor_version: 3.6.0 ... ... intake_esm_attrs:variable_id: tos intake_esm_attrs:grid_label: gn intake_esm_attrs:zstore: gs://cmip6/CMIP6/ScenarioMIP/CMCC/CMCC-... intake_esm_attrs:version: 20210126 intake_esm_attrs:_data_format_: zarr intake_esm_dataset_key: ScenarioMIP.CMCC.CMCC-ESM2.ssp585.r1i1p...ssp585- member_id: 1
- dcpp_init_year: 1
- time: 1980
- x: 292
- y: 362
- vertex: 4
- bnds: 2
- lat(x, y)float64dask.array<chunksize=(292, 362), meta=np.ndarray>
- bounds :
- vertices_latitude
- long_name :
- latitude
- standard_name :
- latitude
- units :
- degrees_north
Array Chunk Bytes 825.81 kiB 825.81 kiB Shape (292, 362) (292, 362) Dask graph 1 chunks in 5 graph layers Data type float64 numpy.ndarray - lon(x, y)float64dask.array<chunksize=(292, 362), meta=np.ndarray>
- bounds :
- vertices_longitude
- long_name :
- longitude
- standard_name :
- longitude
- units :
- degrees_east
Array Chunk Bytes 825.81 kiB 825.81 kiB Shape (292, 362) (292, 362) Dask graph 1 chunks in 8 graph layers Data type float64 numpy.ndarray - time(time)object1850-01-16 12:00:00 ... 2014-12-...
- axis :
- T
- bounds :
- time_bnds
- long_name :
- time
- standard_name :
- time
array([cftime.DatetimeNoLeap(1850, 1, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(1850, 2, 15, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(1850, 3, 16, 12, 0, 0, 0, has_year_zero=True), ..., cftime.DatetimeNoLeap(2014, 10, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2014, 11, 16, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2014, 12, 16, 12, 0, 0, 0, has_year_zero=True)], dtype=object) - lat_verticies(x, y, vertex)float64dask.array<chunksize=(292, 362, 4), meta=np.ndarray>
- units :
- degrees_north
Array Chunk Bytes 3.23 MiB 3.23 MiB Shape (292, 362, 4) (292, 362, 4) Dask graph 1 chunks in 3 graph layers Data type float64 numpy.ndarray - lon_verticies(x, y, vertex)float64dask.array<chunksize=(292, 362, 4), meta=np.ndarray>
- units :
- degrees_east
Array Chunk Bytes 3.23 MiB 3.23 MiB Shape (292, 362, 4) (292, 362, 4) Dask graph 1 chunks in 6 graph layers Data type float64 numpy.ndarray - time_bounds(time, bnds)objectdask.array<chunksize=(1980, 2), meta=np.ndarray>
Array Chunk Bytes 30.94 kiB 30.94 kiB Shape (1980, 2) (1980, 2) Dask graph 1 chunks in 2 graph layers Data type object numpy.ndarray - x(x)int640 1 2 3 4 5 ... 287 288 289 290 291
array([ 0, 1, 2, ..., 289, 290, 291])
- y(y)int640 1 2 3 4 5 ... 357 358 359 360 361
array([ 0, 1, 2, ..., 359, 360, 361])
- lon_bounds(bnds, x, y)float64dask.array<chunksize=(1, 292, 362), meta=np.ndarray>
- units :
- degrees_east
Array Chunk Bytes 1.61 MiB 825.81 kiB Shape (2, 292, 362) (1, 292, 362) Dask graph 2 chunks in 15 graph layers Data type float64 numpy.ndarray - lat_bounds(bnds, x, y)float64dask.array<chunksize=(1, 292, 362), meta=np.ndarray>
- units :
- degrees_north
Array Chunk Bytes 1.61 MiB 825.81 kiB Shape (2, 292, 362) (1, 292, 362) Dask graph 2 chunks in 12 graph layers Data type float64 numpy.ndarray - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- areacello(member_id, dcpp_init_year, x, y)float32dask.array<chunksize=(1, 1, 292, 362), meta=np.ndarray>
- cell_methods :
- area: sum
- comment :
- Horizontal area of ocean grid cells
- history :
- 2020-12-25T16:35:15Z altered by CMOR: Converted type from 'd' to 'f'.
- long_name :
- Grid-Cell Area for Ocean Variables
- original_name :
- e1t, e2t
- standard_name :
- cell_area
- units :
- m²
- original_key :
- CMIP.CMCC.CMCC-ESM2.historical.r1i1p1f1.Ofx.areacello.gn.gs://cmip6/CMIP6/CMIP/CMCC/CMCC-ESM2/historical/r1i1p1f1/Ofx/areacello/gn/v20210114/.20210114
- parsed_with :
- xmip/postprocessing/_parse_metric
Array Chunk Bytes 412.91 kiB 412.91 kiB Shape (1, 1, 292, 362) (1, 1, 292, 362) Dask graph 1 chunks in 3 graph layers Data type float32 numpy.ndarray
- tos(member_id, dcpp_init_year, time, x, y)float32dask.array<chunksize=(1, 1, 255, 292, 362), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- comment :
- Temperature of upper boundary of the liquid ocean, including temperatures below sea-ice and floating ice shelves.
- long_name :
- Sea Surface Temperature
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 798.39 MiB 102.82 MiB Shape (1, 1, 1980, 292, 362) (1, 1, 255, 292, 362) Dask graph 8 chunks in 3 graph layers Data type float32 numpy.ndarray
- Conventions :
- CF-1.7 CMIP-6.2
- activity_id :
- CMIP
- branch_method :
- standard
- branch_time_in_child :
- 0.0
- branch_time_in_parent :
- 0.0
- cmor_version :
- 3.6.0
- comment :
- none
- contact :
- T. Lovato
- creation_date :
- 2020-12-25T16:35:42Z
- data_specs_version :
- 01.00.31
- experiment :
- all-forcing simulation of the recent past
- experiment_id :
- historical
- external_variables :
- areacello
- forcing_index :
- 1
- frequency :
- mon
- further_info_url :
- https://furtherinfo.es-doc.org/CMIP6.CMCC.CMCC-ESM2.historical.none.r1i1p1f1
- grid :
- native ocean curvilinear grid
- grid_label :
- gn
- history :
- 2020-12-25T16:35:42Z ;rewrote data to be consistent with CMIP for variable tos found in table Omon.; none
- initialization_index :
- 1
- institution :
- Fondazione Centro Euro-Mediterraneo sui Cambiamenti Climatici, Lecce 73100, Italy
- institution_id :
- CMCC
- license :
- CMIP6 model data produced by CMCC is licensed under a Creative Commons Attribution ShareAlike 4.0 International License (https://creativecommons.org/licenses). Consult https://pcmdi.llnl.gov/CMIP6/TermsOfUse for terms of use governing CMIP6 output, including citation requirements and proper acknowledgment. Further information about this data, including some limitations, can be found via the further_info_url (recorded as a global attribute in this file) and at https:///pcmdi.llnl.gov/. The data producers and data providers make no warranty, either express or implied, including, but not limited to, warranties of merchantability and fitness for a particular purpose. All liabilities arising from the supply of the information (including any liability arising in negligence) are excluded to the fullest extent permitted by law.
- mip_era :
- CMIP6
- nominal_resolution :
- 100 km
- parent_activity_id :
- CMIP
- parent_experiment_id :
- piControl
- parent_mip_era :
- CMIP6
- parent_source_id :
- CMCC-ESM2
- parent_time_units :
- days since 1850-01-01
- parent_variant_label :
- r1i1p1f1
- physics_index :
- 1
- product :
- model-output
- realization_index :
- 1
- realm :
- ocean
- references :
- none
- run_variant :
- 1st realization
- source :
- CMCC-ESM2 (2017): aerosol: MAM3 atmos: CAM5.3 (1deg; 288 x 192 longitude/latitude; 30 levels; top at ~2 hPa) atmosChem: none land: CLM4.5 (BGC mode) landIce: none ocean: NEMO3.6 (ORCA1 tripolar primarly 1 deg lat/lon with meridional refinement down to 1/3 degree in the tropics; 362 x 292 longitude/latitude; 50 vertical levels; top grid cell 0-1 m) ocnBgchem: BFM5.1 seaIce: CICE4.0
- source_id :
- CMCC-ESM2
- source_type :
- AOGCM
- status :
- 2021-01-19;created; by gcs.cmip6.ldeo@gmail.com
- sub_experiment :
- none
- sub_experiment_id :
- none
- table_id :
- Omon
- table_info :
- Creation Date:(05 February 2020) MD5:6a248fd76c55aa6d6f7b3cc6866b5faf
- title :
- CMCC-ESM2 output prepared for CMIP6
- tracking_id :
- hdl:21.14100/688e38fa-19d8-4550-afdd-18f161fa2fdb
- variable_id :
- tos
- variant_label :
- r1i1p1f1
- netcdf_tracking_ids :
- hdl:21.14100/688e38fa-19d8-4550-afdd-18f161fa2fdb
- version_id :
- v20210114
- intake_esm_vars :
- ['tos']
- intake_esm_attrs:activity_id :
- CMIP
- intake_esm_attrs:institution_id :
- CMCC
- intake_esm_attrs:source_id :
- CMCC-ESM2
- intake_esm_attrs:experiment_id :
- historical
- intake_esm_attrs:member_id :
- r1i1p1f1
- intake_esm_attrs:table_id :
- Omon
- intake_esm_attrs:variable_id :
- tos
- intake_esm_attrs:grid_label :
- gn
- intake_esm_attrs:zstore :
- gs://cmip6/CMIP6/CMIP/CMCC/CMCC-ESM2/historical/r1i1p1f1/Omon/tos/gn/v20210114/
- intake_esm_attrs:version :
- 20210114
- intake_esm_attrs:_data_format_ :
- zarr
- intake_esm_dataset_key :
- CMIP.CMCC.CMCC-ESM2.historical.r1i1p1f1.Omon.tos.gn.gs://cmip6/CMIP6/CMIP/CMCC/CMCC-ESM2/historical/r1i1p1f1/Omon/tos/gn/v20210114/.20210114
<xarray.DatasetView> Dimensions: (member_id: 1, dcpp_init_year: 1, time: 1980, x: 292, y: 362, vertex: 4, bnds: 2) Coordinates: (12/13) lat (x, y) float64 dask.array<chunksize=(292, 362), meta=np.ndarray> lon (x, y) float64 dask.array<chunksize=(292, 362), meta=np.ndarray> * time (time) object 1850-01-16 12:00:00 ... 2014-12-16 12:00:00 lat_verticies (x, y, vertex) float64 dask.array<chunksize=(292, 362, 4), meta=np.ndarray> lon_verticies (x, y, vertex) float64 dask.array<chunksize=(292, 362, 4), meta=np.ndarray> time_bounds (time, bnds) object dask.array<chunksize=(1980, 2), meta=np.ndarray> ... ... * y (y) int64 0 1 2 3 4 5 6 7 ... 355 356 357 358 359 360 361 lon_bounds (bnds, x, y) float64 dask.array<chunksize=(1, 292, 362), meta=np.ndarray> lat_bounds (bnds, x, y) float64 dask.array<chunksize=(1, 292, 362), meta=np.ndarray> * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan areacello (member_id, dcpp_init_year, x, y) float32 dask.array<chunksize=(1, 1, 292, 362), meta=np.ndarray> Dimensions without coordinates: vertex, bnds Data variables: tos (member_id, dcpp_init_year, time, x, y) float32 dask.array<chunksize=(1, 1, 255, 292, 362), meta=np.ndarray> Attributes: (12/64) Conventions: CF-1.7 CMIP-6.2 activity_id: CMIP branch_method: standard branch_time_in_child: 0.0 branch_time_in_parent: 0.0 cmor_version: 3.6.0 ... ... intake_esm_attrs:variable_id: tos intake_esm_attrs:grid_label: gn intake_esm_attrs:zstore: gs://cmip6/CMIP6/CMIP/CMCC/CMCC-ESM2/hi... intake_esm_attrs:version: 20210114 intake_esm_attrs:_data_format_: zarr intake_esm_dataset_key: CMIP.CMCC.CMCC-ESM2.historical.r1i1p1f1...historical
<xarray.DatasetView> Dimensions: () Data variables: *empty*CMCC-ESM2
# dt.nbytes / 1e9
Para acceder a los datos dentro de este Xarray.datatree usamos notación de diccionarios de Python
dt["IPSL-CM6A-LR/historical"].ds
<xarray.DatasetView>
Dimensions: (member_id: 1, y: 332, x: 362, dcpp_init_year: 1,
time: 1980, vertex: 4, bnds: 2)
Coordinates: (12/13)
lat (y, x) float32 dask.array<chunksize=(332, 362), meta=np.ndarray>
lon (y, x) float32 dask.array<chunksize=(332, 362), meta=np.ndarray>
* time (time) object 1850-01-16 12:00:00 ... 2014-12-16 12:00:00
lat_verticies (y, x, vertex) float32 dask.array<chunksize=(332, 362, 4), meta=np.ndarray>
lon_verticies (y, x, vertex) float32 dask.array<chunksize=(332, 362, 4), meta=np.ndarray>
time_bounds (time, bnds) object dask.array<chunksize=(1980, 2), meta=np.ndarray>
... ...
* x (x) int64 0 1 2 3 4 5 6 7 ... 355 356 357 358 359 360 361
lon_bounds (bnds, y, x) float32 dask.array<chunksize=(1, 332, 362), meta=np.ndarray>
lat_bounds (bnds, y, x) float32 dask.array<chunksize=(1, 332, 362), meta=np.ndarray>
* member_id (member_id) object 'r1i1p1f1'
* dcpp_init_year (dcpp_init_year) float64 nan
areacello (member_id, dcpp_init_year, y, x) float32 dask.array<chunksize=(1, 1, 332, 362), meta=np.ndarray>
Dimensions without coordinates: vertex, bnds
Data variables:
area (member_id, y, x) float32 dask.array<chunksize=(1, 332, 362), meta=np.ndarray>
tos (member_id, dcpp_init_year, time, y, x) float32 dask.array<chunksize=(1, 1, 252, 332, 362), meta=np.ndarray>
Attributes: (12/67)
CMIP6_CV_version: cv=6.2.3.5-2-g63b123e
Conventions: CF-1.7 CMIP-6.2
EXPID: historical
NCO: "4.6.0"
activity_id: CMIP
branch_method: standard
... ...
intake_esm_attrs:variable_id: tos
intake_esm_attrs:grid_label: gn
intake_esm_attrs:zstore: gs://cmip6/CMIP6/CMIP/IPSL/IPSL-CM6A-LR...
intake_esm_attrs:version: 20180803
intake_esm_attrs:_data_format_: zarr
intake_esm_dataset_key: CMIP.IPSL.IPSL-CM6A-LR.historical.r1i1p...- member_id: 1
- y: 332
- x: 362
- dcpp_init_year: 1
- time: 1980
- vertex: 4
- bnds: 2
- lat(y, x)float32dask.array<chunksize=(332, 362), meta=np.ndarray>
- bounds :
- bounds_nav_lat
- long_name :
- Latitude
- standard_name :
- latitude
- units :
- degrees_north
Array Chunk Bytes 469.47 kiB 469.47 kiB Shape (332, 362) (332, 362) Dask graph 1 chunks in 5 graph layers Data type float32 numpy.ndarray - lon(y, x)float32dask.array<chunksize=(332, 362), meta=np.ndarray>
- bounds :
- bounds_nav_lon
- long_name :
- Longitude
- standard_name :
- longitude
- units :
- degrees_east
Array Chunk Bytes 469.47 kiB 469.47 kiB Shape (332, 362) (332, 362) Dask graph 1 chunks in 8 graph layers Data type float32 numpy.ndarray - time(time)object1850-01-16 12:00:00 ... 2014-12-...
- axis :
- T
- bounds :
- time_bounds
- long_name :
- Time axis
- standard_name :
- time
- time_origin :
- 1850-01-01 00:00:00
array([cftime.DatetimeGregorian(1850, 1, 16, 12, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(1850, 2, 15, 0, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(1850, 3, 16, 12, 0, 0, 0, has_year_zero=False), ..., cftime.DatetimeGregorian(2014, 10, 16, 12, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2014, 11, 16, 0, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2014, 12, 16, 12, 0, 0, 0, has_year_zero=False)], dtype=object) - lat_verticies(y, x, vertex)float32dask.array<chunksize=(332, 362, 4), meta=np.ndarray>
Array Chunk Bytes 1.83 MiB 1.83 MiB Shape (332, 362, 4) (332, 362, 4) Dask graph 1 chunks in 3 graph layers Data type float32 numpy.ndarray - lon_verticies(y, x, vertex)float32dask.array<chunksize=(332, 362, 4), meta=np.ndarray>
Array Chunk Bytes 1.83 MiB 1.83 MiB Shape (332, 362, 4) (332, 362, 4) Dask graph 1 chunks in 6 graph layers Data type float32 numpy.ndarray - time_bounds(time, bnds)objectdask.array<chunksize=(1980, 2), meta=np.ndarray>
Array Chunk Bytes 30.94 kiB 30.94 kiB Shape (1980, 2) (1980, 2) Dask graph 1 chunks in 2 graph layers Data type object numpy.ndarray - y(y)int640 1 2 3 4 5 ... 327 328 329 330 331
array([ 0, 1, 2, ..., 329, 330, 331])
- x(x)int640 1 2 3 4 5 ... 357 358 359 360 361
array([ 0, 1, 2, ..., 359, 360, 361])
- lon_bounds(bnds, y, x)float32dask.array<chunksize=(1, 332, 362), meta=np.ndarray>
Array Chunk Bytes 0.92 MiB 469.47 kiB Shape (2, 332, 362) (1, 332, 362) Dask graph 2 chunks in 15 graph layers Data type float32 numpy.ndarray - lat_bounds(bnds, y, x)float32dask.array<chunksize=(1, 332, 362), meta=np.ndarray>
Array Chunk Bytes 0.92 MiB 469.47 kiB Shape (2, 332, 362) (1, 332, 362) Dask graph 2 chunks in 12 graph layers Data type float32 numpy.ndarray - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- areacello(member_id, dcpp_init_year, y, x)float32dask.array<chunksize=(1, 1, 332, 362), meta=np.ndarray>
- cell_measures :
- area: area
- cell_methods :
- area: sum
- description :
- Cell areas for any grid used to report ocean variables and variables which are requested as used on the model ocean grid (e.g. hfsso, which is a downward heat flux from the atmosphere interpolated onto the ocean grid). These cell areas should be defined to enable exact calculation of global integrals (e.g., of vertical fluxes of energy at the surface and top of the atmosphere).
- history :
- none
- long_name :
- Grid-Cell Area
- online_operation :
- once
- standard_name :
- cell_area
- units :
- m²
- original_key :
- CMIP.IPSL.IPSL-CM6A-LR.historical.r1i1p1f1.Ofx.areacello.gn.gs://cmip6/CMIP6/CMIP/IPSL/IPSL-CM6A-LR/historical/r1i1p1f1/Ofx/areacello/gn/v20180803/.20180803
- parsed_with :
- xmip/postprocessing/_parse_metric
Array Chunk Bytes 469.47 kiB 469.47 kiB Shape (1, 1, 332, 362) (1, 1, 332, 362) Dask graph 1 chunks in 3 graph layers Data type float32 numpy.ndarray
- area(member_id, y, x)float32dask.array<chunksize=(1, 332, 362), meta=np.ndarray>
- standard_name :
- cell_area
- units :
- m²
Array Chunk Bytes 469.47 kiB 469.47 kiB Shape (1, 332, 362) (1, 332, 362) Dask graph 1 chunks in 3 graph layers Data type float32 numpy.ndarray - tos(member_id, dcpp_init_year, time, y, x)float32dask.array<chunksize=(1, 1, 252, 332, 362), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- description :
- This may differ from "surface temperature" in regions of sea ice or floating ice shelves. For models using conservative temperature as the prognostic field, they should report the top ocean layer as surface potential temperature, which is the same as surface in situ temperature.
- history :
- none
- interval_operation :
- 2700 s
- interval_write :
- 1 month
- long_name :
- Sea Surface Temperature
- online_operation :
- average
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 907.76 MiB 115.53 MiB Shape (1, 1, 1980, 332, 362) (1, 1, 252, 332, 362) Dask graph 8 chunks in 3 graph layers Data type float32 numpy.ndarray
- timePandasIndex
PandasIndex(CFTimeIndex([1850-01-16 12:00:00, 1850-02-15 00:00:00, 1850-03-16 12:00:00, 1850-04-16 00:00:00, 1850-05-16 12:00:00, 1850-06-16 00:00:00, 1850-07-16 12:00:00, 1850-08-16 12:00:00, 1850-09-16 00:00:00, 1850-10-16 12:00:00, ... 2014-03-16 12:00:00, 2014-04-16 00:00:00, 2014-05-16 12:00:00, 2014-06-16 00:00:00, 2014-07-16 12:00:00, 2014-08-16 12:00:00, 2014-09-16 00:00:00, 2014-10-16 12:00:00, 2014-11-16 00:00:00, 2014-12-16 12:00:00], dtype='object', length=1980, calendar='standard', freq='None')) - yPandasIndex
PandasIndex(RangeIndex(start=0, stop=332, step=1, name='y'))
- xPandasIndex
PandasIndex(RangeIndex(start=0, stop=362, step=1, name='x'))
- member_idPandasIndex
PandasIndex(Index(['r1i1p1f1'], dtype='object', name='member_id'))
- dcpp_init_yearPandasIndex
PandasIndex(Index([nan], dtype='float64', name='dcpp_init_year'))
- CMIP6_CV_version :
- cv=6.2.3.5-2-g63b123e
- Conventions :
- CF-1.7 CMIP-6.2
- EXPID :
- historical
- NCO :
- "4.6.0"
- activity_id :
- CMIP
- branch_method :
- standard
- branch_time_in_child :
- 0.0
- branch_time_in_parent :
- 21914.0
- contact :
- ipsl-cmip6@listes.ipsl.fr
- creation_date :
- 2018-07-11T07:36:14Z
- data_specs_version :
- 01.00.21
- description :
- CMIP6 historical
- dr2xml_md5sum :
- f1e40c1fc5d8281f865f72fbf4e38f9d
- dr2xml_version :
- 1.11
- experiment :
- all-forcing simulation of the recent past
- experiment_id :
- historical
- external_variables :
- areacello
- forcing_index :
- 1
- frequency :
- mon
- further_info_url :
- https://furtherinfo.es-doc.org/CMIP6.IPSL.IPSL-CM6A-LR.historical.none.r1i1p1f1
- grid :
- native ocean tri-polar grid with 105 k ocean cells
- grid_label :
- gn
- history :
- Sat Dec 1 12:15:54 2018: ncatted -O -a realization_index,global,m,i,1 /ccc/work/cont003/cmip6/cmip6/onhold/CM61-LR-histEXT-03.1910/files+ext/tos_Omon_IPSL-CM6A-LR_historical_r1i1p1f1_gn_185001-201412.nc Sat Dec 1 12:09:05 2018: ncatted -O -a realization_index,global,m,i,1 /ccc/work/cont003/cmip6/cmip6/onhold/CM61-LR-hist-03.1910/files/tos_Omon_IPSL-CM6A-LR_historical_r1i1p1f1_gn_185001-201412.nc Sat Dec 1 10:58:36 2018: ncatted -O -a realization_index,global,m,i,1 /ccc/work/cont003/gencmip6/p86caub/IGCM_OUT/IPSLCM6/PROD/historical/CM61-LR-hist-03.1910/CMIP6/OCE/tos_Omon_IPSL-CM6A-LR_historical_r1i1p1f1_gn_185001-201412.nc Fri Nov 30 16:47:56 2018: ncatted -O -a realization_index,global,m,s,1 /ccc/work/cont003/gencmip6/p86caub/IGCM_OUT/IPSLCM6/PROD/historical/CM61-LR-hist-03.1910/CMIP6/OCE/tos_Omon_IPSL-CM6A-LR_historical_r1i1p1f1_gn_185001-201412.nc Thu Nov 29 16:47:45 2018: ncatted -O -a variant_label,global,m,c,r1i1p1f1 /ccc/work/cont003/gencmip6/p86caub/IGCM_OUT/IPSLCM6/PROD/historical/CM61-LR-hist-03.1910/CMIP6/OCE/tos_Omon_IPSL-CM6A-LR_historical_r3i1p1f1_gn_185001-201412.nc Thu Nov 29 16:47:45 2018: ncatted -O -a further_info_url,global,m,c,https://furtherinfo.es-doc.org/CMIP6.IPSL.IPSL-CM6A-LR.historical.none.r1i1p1f1 /ccc/work/cont003/gencmip6/p86caub/IGCM_OUT/IPSLCM6/PROD/historical/CM61-LR-hist-03.1910/CMIP6/OCE/tos_Omon_IPSL-CM6A-LR_historical_r3i1p1f1_gn_185001-201412.nc Thu Nov 29 16:47:45 2018: ncatted -O -a name,global,m,c,/ccc/work/cont003/gencmip6/p86caub/IGCM_OUT/IPSLCM6/PROD/historical/CM61-LR-hist-03.1910/CMIP6/OCE/tos_Omon_IPSL-CM6A-LR_historical_r1i1p1f1_gn_%start_date%-%end_date% /ccc/work/cont003/gencmip6/p86caub/IGCM_OUT/IPSLCM6/PROD/historical/CM61-LR-hist-03.1910/CMIP6/OCE/tos_Omon_IPSL-CM6A-LR_historical_r3i1p1f1_gn_185001-201412.nc Mon Sep 3 14:53:26 2018: ncatted -O -a parent_variant_label,global,m,c,r1i1p1f1 tos_Omon_IPSL-CM6A-LR_historical_r3i1p1f1_gn_185001-201412.nc Mon Aug 6 17:58:17 2018: ncatted -O -a coordinates,area,o,c,nav_lon nav_lat /ccc/work/cont003/gencmip6/p86caub/IGCM_OUT/IPSLCM6/PROD/historical/CM61-LR-hist-03.1910/CMIP6/OCE/tos_Omon_IPSL-CM6A-LR_historical_r3i1p1f1_gn_185001-201412.nc none
- initialization_index :
- 1
- institution :
- Institut Pierre Simon Laplace, Paris 75252, France
- institution_id :
- IPSL
- license :
- CMIP6 model data produced by IPSL is licensed under a Creative Commons Attribution-NonCommercial-ShareAlike 4.0 International License (https://creativecommons.org/licenses). Consult https://pcmdi.llnl.gov/CMIP6/TermsOfUse for terms of use governing CMIP6 output, including citation requirements and proper acknowledgment. Further information about this data, including some limitations, can be found via the further_info_url (recorded as a global attribute in this file) and at https://cmc.ipsl.fr/. The data producers and data providers make no warranty, either express or implied, including, but not limited to, warranties of merchantability and fitness for a particular purpose. All liabilities arising from the supply of the information (including any liability arising in negligence) are excluded to the fullest extent permitted by law.
- mip_era :
- CMIP6
- model_version :
- 6.1.5
- name :
- /ccc/work/cont003/gencmip6/p86caub/IGCM_OUT/IPSLCM6/PROD/historical/CM61-LR-hist-03.1910/CMIP6/OCE/tos_Omon_IPSL-CM6A-LR_historical_r1i1p1f1_gn_%start_date%-%end_date%
- nominal_resolution :
- 100 km
- parent_activity_id :
- CMIP
- parent_experiment_id :
- piControl
- parent_mip_era :
- CMIP6
- parent_source_id :
- IPSL-CM6A-LR
- parent_time_units :
- days since 1850-01-01 00:00:00
- parent_variant_label :
- r1i1p1f1
- physics_index :
- 1
- product :
- model-output
- realization_index :
- 1
- realm :
- ocean
- source :
- IPSL-CM6A-LR (2017): atmos: LMDZ (NPv6, N96; 144 x 143 longitude/latitude; 79 levels; top level 40000 m) land: ORCHIDEE (v2.0, Water/Carbon/Energy mode) ocean: NEMO-OPA (eORCA1.3, tripolar primarily 1deg; 362 x 332 longitude/latitude; 75 levels; top grid cell 0-2 m) ocnBgchem: NEMO-PISCES seaIce: NEMO-LIM3
- source_id :
- IPSL-CM6A-LR
- source_type :
- AOGCM BGC
- sub_experiment :
- none
- sub_experiment_id :
- none
- table_id :
- Omon
- title :
- IPSL-CM6A-LR model output prepared for CMIP6 / CMIP historical
- tracking_id :
- hdl:21.14100/01f4d96a-9054-4974-b8dd-4f91e73989d2
- variable_id :
- tos
- variant_label :
- r1i1p1f1
- status :
- 2019-11-10;created;by nhn2@columbia.edu
- netcdf_tracking_ids :
- hdl:21.14100/01f4d96a-9054-4974-b8dd-4f91e73989d2
- version_id :
- v20180803
- intake_esm_vars :
- ['tos']
- intake_esm_attrs:activity_id :
- CMIP
- intake_esm_attrs:institution_id :
- IPSL
- intake_esm_attrs:source_id :
- IPSL-CM6A-LR
- intake_esm_attrs:experiment_id :
- historical
- intake_esm_attrs:member_id :
- r1i1p1f1
- intake_esm_attrs:table_id :
- Omon
- intake_esm_attrs:variable_id :
- tos
- intake_esm_attrs:grid_label :
- gn
- intake_esm_attrs:zstore :
- gs://cmip6/CMIP6/CMIP/IPSL/IPSL-CM6A-LR/historical/r1i1p1f1/Omon/tos/gn/v20180803/
- intake_esm_attrs:version :
- 20180803
- intake_esm_attrs:_data_format_ :
- zarr
- intake_esm_dataset_key :
- CMIP.IPSL.IPSL-CM6A-LR.historical.r1i1p1f1.Omon.tos.gn.gs://cmip6/CMIP6/CMIP/IPSL/IPSL-CM6A-LR/historical/r1i1p1f1/Omon/tos/gn/v20180803/.20180803
Ahora sí podemos generar un gráfico de la temperatura media global ponderada por el área de cada celda
dt["IPSL-CM6A-LR/historical"].isel(member_id=0, dcpp_init_year=0, time=0).tos.plot(
cmap="Spectral_r", vmin=-5, vmax=35
)
<matplotlib.collections.QuadMesh at 0x7f050837f050>
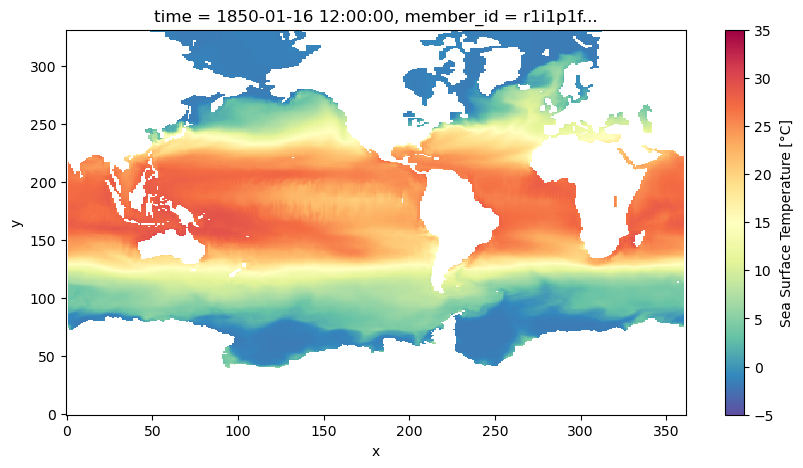
Ahora podemos calcular la anomalía de la temperatura superficial media global ponderada por latitud para cada uno de los modelos.
# Temperatura media global
def global_mean_sst(ds):
return ds.tos.weighted(ds.areacello.fillna(0)).mean(["x", "y"]).persist()
timeseries = dt.map_over_subtree(global_mean_sst)
timeseries
<xarray.DatasetView>
Dimensions: ()
Data variables:
*empty*- time: 1032
- member_id: 1
- dcpp_init_year: 1
- time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bounds
- long_name :
- Time axis
- standard_name :
- time
- time_origin :
- 2015-01-01 00:00:00
array([cftime.DatetimeGregorian(2015, 1, 16, 12, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2015, 2, 15, 0, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2015, 3, 16, 12, 0, 0, 0, has_year_zero=False), ..., cftime.DatetimeGregorian(2100, 10, 16, 12, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2100, 11, 16, 0, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2100, 12, 16, 12, 0, 0, 0, has_year_zero=False)], dtype=object) - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- tos(member_id, dcpp_init_year, time)float32dask.array<chunksize=(1, 1, 250), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- description :
- This may differ from "surface temperature" in regions of sea ice or floating ice shelves. For models using conservative temperature as the prognostic field, they should report the top ocean layer as surface potential temperature, which is the same as surface in situ temperature.
- history :
- none
- interval_operation :
- 2700 s
- interval_write :
- 1 month
- long_name :
- Sea Surface Temperature
- online_operation :
- average
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 4.03 kiB 0.98 kiB Shape (1, 1, 1032) (1, 1, 250) Dask graph 5 chunks in 1 graph layer Data type float32 numpy.ndarray
<xarray.DatasetView> Dimensions: (time: 1032, member_id: 1, dcpp_init_year: 1) Coordinates: * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan Data variables: tos (member_id, dcpp_init_year, time) float32 dask.array<chunksize=(1, 1, 250), meta=np.ndarray>ssp126- time: 1032
- member_id: 1
- dcpp_init_year: 1
- time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bounds
- long_name :
- Time axis
- standard_name :
- time
- time_origin :
- 2015-01-01 00:00:00
array([cftime.DatetimeGregorian(2015, 1, 16, 12, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2015, 2, 15, 0, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2015, 3, 16, 12, 0, 0, 0, has_year_zero=False), ..., cftime.DatetimeGregorian(2100, 10, 16, 12, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2100, 11, 16, 0, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2100, 12, 16, 12, 0, 0, 0, has_year_zero=False)], dtype=object) - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- tos(member_id, dcpp_init_year, time)float32dask.array<chunksize=(1, 1, 120), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- description :
- This may differ from "surface temperature" in regions of sea ice or floating ice shelves. For models using conservative temperature as the prognostic field, they should report the top ocean layer as surface potential temperature, which is the same as surface in situ temperature.
- history :
- none
- interval_operation :
- 2700 s
- interval_write :
- 1 month
- long_name :
- Sea Surface Temperature
- online_operation :
- average
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 4.03 kiB 480 B Shape (1, 1, 1032) (1, 1, 120) Dask graph 9 chunks in 1 graph layer Data type float32 numpy.ndarray
<xarray.DatasetView> Dimensions: (time: 1032, member_id: 1, dcpp_init_year: 1) Coordinates: * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan Data variables: tos (member_id, dcpp_init_year, time) float32 dask.array<chunksize=(1, 1, 120), meta=np.ndarray>ssp585- time: 1980
- member_id: 1
- dcpp_init_year: 1
- time(time)object1850-01-16 12:00:00 ... 2014-12-...
- axis :
- T
- bounds :
- time_bounds
- long_name :
- Time axis
- standard_name :
- time
- time_origin :
- 1850-01-01 00:00:00
array([cftime.DatetimeGregorian(1850, 1, 16, 12, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(1850, 2, 15, 0, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(1850, 3, 16, 12, 0, 0, 0, has_year_zero=False), ..., cftime.DatetimeGregorian(2014, 10, 16, 12, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2014, 11, 16, 0, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2014, 12, 16, 12, 0, 0, 0, has_year_zero=False)], dtype=object) - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- tos(member_id, dcpp_init_year, time)float32dask.array<chunksize=(1, 1, 252), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- description :
- This may differ from "surface temperature" in regions of sea ice or floating ice shelves. For models using conservative temperature as the prognostic field, they should report the top ocean layer as surface potential temperature, which is the same as surface in situ temperature.
- history :
- none
- interval_operation :
- 2700 s
- interval_write :
- 1 month
- long_name :
- Sea Surface Temperature
- online_operation :
- average
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 7.73 kiB 0.98 kiB Shape (1, 1, 1980) (1, 1, 252) Dask graph 8 chunks in 1 graph layer Data type float32 numpy.ndarray
<xarray.DatasetView> Dimensions: (time: 1980, member_id: 1, dcpp_init_year: 1) Coordinates: * time (time) object 1850-01-16 12:00:00 ... 2014-12-16 12:00:00 * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan Data variables: tos (member_id, dcpp_init_year, time) float32 dask.array<chunksize=(1, 1, 252), meta=np.ndarray>historical- time: 1032
- member_id: 1
- dcpp_init_year: 1
- time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bounds
- long_name :
- Time axis
- standard_name :
- time
- time_origin :
- 2015-01-01 00:00:00
array([cftime.DatetimeGregorian(2015, 1, 16, 12, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2015, 2, 15, 0, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2015, 3, 16, 12, 0, 0, 0, has_year_zero=False), ..., cftime.DatetimeGregorian(2100, 10, 16, 12, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2100, 11, 16, 0, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2100, 12, 16, 12, 0, 0, 0, has_year_zero=False)], dtype=object) - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- tos(member_id, dcpp_init_year, time)float32dask.array<chunksize=(1, 1, 250), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- description :
- This may differ from "surface temperature" in regions of sea ice or floating ice shelves. For models using conservative temperature as the prognostic field, they should report the top ocean layer as surface potential temperature, which is the same as surface in situ temperature.
- history :
- none
- interval_operation :
- 2700 s
- interval_write :
- 1 month
- long_name :
- Sea Surface Temperature
- online_operation :
- average
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 4.03 kiB 0.98 kiB Shape (1, 1, 1032) (1, 1, 250) Dask graph 5 chunks in 1 graph layer Data type float32 numpy.ndarray
<xarray.DatasetView> Dimensions: (time: 1032, member_id: 1, dcpp_init_year: 1) Coordinates: * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan Data variables: tos (member_id, dcpp_init_year, time) float32 dask.array<chunksize=(1, 1, 250), meta=np.ndarray>ssp370- time: 1032
- member_id: 1
- dcpp_init_year: 1
- time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bounds
- long_name :
- Time axis
- standard_name :
- time
- time_origin :
- 2015-01-01 00:00:00
array([cftime.DatetimeGregorian(2015, 1, 16, 12, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2015, 2, 15, 0, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2015, 3, 16, 12, 0, 0, 0, has_year_zero=False), ..., cftime.DatetimeGregorian(2100, 10, 16, 12, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2100, 11, 16, 0, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2100, 12, 16, 12, 0, 0, 0, has_year_zero=False)], dtype=object) - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- tos(member_id, dcpp_init_year, time)float32dask.array<chunksize=(1, 1, 120), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- description :
- This may differ from "surface temperature" in regions of sea ice or floating ice shelves. For models using conservative temperature as the prognostic field, they should report the top ocean layer as surface potential temperature, which is the same as surface in situ temperature.
- history :
- none
- interval_operation :
- 2700 s
- interval_write :
- 1 month
- long_name :
- Sea Surface Temperature
- online_operation :
- average
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 4.03 kiB 480 B Shape (1, 1, 1032) (1, 1, 120) Dask graph 9 chunks in 1 graph layer Data type float32 numpy.ndarray
<xarray.DatasetView> Dimensions: (time: 1032, member_id: 1, dcpp_init_year: 1) Coordinates: * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan Data variables: tos (member_id, dcpp_init_year, time) float32 dask.array<chunksize=(1, 1, 120), meta=np.ndarray>ssp245
<xarray.DatasetView> Dimensions: () Data variables: *empty*IPSL-CM6A-LR- time: 1032
- member_id: 1
- dcpp_init_year: 1
- time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bnds
- long_name :
- time
- standard_name :
- time
array([cftime.DatetimeProlepticGregorian(2015, 1, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2015, 2, 15, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2015, 3, 16, 12, 0, 0, 0, has_year_zero=True), ..., cftime.DatetimeProlepticGregorian(2100, 10, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2100, 11, 16, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2100, 12, 16, 12, 0, 0, 0, has_year_zero=True)], dtype=object) - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- tos(member_id, dcpp_init_year, time)float32dask.array<chunksize=(1, 1, 394), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- comment :
- Temperature of upper boundary of the liquid ocean, including temperatures below sea-ice and floating ice shelves.
- long_name :
- Sea Surface Temperature
- original_name :
- tos
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 4.03 kiB 1.54 kiB Shape (1, 1, 1032) (1, 1, 394) Dask graph 3 chunks in 1 graph layer Data type float32 numpy.ndarray
<xarray.DatasetView> Dimensions: (time: 1032, member_id: 1, dcpp_init_year: 1) Coordinates: * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan Data variables: tos (member_id, dcpp_init_year, time) float32 dask.array<chunksize=(1, 1, 394), meta=np.ndarray>ssp126- time: 1032
- member_id: 1
- dcpp_init_year: 1
- time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bnds
- long_name :
- time
- standard_name :
- time
array([cftime.DatetimeProlepticGregorian(2015, 1, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2015, 2, 15, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2015, 3, 16, 12, 0, 0, 0, has_year_zero=True), ..., cftime.DatetimeProlepticGregorian(2100, 10, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2100, 11, 16, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2100, 12, 16, 12, 0, 0, 0, has_year_zero=True)], dtype=object) - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- tos(member_id, dcpp_init_year, time)float32dask.array<chunksize=(1, 1, 394), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- comment :
- Temperature of upper boundary of the liquid ocean, including temperatures below sea-ice and floating ice shelves.
- long_name :
- Sea Surface Temperature
- original_name :
- tos
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 4.03 kiB 1.54 kiB Shape (1, 1, 1032) (1, 1, 394) Dask graph 3 chunks in 1 graph layer Data type float32 numpy.ndarray
<xarray.DatasetView> Dimensions: (time: 1032, member_id: 1, dcpp_init_year: 1) Coordinates: * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan Data variables: tos (member_id, dcpp_init_year, time) float32 dask.array<chunksize=(1, 1, 394), meta=np.ndarray>ssp245- time: 1032
- member_id: 1
- dcpp_init_year: 1
- time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bnds
- long_name :
- time
- standard_name :
- time
array([cftime.DatetimeProlepticGregorian(2015, 1, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2015, 2, 15, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2015, 3, 16, 12, 0, 0, 0, has_year_zero=True), ..., cftime.DatetimeProlepticGregorian(2100, 10, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2100, 11, 16, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2100, 12, 16, 12, 0, 0, 0, has_year_zero=True)], dtype=object) - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- tos(member_id, dcpp_init_year, time)float32dask.array<chunksize=(1, 1, 394), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- comment :
- Temperature of upper boundary of the liquid ocean, including temperatures below sea-ice and floating ice shelves.
- history :
- 2019-11-04T13:44:14Z altered by CMOR: replaced missing value flag (-9e+33) and corresponding data with standard missing value (1e+20).
- long_name :
- Sea Surface Temperature
- original_name :
- tos
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 4.03 kiB 1.54 kiB Shape (1, 1, 1032) (1, 1, 394) Dask graph 3 chunks in 1 graph layer Data type float32 numpy.ndarray
<xarray.DatasetView> Dimensions: (time: 1032, member_id: 1, dcpp_init_year: 1) Coordinates: * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan Data variables: tos (member_id, dcpp_init_year, time) float32 dask.array<chunksize=(1, 1, 394), meta=np.ndarray>ssp370- time: 1032
- member_id: 1
- dcpp_init_year: 1
- time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bnds
- long_name :
- time
- standard_name :
- time
array([cftime.DatetimeProlepticGregorian(2015, 1, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2015, 2, 15, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2015, 3, 16, 12, 0, 0, 0, has_year_zero=True), ..., cftime.DatetimeProlepticGregorian(2100, 10, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2100, 11, 16, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2100, 12, 16, 12, 0, 0, 0, has_year_zero=True)], dtype=object) - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- tos(member_id, dcpp_init_year, time)float32dask.array<chunksize=(1, 1, 394), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- comment :
- Temperature of upper boundary of the liquid ocean, including temperatures below sea-ice and floating ice shelves.
- long_name :
- Sea Surface Temperature
- original_name :
- tos
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 4.03 kiB 1.54 kiB Shape (1, 1, 1032) (1, 1, 394) Dask graph 3 chunks in 1 graph layer Data type float32 numpy.ndarray
<xarray.DatasetView> Dimensions: (time: 1032, member_id: 1, dcpp_init_year: 1) Coordinates: * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan Data variables: tos (member_id, dcpp_init_year, time) float32 dask.array<chunksize=(1, 1, 394), meta=np.ndarray>ssp585- time: 1980
- member_id: 1
- dcpp_init_year: 1
- time(time)object1850-01-16 12:00:00 ... 2014-12-...
- axis :
- T
- bounds :
- time_bnds
- long_name :
- time
- standard_name :
- time
array([cftime.DatetimeProlepticGregorian(1850, 1, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(1850, 2, 15, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(1850, 3, 16, 12, 0, 0, 0, has_year_zero=True), ..., cftime.DatetimeProlepticGregorian(2014, 10, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2014, 11, 16, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2014, 12, 16, 12, 0, 0, 0, has_year_zero=True)], dtype=object) - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- tos(member_id, dcpp_init_year, time)float32dask.array<chunksize=(1, 1, 397), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- comment :
- Temperature of upper boundary of the liquid ocean, including temperatures below sea-ice and floating ice shelves.
- history :
- 2019-09-11T14:21:40Z altered by CMOR: replaced missing value flag (-9e+33) and corresponding data with standard missing value (1e+20).
- long_name :
- Sea Surface Temperature
- original_name :
- tos
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 7.73 kiB 1.55 kiB Shape (1, 1, 1980) (1, 1, 397) Dask graph 5 chunks in 1 graph layer Data type float32 numpy.ndarray
<xarray.DatasetView> Dimensions: (time: 1980, member_id: 1, dcpp_init_year: 1) Coordinates: * time (time) object 1850-01-16 12:00:00 ... 2014-12-16 12:00:00 * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan Data variables: tos (member_id, dcpp_init_year, time) float32 dask.array<chunksize=(1, 1, 397), meta=np.ndarray>historical
<xarray.DatasetView> Dimensions: () Data variables: *empty*MPI-ESM1-2-LR- time: 1032
- member_id: 1
- dcpp_init_year: 1
- time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bnds
- long_name :
- time
- standard_name :
- time
array([cftime.DatetimeNoLeap(2015, 1, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2015, 2, 15, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2015, 3, 16, 12, 0, 0, 0, has_year_zero=True), ..., cftime.DatetimeNoLeap(2100, 10, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2100, 11, 16, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2100, 12, 16, 12, 0, 0, 0, has_year_zero=True)], dtype=object) - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- tos(member_id, dcpp_init_year, time)float32dask.array<chunksize=(1, 1, 253), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- comment :
- Temperature of upper boundary of the liquid ocean, including temperatures below sea-ice and floating ice shelves.
- long_name :
- Sea Surface Temperature
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 4.03 kiB 0.99 kiB Shape (1, 1, 1032) (1, 1, 253) Dask graph 5 chunks in 1 graph layer Data type float32 numpy.ndarray
<xarray.DatasetView> Dimensions: (time: 1032, member_id: 1, dcpp_init_year: 1) Coordinates: * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan Data variables: tos (member_id, dcpp_init_year, time) float32 dask.array<chunksize=(1, 1, 253), meta=np.ndarray>ssp126- time: 1032
- member_id: 1
- dcpp_init_year: 1
- time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bnds
- long_name :
- time
- standard_name :
- time
array([cftime.DatetimeNoLeap(2015, 1, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2015, 2, 15, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2015, 3, 16, 12, 0, 0, 0, has_year_zero=True), ..., cftime.DatetimeNoLeap(2100, 10, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2100, 11, 16, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2100, 12, 16, 12, 0, 0, 0, has_year_zero=True)], dtype=object) - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- tos(member_id, dcpp_init_year, time)float32dask.array<chunksize=(1, 1, 252), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- comment :
- Temperature of upper boundary of the liquid ocean, including temperatures below sea-ice and floating ice shelves.
- long_name :
- Sea Surface Temperature
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 4.03 kiB 0.98 kiB Shape (1, 1, 1032) (1, 1, 252) Dask graph 5 chunks in 1 graph layer Data type float32 numpy.ndarray
<xarray.DatasetView> Dimensions: (time: 1032, member_id: 1, dcpp_init_year: 1) Coordinates: * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan Data variables: tos (member_id, dcpp_init_year, time) float32 dask.array<chunksize=(1, 1, 252), meta=np.ndarray>ssp370- time: 1032
- member_id: 1
- dcpp_init_year: 1
- time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bnds
- long_name :
- time
- standard_name :
- time
array([cftime.DatetimeNoLeap(2015, 1, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2015, 2, 15, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2015, 3, 16, 12, 0, 0, 0, has_year_zero=True), ..., cftime.DatetimeNoLeap(2100, 10, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2100, 11, 16, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2100, 12, 16, 12, 0, 0, 0, has_year_zero=True)], dtype=object) - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- tos(member_id, dcpp_init_year, time)float32dask.array<chunksize=(1, 1, 252), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- comment :
- Temperature of upper boundary of the liquid ocean, including temperatures below sea-ice and floating ice shelves.
- long_name :
- Sea Surface Temperature
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 4.03 kiB 0.98 kiB Shape (1, 1, 1032) (1, 1, 252) Dask graph 5 chunks in 1 graph layer Data type float32 numpy.ndarray
<xarray.DatasetView> Dimensions: (time: 1032, member_id: 1, dcpp_init_year: 1) Coordinates: * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan Data variables: tos (member_id, dcpp_init_year, time) float32 dask.array<chunksize=(1, 1, 252), meta=np.ndarray>ssp245- time: 1032
- member_id: 1
- dcpp_init_year: 1
- time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bnds
- long_name :
- time
- standard_name :
- time
array([cftime.DatetimeNoLeap(2015, 1, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2015, 2, 15, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2015, 3, 16, 12, 0, 0, 0, has_year_zero=True), ..., cftime.DatetimeNoLeap(2100, 10, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2100, 11, 16, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2100, 12, 16, 12, 0, 0, 0, has_year_zero=True)], dtype=object) - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- tos(member_id, dcpp_init_year, time)float32dask.array<chunksize=(1, 1, 253), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- comment :
- Temperature of upper boundary of the liquid ocean, including temperatures below sea-ice and floating ice shelves.
- long_name :
- Sea Surface Temperature
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 4.03 kiB 0.99 kiB Shape (1, 1, 1032) (1, 1, 253) Dask graph 5 chunks in 1 graph layer Data type float32 numpy.ndarray
<xarray.DatasetView> Dimensions: (time: 1032, member_id: 1, dcpp_init_year: 1) Coordinates: * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan Data variables: tos (member_id, dcpp_init_year, time) float32 dask.array<chunksize=(1, 1, 253), meta=np.ndarray>ssp585- time: 1980
- member_id: 1
- dcpp_init_year: 1
- time(time)object1850-01-16 12:00:00 ... 2014-12-...
- axis :
- T
- bounds :
- time_bnds
- long_name :
- time
- standard_name :
- time
array([cftime.DatetimeNoLeap(1850, 1, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(1850, 2, 15, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(1850, 3, 16, 12, 0, 0, 0, has_year_zero=True), ..., cftime.DatetimeNoLeap(2014, 10, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2014, 11, 16, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2014, 12, 16, 12, 0, 0, 0, has_year_zero=True)], dtype=object) - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- tos(member_id, dcpp_init_year, time)float32dask.array<chunksize=(1, 1, 255), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- comment :
- Temperature of upper boundary of the liquid ocean, including temperatures below sea-ice and floating ice shelves.
- long_name :
- Sea Surface Temperature
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 7.73 kiB 1.00 kiB Shape (1, 1, 1980) (1, 1, 255) Dask graph 8 chunks in 1 graph layer Data type float32 numpy.ndarray
<xarray.DatasetView> Dimensions: (time: 1980, member_id: 1, dcpp_init_year: 1) Coordinates: * time (time) object 1850-01-16 12:00:00 ... 2014-12-16 12:00:00 * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan Data variables: tos (member_id, dcpp_init_year, time) float32 dask.array<chunksize=(1, 1, 255), meta=np.ndarray>historical
<xarray.DatasetView> Dimensions: () Data variables: *empty*CMCC-ESM2
Podemos generar una serie de tiempo para inspeccionar los resultados. Usemos el modelo IPSl con el escenario ssp585.
ds_IPSL_ssp585 = timeseries["/IPSL-CM6A-LR/ssp585"].ds
ds_IPSL_ssp585 = ds_IPSL_ssp585.assign_coords(
time=("time", ds_IPSL_ssp585.time.data.astype("datetime64[ns]"))
)
ds_IPSL_ssp585["tos"].plot()
[<matplotlib.lines.Line2D at 0x7f0508b01b90>]
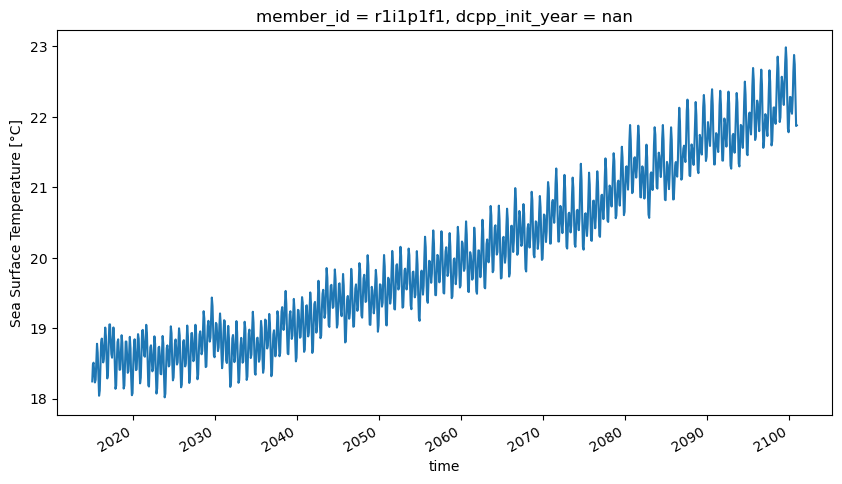
4. Anomalía de la temperatura media global ponderada
De igual manera podemos remover el ciclo estacional, también llamado “climatología”, para calcular la anomalía de la temperatura superficial del Océano.
# Valor medio histórico de referencia
def get_ref_value(ds):
return ds.sel(time=slice("1950", "1980")).mean("time")
anomaly = DataTree()
for model_name, model in timeseries.children.items():
# model-specific base period
base_period = get_ref_value(model["historical"].ds)
anomaly[model_name] = model - base_period # subtree - Dataset
display(anomaly)
<xarray.DatasetView>
Dimensions: ()
Data variables:
*empty*- time: 1032
- member_id: 1
- dcpp_init_year: 1
- time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bounds
- long_name :
- Time axis
- standard_name :
- time
- time_origin :
- 2015-01-01 00:00:00
array([cftime.DatetimeGregorian(2015, 1, 16, 12, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2015, 2, 15, 0, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2015, 3, 16, 12, 0, 0, 0, has_year_zero=False), ..., cftime.DatetimeGregorian(2100, 10, 16, 12, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2100, 11, 16, 0, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2100, 12, 16, 12, 0, 0, 0, has_year_zero=False)], dtype=object) - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- tos(member_id, dcpp_init_year, time)float32dask.array<chunksize=(1, 1, 250), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- description :
- This may differ from "surface temperature" in regions of sea ice or floating ice shelves. For models using conservative temperature as the prognostic field, they should report the top ocean layer as surface potential temperature, which is the same as surface in situ temperature.
- history :
- none
- interval_operation :
- 2700 s
- interval_write :
- 1 month
- long_name :
- Sea Surface Temperature
- online_operation :
- average
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 4.03 kiB 0.98 kiB Shape (1, 1, 1032) (1, 1, 250) Dask graph 5 chunks in 8 graph layers Data type float32 numpy.ndarray
<xarray.DatasetView> Dimensions: (time: 1032, member_id: 1, dcpp_init_year: 1) Coordinates: * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan Data variables: tos (member_id, dcpp_init_year, time) float32 dask.array<chunksize=(1, 1, 250), meta=np.ndarray>ssp126- time: 1032
- member_id: 1
- dcpp_init_year: 1
- time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bounds
- long_name :
- Time axis
- standard_name :
- time
- time_origin :
- 2015-01-01 00:00:00
array([cftime.DatetimeGregorian(2015, 1, 16, 12, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2015, 2, 15, 0, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2015, 3, 16, 12, 0, 0, 0, has_year_zero=False), ..., cftime.DatetimeGregorian(2100, 10, 16, 12, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2100, 11, 16, 0, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2100, 12, 16, 12, 0, 0, 0, has_year_zero=False)], dtype=object) - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- tos(member_id, dcpp_init_year, time)float32dask.array<chunksize=(1, 1, 120), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- description :
- This may differ from "surface temperature" in regions of sea ice or floating ice shelves. For models using conservative temperature as the prognostic field, they should report the top ocean layer as surface potential temperature, which is the same as surface in situ temperature.
- history :
- none
- interval_operation :
- 2700 s
- interval_write :
- 1 month
- long_name :
- Sea Surface Temperature
- online_operation :
- average
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 4.03 kiB 480 B Shape (1, 1, 1032) (1, 1, 120) Dask graph 9 chunks in 8 graph layers Data type float32 numpy.ndarray
<xarray.DatasetView> Dimensions: (time: 1032, member_id: 1, dcpp_init_year: 1) Coordinates: * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan Data variables: tos (member_id, dcpp_init_year, time) float32 dask.array<chunksize=(1, 1, 120), meta=np.ndarray>ssp585- time: 1980
- member_id: 1
- dcpp_init_year: 1
- time(time)object1850-01-16 12:00:00 ... 2014-12-...
- axis :
- T
- bounds :
- time_bounds
- long_name :
- Time axis
- standard_name :
- time
- time_origin :
- 1850-01-01 00:00:00
array([cftime.DatetimeGregorian(1850, 1, 16, 12, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(1850, 2, 15, 0, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(1850, 3, 16, 12, 0, 0, 0, has_year_zero=False), ..., cftime.DatetimeGregorian(2014, 10, 16, 12, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2014, 11, 16, 0, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2014, 12, 16, 12, 0, 0, 0, has_year_zero=False)], dtype=object) - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- tos(member_id, dcpp_init_year, time)float32dask.array<chunksize=(1, 1, 252), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- description :
- This may differ from "surface temperature" in regions of sea ice or floating ice shelves. For models using conservative temperature as the prognostic field, they should report the top ocean layer as surface potential temperature, which is the same as surface in situ temperature.
- history :
- none
- interval_operation :
- 2700 s
- interval_write :
- 1 month
- long_name :
- Sea Surface Temperature
- online_operation :
- average
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 7.73 kiB 0.98 kiB Shape (1, 1, 1980) (1, 1, 252) Dask graph 8 chunks in 7 graph layers Data type float32 numpy.ndarray
<xarray.DatasetView> Dimensions: (time: 1980, member_id: 1, dcpp_init_year: 1) Coordinates: * time (time) object 1850-01-16 12:00:00 ... 2014-12-16 12:00:00 * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan Data variables: tos (member_id, dcpp_init_year, time) float32 dask.array<chunksize=(1, 1, 252), meta=np.ndarray>historical- time: 1032
- member_id: 1
- dcpp_init_year: 1
- time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bounds
- long_name :
- Time axis
- standard_name :
- time
- time_origin :
- 2015-01-01 00:00:00
array([cftime.DatetimeGregorian(2015, 1, 16, 12, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2015, 2, 15, 0, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2015, 3, 16, 12, 0, 0, 0, has_year_zero=False), ..., cftime.DatetimeGregorian(2100, 10, 16, 12, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2100, 11, 16, 0, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2100, 12, 16, 12, 0, 0, 0, has_year_zero=False)], dtype=object) - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- tos(member_id, dcpp_init_year, time)float32dask.array<chunksize=(1, 1, 250), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- description :
- This may differ from "surface temperature" in regions of sea ice or floating ice shelves. For models using conservative temperature as the prognostic field, they should report the top ocean layer as surface potential temperature, which is the same as surface in situ temperature.
- history :
- none
- interval_operation :
- 2700 s
- interval_write :
- 1 month
- long_name :
- Sea Surface Temperature
- online_operation :
- average
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 4.03 kiB 0.98 kiB Shape (1, 1, 1032) (1, 1, 250) Dask graph 5 chunks in 8 graph layers Data type float32 numpy.ndarray
<xarray.DatasetView> Dimensions: (time: 1032, member_id: 1, dcpp_init_year: 1) Coordinates: * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan Data variables: tos (member_id, dcpp_init_year, time) float32 dask.array<chunksize=(1, 1, 250), meta=np.ndarray>ssp370- time: 1032
- member_id: 1
- dcpp_init_year: 1
- time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bounds
- long_name :
- Time axis
- standard_name :
- time
- time_origin :
- 2015-01-01 00:00:00
array([cftime.DatetimeGregorian(2015, 1, 16, 12, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2015, 2, 15, 0, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2015, 3, 16, 12, 0, 0, 0, has_year_zero=False), ..., cftime.DatetimeGregorian(2100, 10, 16, 12, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2100, 11, 16, 0, 0, 0, 0, has_year_zero=False), cftime.DatetimeGregorian(2100, 12, 16, 12, 0, 0, 0, has_year_zero=False)], dtype=object) - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- tos(member_id, dcpp_init_year, time)float32dask.array<chunksize=(1, 1, 120), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- description :
- This may differ from "surface temperature" in regions of sea ice or floating ice shelves. For models using conservative temperature as the prognostic field, they should report the top ocean layer as surface potential temperature, which is the same as surface in situ temperature.
- history :
- none
- interval_operation :
- 2700 s
- interval_write :
- 1 month
- long_name :
- Sea Surface Temperature
- online_operation :
- average
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 4.03 kiB 480 B Shape (1, 1, 1032) (1, 1, 120) Dask graph 9 chunks in 8 graph layers Data type float32 numpy.ndarray
<xarray.DatasetView> Dimensions: (time: 1032, member_id: 1, dcpp_init_year: 1) Coordinates: * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan Data variables: tos (member_id, dcpp_init_year, time) float32 dask.array<chunksize=(1, 1, 120), meta=np.ndarray>ssp245
<xarray.DatasetView> Dimensions: () Data variables: *empty*IPSL-CM6A-LR- time: 1032
- member_id: 1
- dcpp_init_year: 1
- time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bnds
- long_name :
- time
- standard_name :
- time
array([cftime.DatetimeProlepticGregorian(2015, 1, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2015, 2, 15, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2015, 3, 16, 12, 0, 0, 0, has_year_zero=True), ..., cftime.DatetimeProlepticGregorian(2100, 10, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2100, 11, 16, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2100, 12, 16, 12, 0, 0, 0, has_year_zero=True)], dtype=object) - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- tos(member_id, dcpp_init_year, time)float32dask.array<chunksize=(1, 1, 394), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- comment :
- Temperature of upper boundary of the liquid ocean, including temperatures below sea-ice and floating ice shelves.
- long_name :
- Sea Surface Temperature
- original_name :
- tos
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 4.03 kiB 1.54 kiB Shape (1, 1, 1032) (1, 1, 394) Dask graph 3 chunks in 8 graph layers Data type float32 numpy.ndarray
<xarray.DatasetView> Dimensions: (time: 1032, member_id: 1, dcpp_init_year: 1) Coordinates: * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan Data variables: tos (member_id, dcpp_init_year, time) float32 dask.array<chunksize=(1, 1, 394), meta=np.ndarray>ssp126- time: 1032
- member_id: 1
- dcpp_init_year: 1
- time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bnds
- long_name :
- time
- standard_name :
- time
array([cftime.DatetimeProlepticGregorian(2015, 1, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2015, 2, 15, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2015, 3, 16, 12, 0, 0, 0, has_year_zero=True), ..., cftime.DatetimeProlepticGregorian(2100, 10, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2100, 11, 16, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2100, 12, 16, 12, 0, 0, 0, has_year_zero=True)], dtype=object) - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- tos(member_id, dcpp_init_year, time)float32dask.array<chunksize=(1, 1, 394), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- comment :
- Temperature of upper boundary of the liquid ocean, including temperatures below sea-ice and floating ice shelves.
- long_name :
- Sea Surface Temperature
- original_name :
- tos
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 4.03 kiB 1.54 kiB Shape (1, 1, 1032) (1, 1, 394) Dask graph 3 chunks in 8 graph layers Data type float32 numpy.ndarray
<xarray.DatasetView> Dimensions: (time: 1032, member_id: 1, dcpp_init_year: 1) Coordinates: * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan Data variables: tos (member_id, dcpp_init_year, time) float32 dask.array<chunksize=(1, 1, 394), meta=np.ndarray>ssp245- time: 1032
- member_id: 1
- dcpp_init_year: 1
- time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bnds
- long_name :
- time
- standard_name :
- time
array([cftime.DatetimeProlepticGregorian(2015, 1, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2015, 2, 15, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2015, 3, 16, 12, 0, 0, 0, has_year_zero=True), ..., cftime.DatetimeProlepticGregorian(2100, 10, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2100, 11, 16, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2100, 12, 16, 12, 0, 0, 0, has_year_zero=True)], dtype=object) - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- tos(member_id, dcpp_init_year, time)float32dask.array<chunksize=(1, 1, 394), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- comment :
- Temperature of upper boundary of the liquid ocean, including temperatures below sea-ice and floating ice shelves.
- history :
- 2019-11-04T13:44:14Z altered by CMOR: replaced missing value flag (-9e+33) and corresponding data with standard missing value (1e+20).
- long_name :
- Sea Surface Temperature
- original_name :
- tos
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 4.03 kiB 1.54 kiB Shape (1, 1, 1032) (1, 1, 394) Dask graph 3 chunks in 8 graph layers Data type float32 numpy.ndarray
<xarray.DatasetView> Dimensions: (time: 1032, member_id: 1, dcpp_init_year: 1) Coordinates: * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan Data variables: tos (member_id, dcpp_init_year, time) float32 dask.array<chunksize=(1, 1, 394), meta=np.ndarray>ssp370- time: 1032
- member_id: 1
- dcpp_init_year: 1
- time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bnds
- long_name :
- time
- standard_name :
- time
array([cftime.DatetimeProlepticGregorian(2015, 1, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2015, 2, 15, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2015, 3, 16, 12, 0, 0, 0, has_year_zero=True), ..., cftime.DatetimeProlepticGregorian(2100, 10, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2100, 11, 16, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2100, 12, 16, 12, 0, 0, 0, has_year_zero=True)], dtype=object) - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- tos(member_id, dcpp_init_year, time)float32dask.array<chunksize=(1, 1, 394), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- comment :
- Temperature of upper boundary of the liquid ocean, including temperatures below sea-ice and floating ice shelves.
- long_name :
- Sea Surface Temperature
- original_name :
- tos
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 4.03 kiB 1.54 kiB Shape (1, 1, 1032) (1, 1, 394) Dask graph 3 chunks in 8 graph layers Data type float32 numpy.ndarray
<xarray.DatasetView> Dimensions: (time: 1032, member_id: 1, dcpp_init_year: 1) Coordinates: * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan Data variables: tos (member_id, dcpp_init_year, time) float32 dask.array<chunksize=(1, 1, 394), meta=np.ndarray>ssp585- time: 1980
- member_id: 1
- dcpp_init_year: 1
- time(time)object1850-01-16 12:00:00 ... 2014-12-...
- axis :
- T
- bounds :
- time_bnds
- long_name :
- time
- standard_name :
- time
array([cftime.DatetimeProlepticGregorian(1850, 1, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(1850, 2, 15, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(1850, 3, 16, 12, 0, 0, 0, has_year_zero=True), ..., cftime.DatetimeProlepticGregorian(2014, 10, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2014, 11, 16, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeProlepticGregorian(2014, 12, 16, 12, 0, 0, 0, has_year_zero=True)], dtype=object) - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- tos(member_id, dcpp_init_year, time)float32dask.array<chunksize=(1, 1, 397), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- comment :
- Temperature of upper boundary of the liquid ocean, including temperatures below sea-ice and floating ice shelves.
- history :
- 2019-09-11T14:21:40Z altered by CMOR: replaced missing value flag (-9e+33) and corresponding data with standard missing value (1e+20).
- long_name :
- Sea Surface Temperature
- original_name :
- tos
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 7.73 kiB 1.55 kiB Shape (1, 1, 1980) (1, 1, 397) Dask graph 5 chunks in 7 graph layers Data type float32 numpy.ndarray
<xarray.DatasetView> Dimensions: (time: 1980, member_id: 1, dcpp_init_year: 1) Coordinates: * time (time) object 1850-01-16 12:00:00 ... 2014-12-16 12:00:00 * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan Data variables: tos (member_id, dcpp_init_year, time) float32 dask.array<chunksize=(1, 1, 397), meta=np.ndarray>historical
<xarray.DatasetView> Dimensions: () Data variables: *empty*MPI-ESM1-2-LR- time: 1032
- member_id: 1
- dcpp_init_year: 1
- time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bnds
- long_name :
- time
- standard_name :
- time
array([cftime.DatetimeNoLeap(2015, 1, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2015, 2, 15, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2015, 3, 16, 12, 0, 0, 0, has_year_zero=True), ..., cftime.DatetimeNoLeap(2100, 10, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2100, 11, 16, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2100, 12, 16, 12, 0, 0, 0, has_year_zero=True)], dtype=object) - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- tos(member_id, dcpp_init_year, time)float32dask.array<chunksize=(1, 1, 253), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- comment :
- Temperature of upper boundary of the liquid ocean, including temperatures below sea-ice and floating ice shelves.
- long_name :
- Sea Surface Temperature
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 4.03 kiB 0.99 kiB Shape (1, 1, 1032) (1, 1, 253) Dask graph 5 chunks in 8 graph layers Data type float32 numpy.ndarray
<xarray.DatasetView> Dimensions: (time: 1032, member_id: 1, dcpp_init_year: 1) Coordinates: * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan Data variables: tos (member_id, dcpp_init_year, time) float32 dask.array<chunksize=(1, 1, 253), meta=np.ndarray>ssp126- time: 1032
- member_id: 1
- dcpp_init_year: 1
- time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bnds
- long_name :
- time
- standard_name :
- time
array([cftime.DatetimeNoLeap(2015, 1, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2015, 2, 15, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2015, 3, 16, 12, 0, 0, 0, has_year_zero=True), ..., cftime.DatetimeNoLeap(2100, 10, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2100, 11, 16, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2100, 12, 16, 12, 0, 0, 0, has_year_zero=True)], dtype=object) - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- tos(member_id, dcpp_init_year, time)float32dask.array<chunksize=(1, 1, 252), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- comment :
- Temperature of upper boundary of the liquid ocean, including temperatures below sea-ice and floating ice shelves.
- long_name :
- Sea Surface Temperature
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 4.03 kiB 0.98 kiB Shape (1, 1, 1032) (1, 1, 252) Dask graph 5 chunks in 8 graph layers Data type float32 numpy.ndarray
<xarray.DatasetView> Dimensions: (time: 1032, member_id: 1, dcpp_init_year: 1) Coordinates: * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan Data variables: tos (member_id, dcpp_init_year, time) float32 dask.array<chunksize=(1, 1, 252), meta=np.ndarray>ssp370- time: 1032
- member_id: 1
- dcpp_init_year: 1
- time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bnds
- long_name :
- time
- standard_name :
- time
array([cftime.DatetimeNoLeap(2015, 1, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2015, 2, 15, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2015, 3, 16, 12, 0, 0, 0, has_year_zero=True), ..., cftime.DatetimeNoLeap(2100, 10, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2100, 11, 16, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2100, 12, 16, 12, 0, 0, 0, has_year_zero=True)], dtype=object) - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- tos(member_id, dcpp_init_year, time)float32dask.array<chunksize=(1, 1, 252), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- comment :
- Temperature of upper boundary of the liquid ocean, including temperatures below sea-ice and floating ice shelves.
- long_name :
- Sea Surface Temperature
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 4.03 kiB 0.98 kiB Shape (1, 1, 1032) (1, 1, 252) Dask graph 5 chunks in 8 graph layers Data type float32 numpy.ndarray
<xarray.DatasetView> Dimensions: (time: 1032, member_id: 1, dcpp_init_year: 1) Coordinates: * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan Data variables: tos (member_id, dcpp_init_year, time) float32 dask.array<chunksize=(1, 1, 252), meta=np.ndarray>ssp245- time: 1032
- member_id: 1
- dcpp_init_year: 1
- time(time)object2015-01-16 12:00:00 ... 2100-12-...
- axis :
- T
- bounds :
- time_bnds
- long_name :
- time
- standard_name :
- time
array([cftime.DatetimeNoLeap(2015, 1, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2015, 2, 15, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2015, 3, 16, 12, 0, 0, 0, has_year_zero=True), ..., cftime.DatetimeNoLeap(2100, 10, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2100, 11, 16, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2100, 12, 16, 12, 0, 0, 0, has_year_zero=True)], dtype=object) - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- tos(member_id, dcpp_init_year, time)float32dask.array<chunksize=(1, 1, 253), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- comment :
- Temperature of upper boundary of the liquid ocean, including temperatures below sea-ice and floating ice shelves.
- long_name :
- Sea Surface Temperature
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 4.03 kiB 0.99 kiB Shape (1, 1, 1032) (1, 1, 253) Dask graph 5 chunks in 8 graph layers Data type float32 numpy.ndarray
<xarray.DatasetView> Dimensions: (time: 1032, member_id: 1, dcpp_init_year: 1) Coordinates: * time (time) object 2015-01-16 12:00:00 ... 2100-12-16 12:00:00 * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan Data variables: tos (member_id, dcpp_init_year, time) float32 dask.array<chunksize=(1, 1, 253), meta=np.ndarray>ssp585- time: 1980
- member_id: 1
- dcpp_init_year: 1
- time(time)object1850-01-16 12:00:00 ... 2014-12-...
- axis :
- T
- bounds :
- time_bnds
- long_name :
- time
- standard_name :
- time
array([cftime.DatetimeNoLeap(1850, 1, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(1850, 2, 15, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(1850, 3, 16, 12, 0, 0, 0, has_year_zero=True), ..., cftime.DatetimeNoLeap(2014, 10, 16, 12, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2014, 11, 16, 0, 0, 0, 0, has_year_zero=True), cftime.DatetimeNoLeap(2014, 12, 16, 12, 0, 0, 0, has_year_zero=True)], dtype=object) - member_id(member_id)object'r1i1p1f1'
array(['r1i1p1f1'], dtype=object)
- dcpp_init_year(dcpp_init_year)float64nan
array([nan])
- tos(member_id, dcpp_init_year, time)float32dask.array<chunksize=(1, 1, 255), meta=np.ndarray>
- cell_measures :
- area: areacello
- cell_methods :
- area: mean where sea time: mean
- comment :
- Temperature of upper boundary of the liquid ocean, including temperatures below sea-ice and floating ice shelves.
- long_name :
- Sea Surface Temperature
- standard_name :
- sea_surface_temperature
- units :
- °C
Array Chunk Bytes 7.73 kiB 1.00 kiB Shape (1, 1, 1980) (1, 1, 255) Dask graph 8 chunks in 7 graph layers Data type float32 numpy.ndarray
<xarray.DatasetView> Dimensions: (time: 1980, member_id: 1, dcpp_init_year: 1) Coordinates: * time (time) object 1850-01-16 12:00:00 ... 2014-12-16 12:00:00 * member_id (member_id) object 'r1i1p1f1' * dcpp_init_year (dcpp_init_year) float64 nan Data variables: tos (member_id, dcpp_init_year, time) float32 dask.array<chunksize=(1, 1, 255), meta=np.ndarray>historical
<xarray.DatasetView> Dimensions: () Data variables: *empty*CMCC-ESM2
Los datos de los modelos de cambio climático traen una estampa de tiempo en formato cftime.DatetimeGregorian. Xarray y Matplotlib generalmente trabajan con estampas de tiempo de datetime64. Debemos convertir estos índices usando la función replace_time. Luego podemos agrupar los datos por escenario usando un diccionario como se muestra a continuación:
# anomaly['/IPSL-CM6A-LR/ssp585'].time
def replace_time(ds):
start_date = ds.time.data[0]
new_time = date_range(
f"{start_date.year}-{start_date.month:02}", freq="1MS", periods=len(ds.time)
)
ds_new_cal = ds.assign_coords(time=new_time, source_id=model_name)
return ds_new_cal
experiment_dict = {
k: [] for k in ["historical", "ssp126", "ssp370", "ssp245", "ssp585"]
}
experiment_dict
{'historical': [], 'ssp126': [], 'ssp370': [], 'ssp245': [], 'ssp585': []}
Iteramos sobre cada uno de los modelos en el objeto datatree donde tenemos las anomalías de la temperatura superficial del mar y las agregamos a cada una de las llaves del diccionario
for model_name, model in list(anomaly.children.items()):
for experiment_name, experiment in model.children.items():
# replace the time dimension
ds_new_cal = replace_time(experiment.ds)
experiment_dict[experiment_name].append(ds_new_cal.load())
Concatenamos cada escenario a lo largo de la dimension source_id
# Concatenar todos los modelos para un determinado experimento
plot_dict = {
k: xr.concat(ds_lst, dim="source_id") for k, ds_lst in experiment_dict.items()
}
plot_dict
{'historical': <xarray.Dataset>
Dimensions: (member_id: 1, dcpp_init_year: 1, source_id: 3, time: 1980)
Coordinates:
* member_id (member_id) object 'r1i1p1f1'
* dcpp_init_year (dcpp_init_year) float64 nan
* time (time) datetime64[ns] 1850-01-01 1850-02-01 ... 2014-12-01
* source_id (source_id) <U13 'IPSL-CM6A-LR' 'MPI-ESM1-2-LR' 'CMCC-ESM2'
Data variables:
tos (source_id, member_id, dcpp_init_year, time) float32 -0.4...,
'ssp126': <xarray.Dataset>
Dimensions: (member_id: 1, dcpp_init_year: 1, source_id: 3, time: 1032)
Coordinates:
* member_id (member_id) object 'r1i1p1f1'
* dcpp_init_year (dcpp_init_year) float64 nan
* time (time) datetime64[ns] 2015-01-01 2015-02-01 ... 2100-12-01
* source_id (source_id) <U13 'IPSL-CM6A-LR' 'MPI-ESM1-2-LR' 'CMCC-ESM2'
Data variables:
tos (source_id, member_id, dcpp_init_year, time) float32 0.35...,
'ssp370': <xarray.Dataset>
Dimensions: (member_id: 1, dcpp_init_year: 1, source_id: 3, time: 1032)
Coordinates:
* member_id (member_id) object 'r1i1p1f1'
* dcpp_init_year (dcpp_init_year) float64 nan
* time (time) datetime64[ns] 2015-01-01 2015-02-01 ... 2100-12-01
* source_id (source_id) <U13 'IPSL-CM6A-LR' 'MPI-ESM1-2-LR' 'CMCC-ESM2'
Data variables:
tos (source_id, member_id, dcpp_init_year, time) float32 0.38...,
'ssp245': <xarray.Dataset>
Dimensions: (member_id: 1, dcpp_init_year: 1, source_id: 3, time: 1032)
Coordinates:
* member_id (member_id) object 'r1i1p1f1'
* dcpp_init_year (dcpp_init_year) float64 nan
* time (time) datetime64[ns] 2015-01-01 2015-02-01 ... 2100-12-01
* source_id (source_id) <U13 'IPSL-CM6A-LR' 'MPI-ESM1-2-LR' 'CMCC-ESM2'
Data variables:
tos (source_id, member_id, dcpp_init_year, time) float32 0.34...,
'ssp585': <xarray.Dataset>
Dimensions: (member_id: 1, dcpp_init_year: 1, source_id: 3, time: 1032)
Coordinates:
* member_id (member_id) object 'r1i1p1f1'
* dcpp_init_year (dcpp_init_year) float64 nan
* time (time) datetime64[ns] 2015-01-01 2015-02-01 ... 2100-12-01
* source_id (source_id) <U13 'IPSL-CM6A-LR' 'MPI-ESM1-2-LR' 'CMCC-ESM2'
Data variables:
tos (source_id, member_id, dcpp_init_year, time) float32 0.36...}
Comprobamos que nuestras estampas de tiempo esten en el formato datetime64
plot_dict["historical"].time
<xarray.DataArray 'time' (time: 1980)>
array(['1850-01-01T00:00:00.000000000', '1850-02-01T00:00:00.000000000',
'1850-03-01T00:00:00.000000000', ..., '2014-10-01T00:00:00.000000000',
'2014-11-01T00:00:00.000000000', '2014-12-01T00:00:00.000000000'],
dtype='datetime64[ns]')
Coordinates:
* time (time) datetime64[ns] 1850-01-01 1850-02-01 ... 2014-12-01- time: 1980
- 1850-01-01 1850-02-01 1850-03-01 ... 2014-10-01 2014-11-01 2014-12-01
array(['1850-01-01T00:00:00.000000000', '1850-02-01T00:00:00.000000000', '1850-03-01T00:00:00.000000000', ..., '2014-10-01T00:00:00.000000000', '2014-11-01T00:00:00.000000000', '2014-12-01T00:00:00.000000000'], dtype='datetime64[ns]') - time(time)datetime64[ns]1850-01-01 ... 2014-12-01
array(['1850-01-01T00:00:00.000000000', '1850-02-01T00:00:00.000000000', '1850-03-01T00:00:00.000000000', ..., '2014-10-01T00:00:00.000000000', '2014-11-01T00:00:00.000000000', '2014-12-01T00:00:00.000000000'], dtype='datetime64[ns]')
- timePandasIndex
PandasIndex(DatetimeIndex(['1850-01-01', '1850-02-01', '1850-03-01', '1850-04-01', '1850-05-01', '1850-06-01', '1850-07-01', '1850-08-01', '1850-09-01', '1850-10-01', ... '2014-03-01', '2014-04-01', '2014-05-01', '2014-06-01', '2014-07-01', '2014-08-01', '2014-09-01', '2014-10-01', '2014-11-01', '2014-12-01'], dtype='datetime64[ns]', name='time', length=1980, freq='MS'))
5. Esamble Multimodelo
Finalmente generamos nuestra gráfica de las proyecciones de la anomalía de temperatura superficial del mar bajo los distintos escenarios. Para esto podemos usar la función shaded_line_plot que produce un gráfico de líneas con intervalos sombreados según la extensión del datarray en la dimensión source_id. Creamos nuestro ensamble multimodelo!!!
fig, ax = plt.subplots(figsize=(8, 4))
color_dict = {
"historical": "0.5",
"ssp126": "C2",
"ssp245": "gold",
"ssp370": "C1",
"ssp585": "C3",
"observations": "C5",
}
for experiment, ds in plot_dict.items():
color = color_dict[experiment]
smooth = (
ds["tos"]
.sel(time=slice(None, "2100"))
.isel(member_id=0)
.rolling(time=2 * 12)
.mean()
.squeeze(["dcpp_init_year"])
)
lw = 2 if experiment == "observations" else 1.5
shaded_line_plot(
smooth,
"source_id",
ax=ax,
spreads=[2.0],
alphas=[0.2],
line_kwargs=dict(
color=color, label=f"{experiment} ({len(ds.source_id)})", lw=lw
),
)
plt.legend(loc=2)
plt.grid()
plt.tight_layout()
/usr/share/miniconda3/envs/atmoscol2023/lib/python3.11/site-packages/numpy/lib/nanfunctions.py:1879: RuntimeWarning: Degrees of freedom <= 0 for slice.
var = nanvar(a, axis=axis, dtype=dtype, out=out, ddof=ddof,
/usr/share/miniconda3/envs/atmoscol2023/lib/python3.11/site-packages/numpy/lib/nanfunctions.py:1879: RuntimeWarning: Degrees of freedom <= 0 for slice.
var = nanvar(a, axis=axis, dtype=dtype, out=out, ddof=ddof,
/usr/share/miniconda3/envs/atmoscol2023/lib/python3.11/site-packages/numpy/lib/nanfunctions.py:1879: RuntimeWarning: Degrees of freedom <= 0 for slice.
var = nanvar(a, axis=axis, dtype=dtype, out=out, ddof=ddof,
/usr/share/miniconda3/envs/atmoscol2023/lib/python3.11/site-packages/numpy/lib/nanfunctions.py:1879: RuntimeWarning: Degrees of freedom <= 0 for slice.
var = nanvar(a, axis=axis, dtype=dtype, out=out, ddof=ddof,
/usr/share/miniconda3/envs/atmoscol2023/lib/python3.11/site-packages/numpy/lib/nanfunctions.py:1879: RuntimeWarning: Degrees of freedom <= 0 for slice.
var = nanvar(a, axis=axis, dtype=dtype, out=out, ddof=ddof,
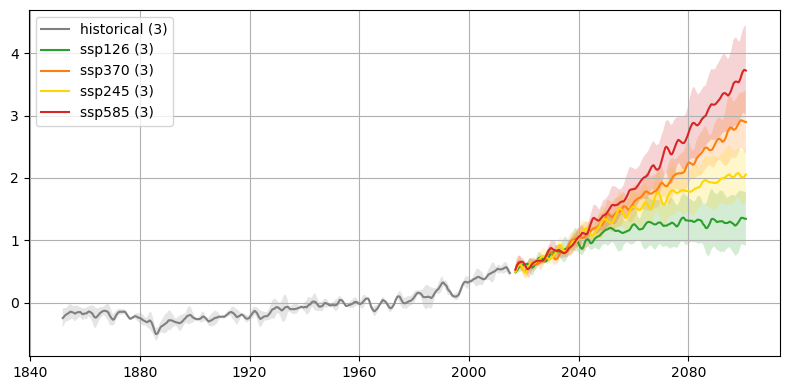
Resumen final
En este cuadernillo (notebook) revisamos temas relacionados con el acceso a la información proveniente de diferentes escenarios de Cambio Climático a partir de los conjuntos de datos disponibles en CMIP. Promediamos la temperatura superficial teniendo en cuenta el tamaño de las celdas del modelo y, reprodujimos la gráfica multimodelo de las proyecciones de cambio climático.
Fuentes y referencias
ClimateMatch Academy: Computational Tools for Climate Science. (s. f.). https://comptools.climatematch.io/tutorials/intro.html
Abernathey, R. 2023. An Introduction to Earth and Environmental Data Science. Retrieved from Earth and Environmental Data Science: https://earth-env-data-science.github.io/intro.html
Easy IPCC Part 1: Multi-Model Datatree. Nicholas T. 2019. https://medium.com/pangeo/easy-ipcc-part-1-multi-model-datatree-469b87cf9114